2° Ano E Biologia Profa Solange
Período para entrega: Até 28/10/2020
4° Bimestre: Unidade Temática: DNA- Tecnologias de Manipulação do DNA/ Biotecnologia
Instruções:
1- Assistir a vídeo aula (Khan Academy), link disponibilizado abaixo.
2- Fazer a Leitura e Interpretação de Texto.
3- Responder a atividade: Exercícios de Aprendizagem.
4- Postar no Blogger e enviar para o e-mail da professora: solangestandbyme@gmail.com
https://youtu.be/EDF_T0t7gN0
Introdução à biotecnologia
O que é biotecnologia. Visão geral da tecnologia do DNA. Questões éticas em biotecnologia.
Pontos Principais:
- Biotecnologia é o uso de um organismo, componente do organismo ou qualquer outro sistema biológico para fazer um produto ou processo.
- Muitas formas da biotecnologia moderna envolvem a tecnologia do DNA.
- A tecnologia do DNA é o sequenciamento, análise e corte e colagem do DNA.
- As formas típicas de tecnologia do DNA incluem o sequenciamento de DNA, reação em cadeia da polimerase, clonagem de DNA e eletroforese em gel.
- As invenções da biotecnologia podem levantar novas preocupações práticas e questões éticas que devem ser discutidas com a participação de toda a sociedade.
Introdução
O que lhe vem à mente quando você ouve a palavra "biotecnologia"? Talvez algumas notícias que você tenha visto, tais como a ovelha clonada Dolly, organismos geneticamente modificados, ou terapia gênica.
Se foi nisso que pensou, você está absolutamente certo: todos são exemplos de biotecnologia. E quanto à fabricação de cerveja, cultivares e o antibiótico penicilina? Esses processos e produtos – alguns existentes há milhares de anos – também são exemplos de biotecnologia.
Neste artigo, examinaremos primeiro a definição de biotecnologia e como ela abrange diversos usos dos organismos (e moléculas ou sistemas derivados dos organismos) para gerar produtos úteis. Depois, daremos uma olhada mais profunda na tecnologia do DNA, técnicas de manipulação e sequenciamento de DNA. A tecnologia do DNA é fundamental para as diversas formas modernas da biotecnologia.
O que é biotecnologia?
Biotecnologia é o uso de um organismo, componente de um organismo ou qualquer outro sistema biológico para fazer um produto ou processo para um uso específico.
Esta definição é muito ampla e, como mencionado acima, pode incluir tanto técnicas laboratoriais de ponta, técnicas agrícolas tradicionais e técnicas culinárias praticadas há centenas de anos. Vejamos três exemplos de biotecnologia e como eles se encaixam na definição:
- Fabricação de cerveja. Na fabricação de cerveja, minúsculos fungos (leveduras) são introduzidos em uma solução de açúcar de cevada maltada, que é vorazmente metabolizada por eles através de um processo chamado fermentação. O subproduto da fermentação é o álcool encontrado na cerveja. Aqui, vemos um organismo – a levedura – sendo usado para fazer um produto para o consumo humano.
- Penicillina. O antibiótico penicilina é gerado por certos fungos. Para obter as pequenas quantidades de penicilina utilizadas nos primeiros ensaios clínicos, pesquisadores tiveram que cultivar quase 500 litros de "caldo de fungo" por semanastart superscript, 1, end superscript. O processo foi então aperfeiçoado para a produção industrial, com o uso de cepas de fungos mais produtivos e melhores condições de cultura para aumentar o rendimentosquared. Aqui, vemos um organismo (fungo), sendo usado para fazer um produto de uso humano – neste caso, um antibiótico para tratar infecções bacterianas.
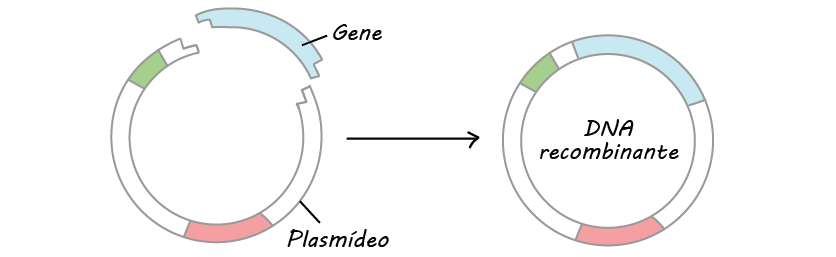
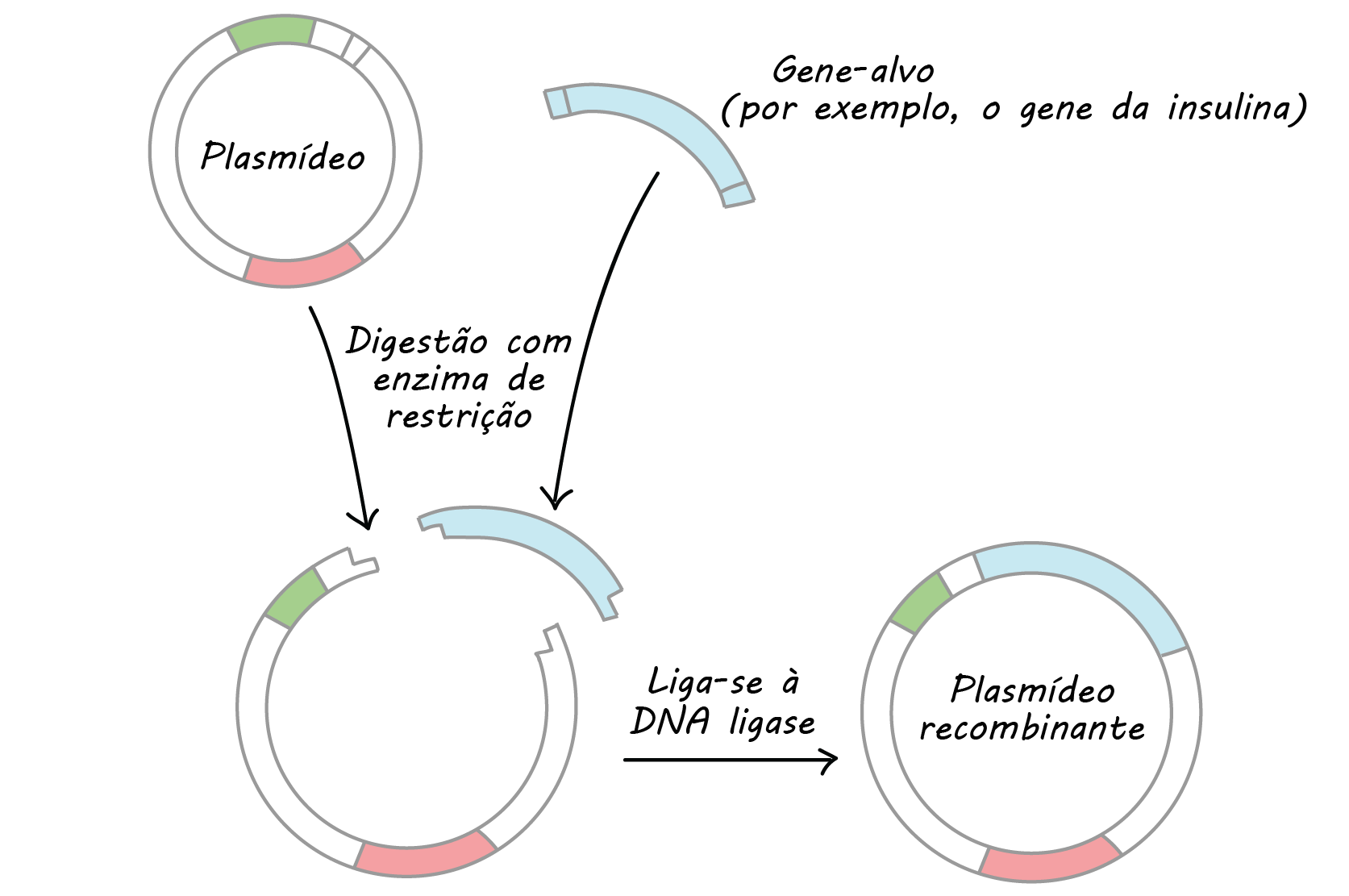
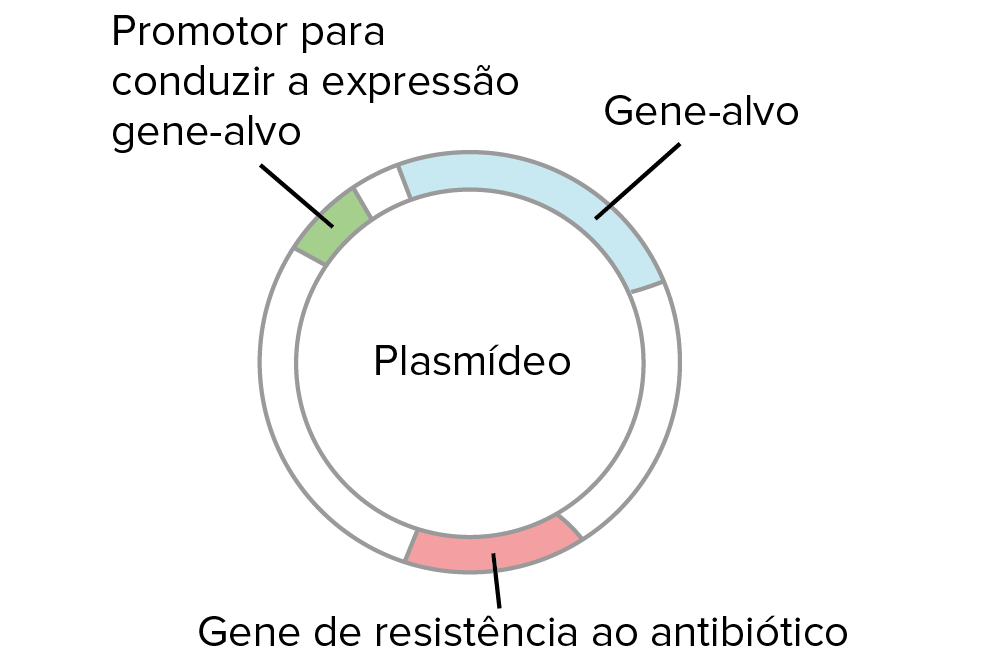
- Terapia gênica A terapia gênica é uma técnica emergente usada para tratar doenças genéticas causadas por um gene defeituoso. Ela funciona fornecendo às células do corpo o gene do DNA "deficiente" . Por exemplo, na doença genética fibrose cística, o gene não possui a capacidade de produzir canais de íons cloreto nos pulmões. Em um recente experimento clínico de terapia gênica, uma cópia do gene funcional foi inserida em uma molécula de DNA circular chamada plasmídeo que foi então fornecida às células pulmonares dos pacientes em esferas de lipídios (na forma de um spray)cubed.Nesse exemplo, componentes biológicos de fontes diferentes (gene humano, plasmídeo proveniente de bactéria) foram combinados para fazer um novo produto que ajudou a manter a função pulmonar dos pacientes com fibrose cística.
Como mostram esses exemplos, a biotecnologia é usada na produção de produtos encontrados na vida cotidiana, tais como o álcool e a penicilina. Ela também pode ser usada para desenvolver novos tratamentos médicos, tais como a terapia gênica para tratamento da fibrose cística. A biotecnologia tem outras aplicações em áreas como a produção de alimentos e a remediação (limpeza) da poluição ambiental.
O que é tecnologia do DNA?
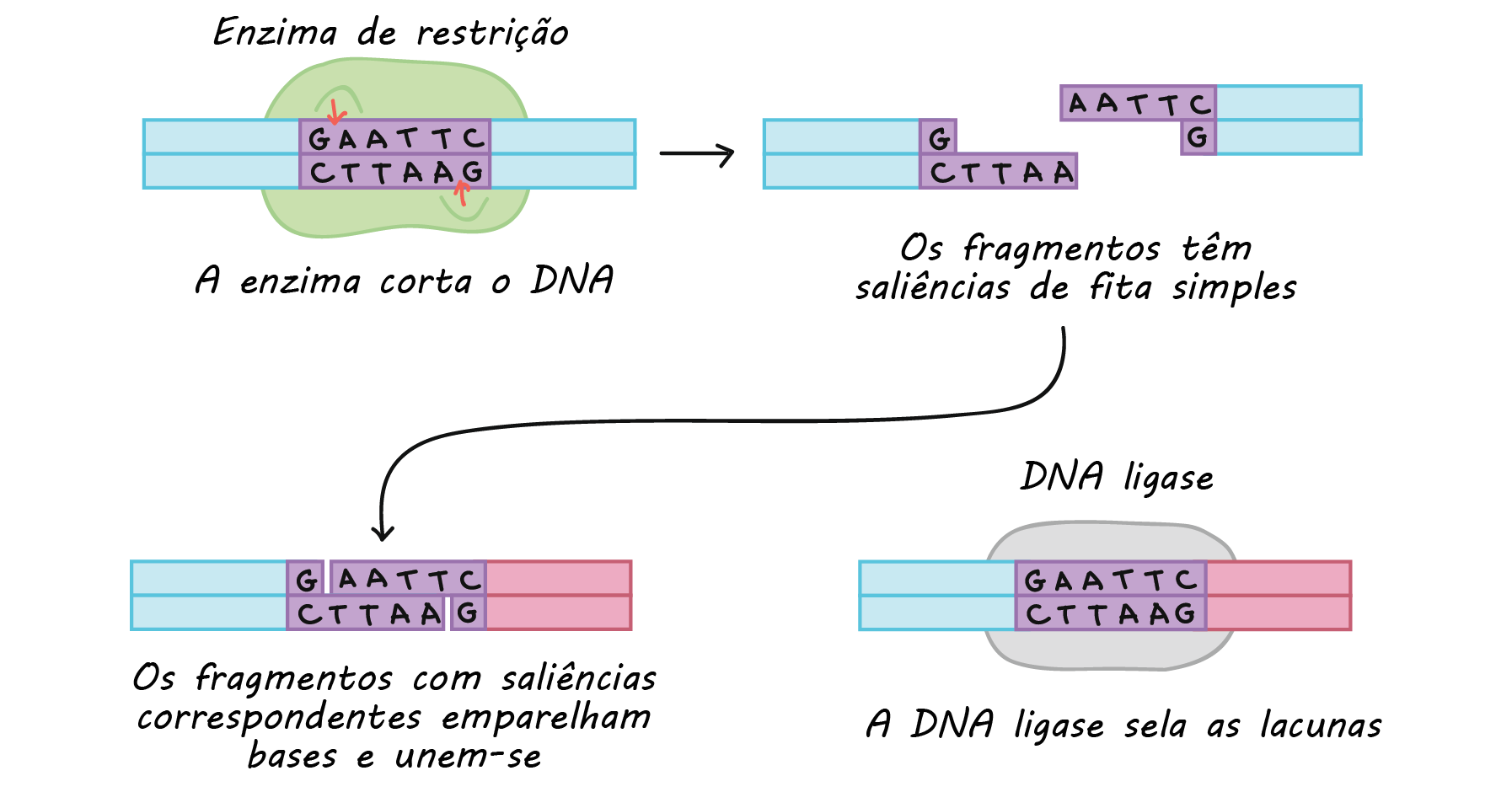
Muitos exemplos da biotecnologia moderna dependem da capacidade de analisar, manipular, cortar e colar pedaços do DNA. Ocasionalmente, as manobras de sequenciamento e manipulação de DNA são chamadas de tecnologia do DNAstart superscript, 4, end superscript. Por exemplo, no experimento da terapia gênica para a fibrose cística os pesquisadores usaram técnicas de manipulação de DNA para inserir o gene do canal de cloreto em uma parte do DNA transportador (um vetor) que permitiu a expressão gênica nas células pulmonares humanas.
A tecnologia do DNA é importante para a biologia geral e aplicada (prática). Por exemplo, uma técnica usada para fazer muitas cópias de uma sequência de DNA chamada reação em cadeia da polimerase (PCR) é usada em muitos testes de diagnóstico médico e aplicações forenses, bem como em pesquisas de laboratório geral.
Exemplos de tecnologia do DNA
Vejamos alguns exemplos de técnicas de análise e manipulação de DNA comumente usadas na biologia molecular moderna. Você pode usar os links abaixo para encontrar informações mais detalhadas sobre estas técnicas.
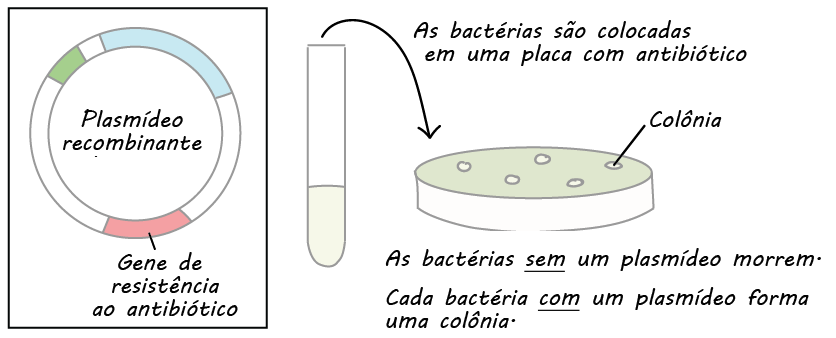
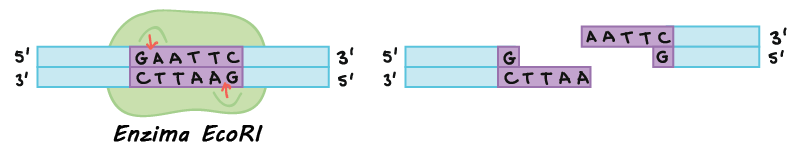
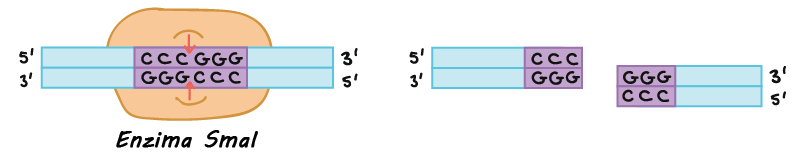
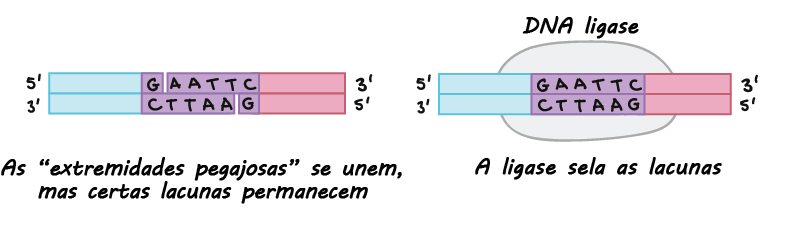
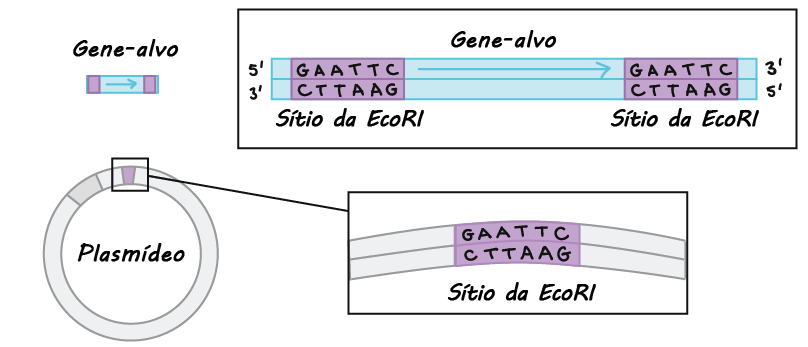
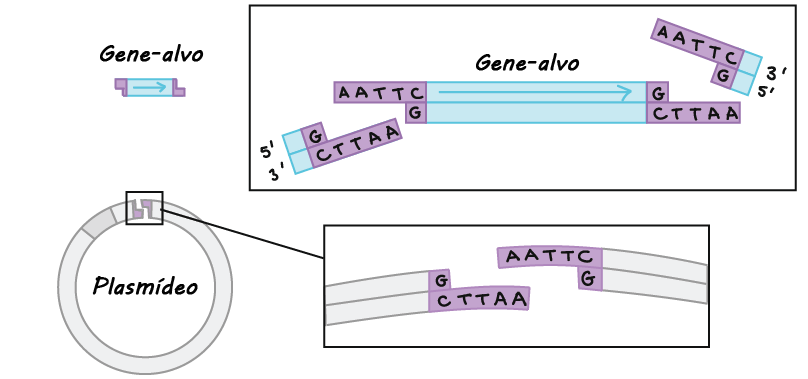
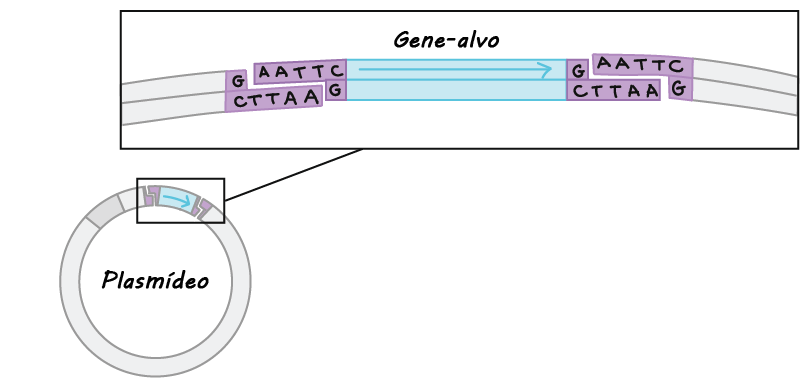
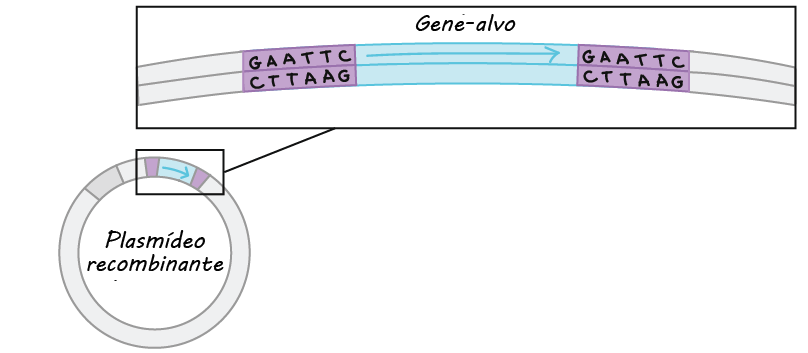
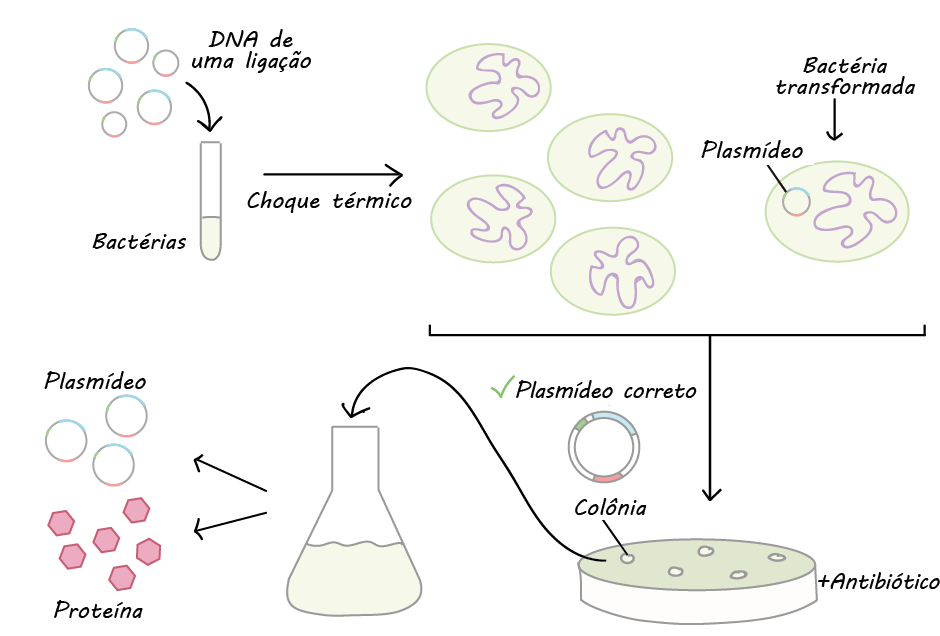
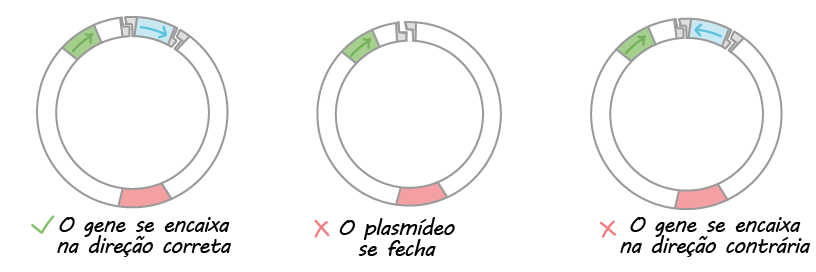
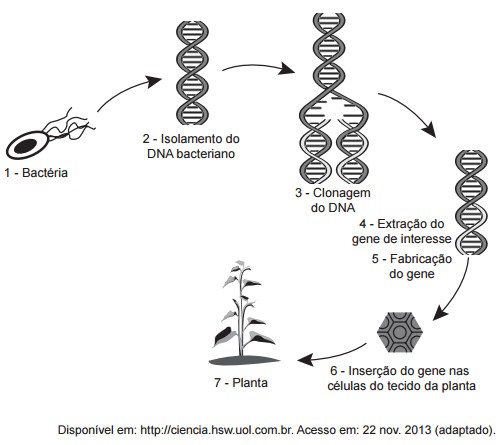
- Clonagem do DNA. Na clonagem do DNA, os pesquisadores "clonam" – fazem muitas cópias do fragmento de DNA de interesse, tal como um gene. Em muitos casos, a clonagem do DNA envolve a inserção do gene de interesse em uma molécula de DNA circular chamada plasmídeo. O plasmídeo pode ser replicado nas bactérias, fazendo muitas cópias do gene de interesse. Em alguns casos, o gene é expresso também nas bactérias pela formação de uma proteína (por exemplo, a insulina usada pelos diabéticos).
- Reação em cadeia da polimerase (PCR). A reação em cadeia da polimerase é a técnica de manipulação do DNA mais amplamente utilizada, com aplicações em quase todas as áreas da biologia moderna. As reações da PCR produzem várias cópias da sequência de DNA de interesse, a partir da sequência de um DNA modelo. Essa técnica pode ser usada para fazer muitas cópias de um DNA que está presente em pequenas quantidades (por exemplo, uma gotícula de sangue na cena do crime).
- Eletroforese em gel. A eletroforese em gel é uma técnica usada para visualizar os fragmentos de DNA (exame direto). Por exemplo, os pesquisadores podem analisar os resultados da PCR examinando os fragmentos de DNA que ela produz na reação em um gel. A eletroforese em gel separa os fragmentos de DNA com base em seu tamanho, em seguida os fragmentos são pigmentados com um corante que possibilita a visualização pelo pesquisador.
- Sequenciamento de DNA. O sequenciamento de DNA consiste em determinar a sequência das bases dos nucleotídeos (As, Ts, Cs e Gs) na molécula de DNA. Em alguns casos, apenas um fragmento de DNA é sequenciado por vez, enquanto em outros casos muitos fragmentos de DNA (como um genoma inteiro) podem ser sequenciados conjuntamente.
O genoma é todo o DNA de um organismo.
Em eucariontes, os quais possuem um núcleo celular para conter o seu DNA, a palavra genoma é normalmente usada para se referir ao genoma nuclear (DNA encontrado no núcleo), o que exclui o DNA encontrado nas organelas tais como os cloroplastos ou mitocôndrias.
Veja nos links como essas técnicas funcionam mais detalhadamente. Veja também exemplos de uso das técnicas em pesquisa, medicina e outras aplicações práticas.
Biotecnologia e novas questões éticas
A biotecnologia tem potencial para trazer benefícios às pessoas e à sociedade, mas também pode ter efeitos negativos ou consequências não intencionais. Isto é verdade em todas as formas de tecnologia, não só na biotecnologia. Contudo, a biotecnologia pode oferecer tipos de benefícios diferentes, apresentando tipos de dilemas diferentes das outras formas de tecnologia.
É importante que as inovações biotecnológicas (assim como outras inovações tecnológicas) sejam cuidadosamente testadas e analisadas antes de serem liberadas para uso geral. Os ensaios clínicos e a regulamentação governamental ajudam a garantir que os produtos biotecnológicos colocados no mercado sejam seguros e eficazes. No entanto, às vezes surgem novas informações que obrigam as empresas e agências governamentais a reconsiderar a segurança ou utilidade de uma inovação. É o caso quando uma determinada medicação é retirada do mercado.
Ademais, as inovações biotecnológicas podem levantar novas questões éticas sobre se utilizar ou não da informação, das técnicas e do conhecimento.
- Algumas delas dizem respeito à privacidade e não discriminação. Por exemplo, sua companhia de seguro de saúde deveria lhe cobrar mais se você tivesse uma variante de gene que o tornasse propenso a desenvolver alguma doença? Como você se sentiria se sua escola ou empregador tivesse acesso ao seu genoma?
- Outras questões são relativas a segurança, efeitos na saúde e impactos ecológicos das biotecnologias. Por exemplo, cultivares geneticamente modificados para produzir seu próprio inseticida reduzem a necessidade de pulverização química, mas também suscitam preocupações caso as plantas escapem para a vida selvagem ou cruzem com espécies locais (potencialmente causando consequências ecológicas não intencionais).
- A biotecnologia pode proporcionar conhecimentos que criam dilemas difíceis para os indivíduos. Por exemplo, um casal descobre no teste pré-natal que seu feto tem uma doença genética. Ou ainda, por curiosidade uma pessoa tem o seu genoma sequenciado e descobre que vai desenvolver uma doença genética incurável tardia, tal como a doença de Huntington.
A pesquisa científica e desenvolvimento podem disponibilizar novas informações, técnicas e conhecimentos. No entanto, a ciência sozinha não pode responder a perguntas sobre como essas técnicas devem ou não ser usadas. É importante que todos os membros da sociedade sejam ouvidos no debate sobre como as invenções e produtos da biotecnologia podem afetar nossa vida cotidiana.
Eduque-se e compartilhe sua opinião
Compreender a biologia básica por trás de qualquer forma de biotecnologia é o primeiro passo importante para julgar seus benefícios e possíveis riscos. As informações desta seção lhe ajudarão a iniciar a montagem do seu conjunto de ferramentas para compreender e avaliar as novas invenções da biotecnologia.
Se você está curioso sobre algum tipo específico de biotecnologia ou preocupado com suas possíveis consequências, uma ótima ideia é pesquisar por conta própria. Procure em fontes confiáveis e não tendenciosas. Se houver controvérsia. tente entender as opiniões dos dois lados. Certifique-se de que você compreende a ciência por trás da invenção, o que se sabe (e não se sabe) sobre ela e quais são os prós e contras. Assim, você poderá formar uma opinião bem embasada e ponderada sobre como a tecnologia deve ser usada ou não.











Nenhum comentário:
Postar um comentário