2° Ano E Biologia Profa Solange
Período para entrega: Até 14/10/2020
Unidade Temática: DNA como material genético- DNA- Receita da Vida e seu Código.
ATIVIDADE DE RECUPERAÇÃO E APROFUNDAMENTO
Instruções:
1- Assistir as vídeo aulas (Khan Academy).
2- Fazer a Leitura e Interpretação de Texto.
3- Responder a atividade: Exercícios de Fixação.
4- Postar no Blogger e enviar para o e-mail da professora: solangestandbyme@gmail.com
Herança poligênica e efeitos ambientais
Como é a herança da altura?
- Variação contínua. Diferentemente das ervilhas de Mendel, os seres humanos não vêm em apenas duas nítidas variedades "baixo" e "alto". De fato, eles não vêm nem mesmo em quatro, oito ou dezesseis alturas. Ao contrário, é possível termos humanos de muitas alturas diferentes, e a altura pode variar em incrementos de centímetros ou frações de centímetros1start superscript, 1, end superscript.
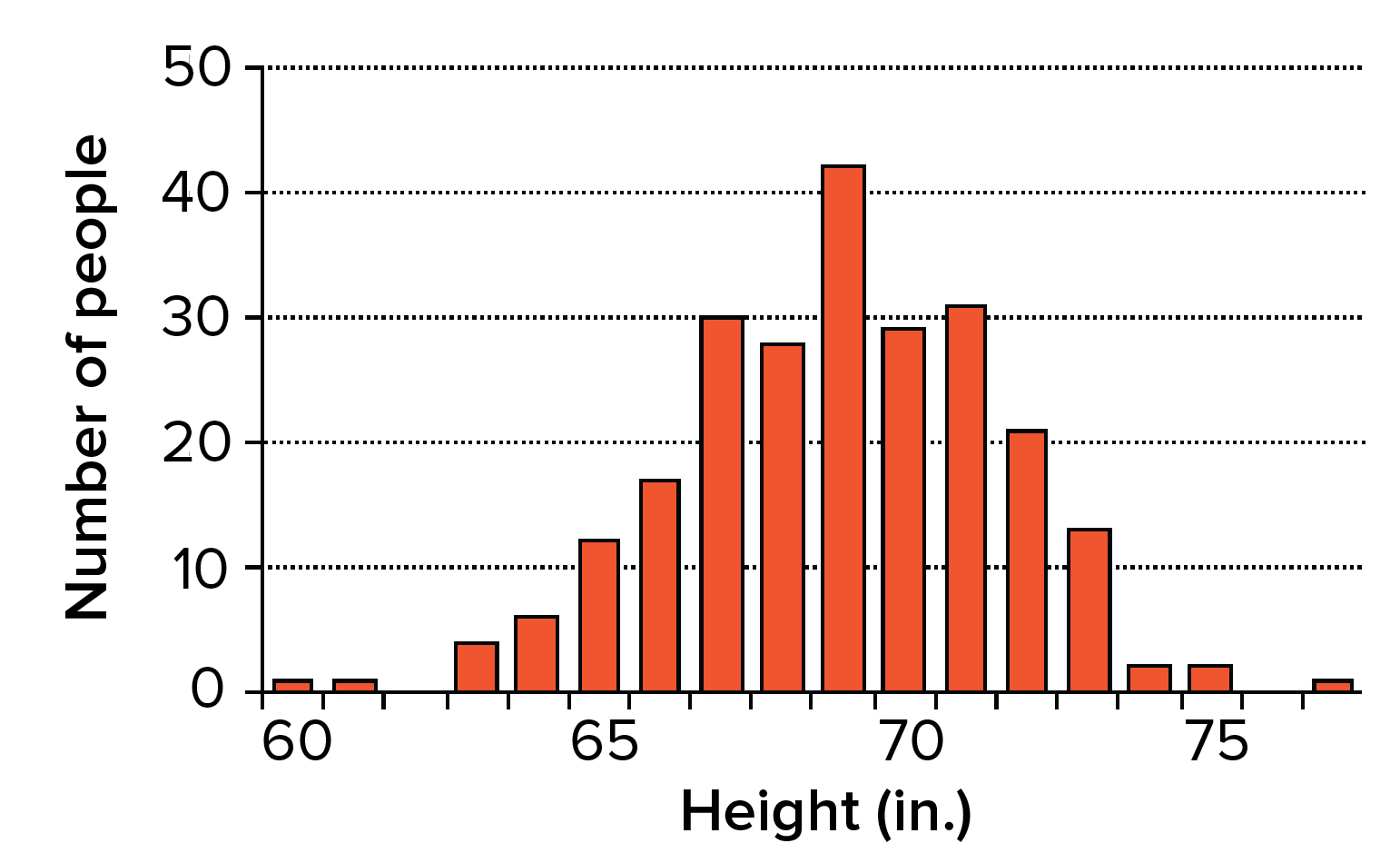 Histograma mostrando as alturas, em polegadas, de alunos homens do terceiro ano do Ensino Médio de uma amostra. O histograma tem forma mais ou menos de sino, com apenas alguns indivíduos nas caudas (60 polegadas e 77 polegadas) e vários indivíduos no meio, aproximadamente 69 polegadas.As alturas de um grupo de formandos do Ensino Médio. Imagem modificada de "Continuous variation: Quantitative traits," by J. W. Kimball (CC BY 3.0)
Histograma mostrando as alturas, em polegadas, de alunos homens do terceiro ano do Ensino Médio de uma amostra. O histograma tem forma mais ou menos de sino, com apenas alguns indivíduos nas caudas (60 polegadas e 77 polegadas) e vários indivíduos no meio, aproximadamente 69 polegadas.As alturas de um grupo de formandos do Ensino Médio. Imagem modificada de "Continuous variation: Quantitative traits," by J. W. Kimball (CC BY 3.0) - Um complexo padrão de herança . Você deve ter reparado que pais altos podem ter um filho baixo, pais baixos podem ter um filho alto, e dois pais de tamanhos diferentes podem ou não ter um filho mediano. Também, irmãos com os mesmos pais podem apresentar uma variação de alturas, que não se enquadram em categorias distintas.
Herança poligênica
Efeitos ambientais
Expressividade variável, penetrância incompleta

Referências:
- Wood, A. R., Esko, T., Yang, J., Vedantam, S., Pers, T. H., Gustafsson, S., ... Frayling, T. M. (2014). Defining the role of common variation in the genomic and biological architecture of adult human height. Nature Genetics, 46, 1173-1186. http://dx.doi.org/10.1038/ng.3097.
- White, D. and Rabago-Smith, M. (2011). Genotype-phenotype associations and human eye color. Journal of Human Genetics, 56, 5-7. http://dx.doi.org/10.1038/jhg.2010.126.
- Department of Agronomy, Iowa State University. (2016). Inheritance of a quantitative trait. Acesso em 26 de Julho, 2016 em https://masters.agron.iastate.edu/classes/527/lesson07/detail/kernelColor.html.
- Armstrong, W. P. (n.d.). Continuous variation and Rh blood factor. In Wayne's Word. Acesso em 26 de Julho, 2016 em http://waynesword.palomar.edu/lmexer5.htm.
- Bergmann, D. C. (2011). Variations on and complications of the basic Mendelian rules. In Genetics lecture notes. Biosci41, Stanford University, 10.
- PAH. (2008). In Genetics home reference.Disponível em http://ghr.nlm.nih.gov/gene/PAH.
- Phenylketonuria. (2012). In Genetics home reference. Disponível em http://ghr.nlm.nih.gov/condition/phenylketonuria.
- National Cancer Institute. (2015, August 14). Retinoblastoma treatment – for health professionals. In Cancer types. Disponível em http://www.cancer.gov/types/retinoblastoma/hp/retinoblastoma-treatment-pdq.
- Carr, S. M. (2014). Penetrance versus expressivity. Disponivel em https://www.mun.ca/biology/scarr/Penetrance_vs_Expressivity.html.
- Kelly, Jane. (2015, September 14). Retinoblastoma. In OMIM. Disponível em http://www.omim.org/entry/180200.
- Griffiths, A. J. F., Miller, J. H., Suzuki, D. T., Lewontin, R. C., and Gelbart, W. M. (2000). Penetrance and expressivity. In An introduction to genetic analysis (7th ed.). Disponível em http://www.ncbi.nlm.nih.gov/books/NBK22090/.
Referências:
Ligação genética e mapeamento
Pontos Principais:
- Quando os genes são encontrados em cromossomos diferentes ou bem distantes mas no mesmo cromossomo, eles ordenam-se independentemente e são ditos ser não ligados.
- Quando os genes estão bem próximos no mesmo cromossomo, diz-se que estão ligados. Isso significa que os alelos, ou as versões gênicas, juntas em um cromossomo serão mais frequentemente herdadas como uma unidade do que separadamente.
- Podemos ver se dois genes estão ligados, e quão próximos estão, usando informação de cruzamentos genéticos para calcular a frequência de recombinação.
- Encontrando as frequências de recombinação para muitos pares de genes, podemos formar mapas de ligação que mostram a ordem e as distâncias relativas dos genes nos cromossomos.
Introdução
O que é ligação genética?
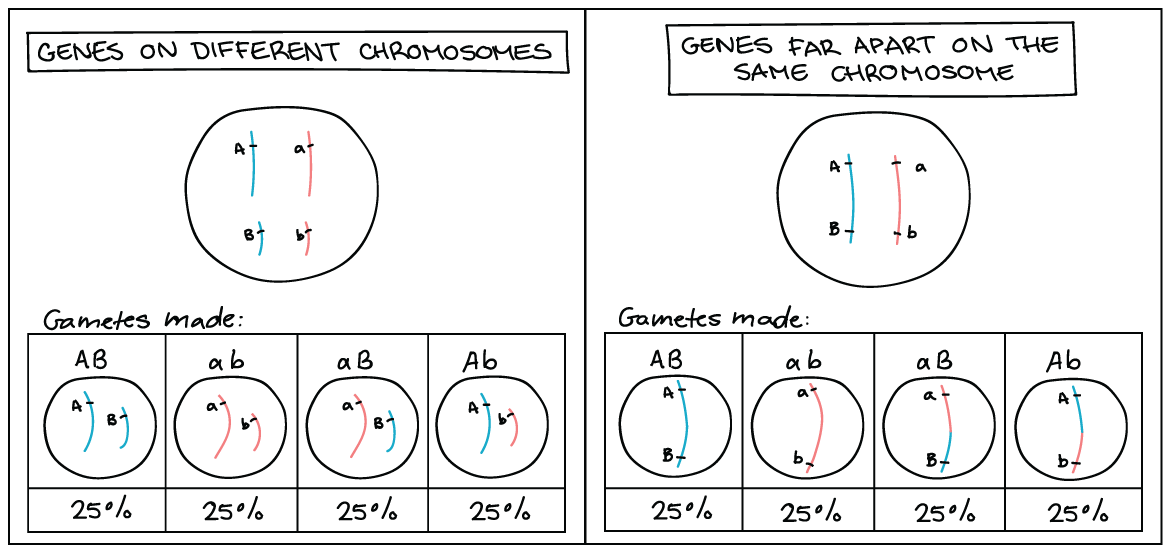
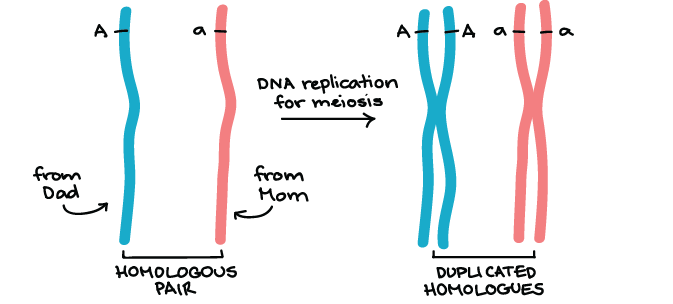

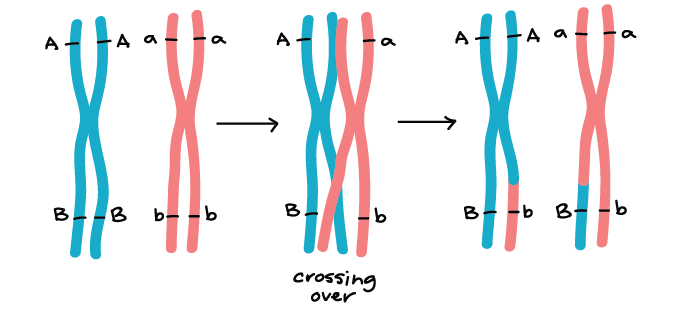
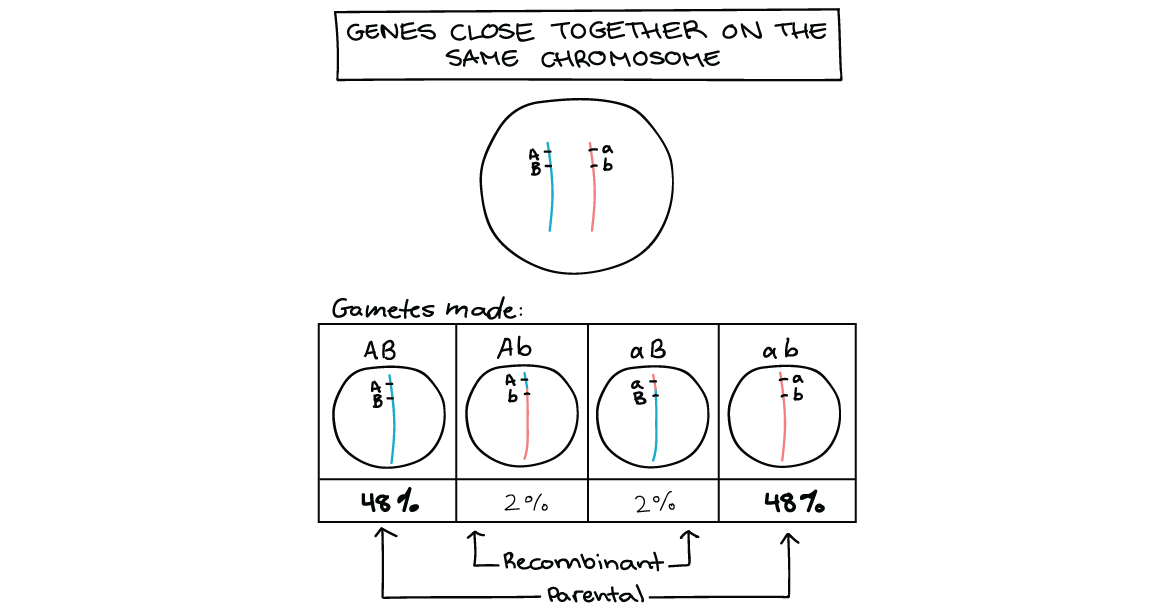
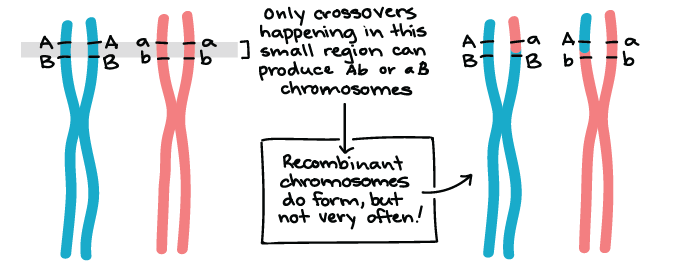
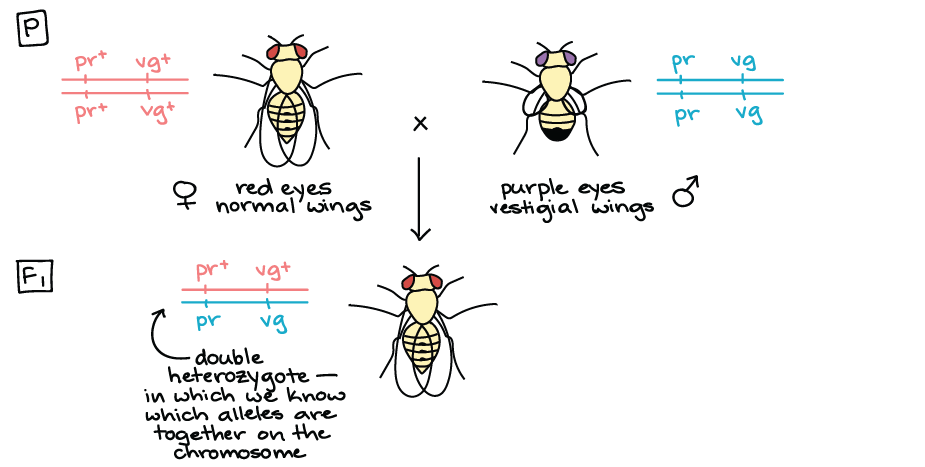
Determinação da frequência de recombinação
- O gene roxo, com o alelo dominante pr+start superscript, plus, end superscript que especifica o fenótipo normal, olhos vermelhos; e o alelo recessivo pr que especifica olhos roxos.
- O gene vestigial, com o alelo dominante vg+start superscript, plus, end superscript que especifica o fenótipo normal, asas longas; e o alelo recessivo vg que especifica asas curtas, "vestigiais".
- Homozigoto significa que o organismo tem dois alelos iguais. Por exemplo: uma mosca b b seria homozigota.
- Heterozigoto significa que o organismo tem dois alelos diferentes. Por exemplo: uma mosca b b+start superscript, plus, end superscript seria heterozigota.
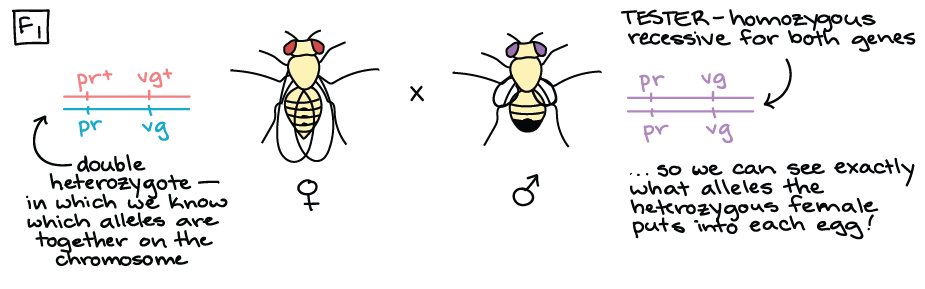
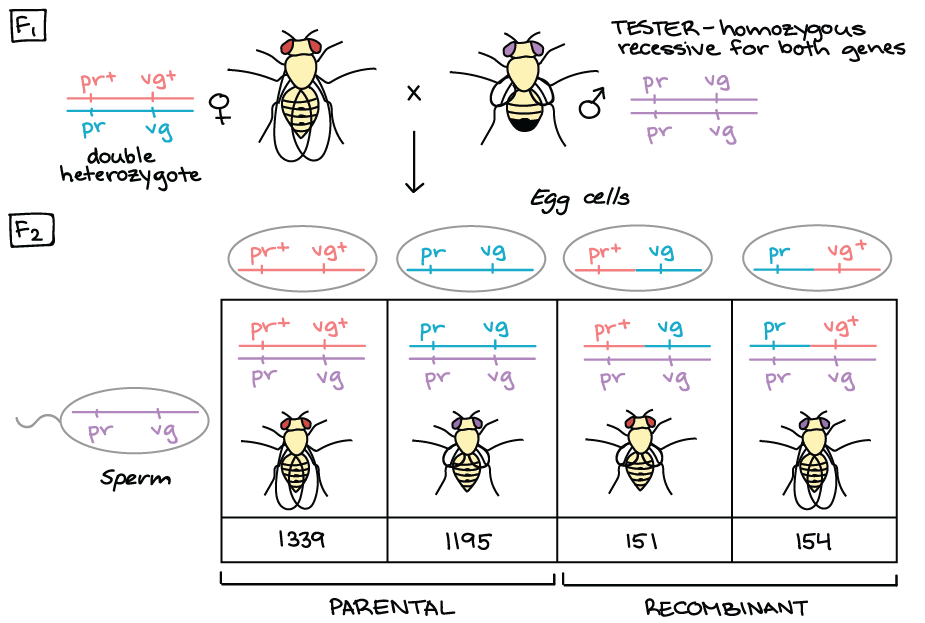
Frequência de recombinação e mapas de ligação
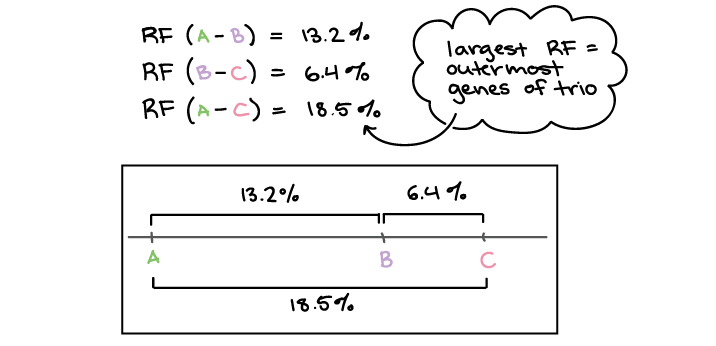

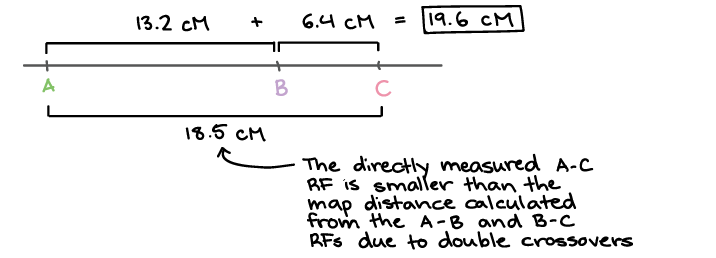
Trabalhos citados
- Jyoti Madhusoodanan, "Human Gene Set Shrinks Again," The Scientist, última modificação em 8 de julho de 2014, http://www.the-scientist.com/?articles.view/articleNo/40441/title/Human-Gene-Set-Shrinks-Again/.
- "Genomic DNA Sequencing Project FAQ," Berkeley Drosophila Genome Project, última modificação em 8 de outubro de 2015, http://www.fruitfly.org/sequence/faq.html.
- Dominique C. Bergmann, Genetics Lecture Notes (Stanford: BioSci 41, 2011), 27.
- Dominique C. Bergmann, Genetics Lecture Notes (Stanford: BioSci 41, 2011), 34.
Referências
Bergmann, Dominique C. Genetics Lecture Notes. Stanford: BioSci 41, 2011.Campbell, Neil A., Jane B. Reece, Lisa A. Urry, Michael L. Cain, Steven A. Wasserman, Peter V. Minorsky, and Robert B. Jackson. "Linked Genes Tend to be Inherited Together Because They Are Located Near Each Other on the Same Chromosome." In Campbell Biology, 292-296. 8th ed. San Francisco: Benjamin Cummings, 2008."Genomic DNA Sequencing Project FAQ." Berkeley Drosophila Genome Project. Última modificação em 8 de outubro de 2015. http://www.fruitfly.org/sequence/faq.html#seq-01.Kimball, John W. "Genetic Linkage and Genetic Maps." Kimball's Biology Pages. Última modificação em 24 de abril de 2014. http://www.biology-pages.info/L/Linkage.html.Madhusoodanan, Jyoti. "Human Gene Set Shrinks Again." The Scientist. Última modificação em 8 de julho de 2014. http://www.the-scientist.com/?articles.view/articleNo/40441/title/Human-Gene-Set-Shrinks-Again/.OpenStax College, Biology. "Chromosomal Theory and Genetic Linkage." OpenStax CNX. Última modificação em 27 de maio de 2016. http://cnx.org/contents/GFy_h8cu@10.53:qdHTV9py@8/Chromosomal-Theory-and-Genetic.Purves, William K., David Sadava, Gordon H. Orians, and H. Craig Heller. "Genes and Chromosomes." In Life: The Science of Biology, 202-205. 7th ed. Sunderland, MA: Sinauer Associates, 2003.Raven, Peter H., George B. Johnson, Kenneth A. Mason, Jonathan B. Losos, and Susan R. Singer. "Genetic Mapping." In Biology. 244-248. 10th ed., AP ed. New York: McGraw-Hill, 2014.Reece, Jane B., Lisa A. Urry, Michael L. Cain, Steven A. Wasserman, Peter V. Minorsky e Robert B. Jackson. "Phylogeny and the Tree of Life." In Campbell Biology, 547-558. 10th ed. San Francisco: Pearson, 2011.
Inativação do X
Como fêmeas humanas XX (e outras fêmeas mamíferas) inativam um dos seus cromossomos X em cada célula. Desordens do número de cromossomos sexuais: síndromes de Klinefelter, triplo X e Turner.Introdução
Ter cromossomos a mais ou faltando geralmene não é uma coisa boa. Inclusive, para a maioria dos cromossomos, ter uma cópia a mais ou a menos é letal para os humanos (causando a morte do embrião no início do desenvolvimento).Entretanto, fêmeas humanas têm dois cromossomos X (XX), enquanto machos humanos têm apenas um (XY). Por que não causa problemas os homens terem apenas uma cópia do cromossomo X, enquanto as mulheres têm duas?Inativação do X
Na realidade, o nível de atividade gênica produzida por um único cromossomo X é a "dosagem" normal para um humano. Homens têm essa dosagem porque, bem, eles têm apenas um cromossomo X! As mulheres têm a mesma dosagem por um motivo diferente: elas desligam um de seus dois cromossomos X em um processo chamado inativação do X.Na inativação do X, um cromossomo X é compactado (ou, como meu professor de introdução à biologia gostava de dizer, "amassado em uma bolinha"), para formar uma estrutura pequena e densa chamada de corpúsculo de Barr. A maioria dos genes no corpúsculo de Barr são inativos, o que significa que eles não são transcritos. O processo de inativação do X foi descoberto pela geneticista britânica Mary F. Lyon, e algumas vezes é chamado de lyonização em sua homenagem1start superscript, 1, end superscript. Fotografia de Mary F. Lyon, a geneticista que descobriu a inativação do X._Imagem modificada de "Photo of an English geneticist, Mary Frances Lyon," por Jane Gitschier (CC BY 2.5)._Uma mulher tem dois cromossomos X, um do pai e um da mãe. Qual deles ela irá inativar? A inativação do X é um processo aleatório que ocorre separadamente em células individuais durante o desenvolvimento embrionário. Uma célula pode desligar o X paterno, enquanto sua vizinha pode desligar o X materno ao invés. Todas as células descendentes de uma dessas células originais irão manter o mesmo padrão de inativação do X.Nota interessante: se você fosse um canguru, o que eu acabei de dizer não seria verdade! Nos cangurus e outros marsupiais, é sempre o cromossomo X paterno que passa pela inativação do X.2squared
Fotografia de Mary F. Lyon, a geneticista que descobriu a inativação do X._Imagem modificada de "Photo of an English geneticist, Mary Frances Lyon," por Jane Gitschier (CC BY 2.5)._Uma mulher tem dois cromossomos X, um do pai e um da mãe. Qual deles ela irá inativar? A inativação do X é um processo aleatório que ocorre separadamente em células individuais durante o desenvolvimento embrionário. Uma célula pode desligar o X paterno, enquanto sua vizinha pode desligar o X materno ao invés. Todas as células descendentes de uma dessas células originais irão manter o mesmo padrão de inativação do X.Nota interessante: se você fosse um canguru, o que eu acabei de dizer não seria verdade! Nos cangurus e outros marsupiais, é sempre o cromossomo X paterno que passa pela inativação do X.2squaredExemplo de inativação do X: o gato malhado
Um exemplo clássico da inativação do X é visto em gatos. Se uma gata for heterozigota para alelos de pelagem preta e amarela encontrados no cromossomo X, ela irá inativar os dois X (e, assim, os dois alelos do gene para cor de pelo) aleatoriamente em diferentes células durante o desenvolvimento.O resultado é um padrão de pelagem semelhante a um casco de tartaruga, composto de tufos alternantes de pelo preto e amarelo. Os tufos pretos vêm de grupos de células nas quais o X com o alelo para a cor preta está ativo, enquanto os tufos amarelos vêm das células nas quais o X com o alelo para a cor amarela está ativo.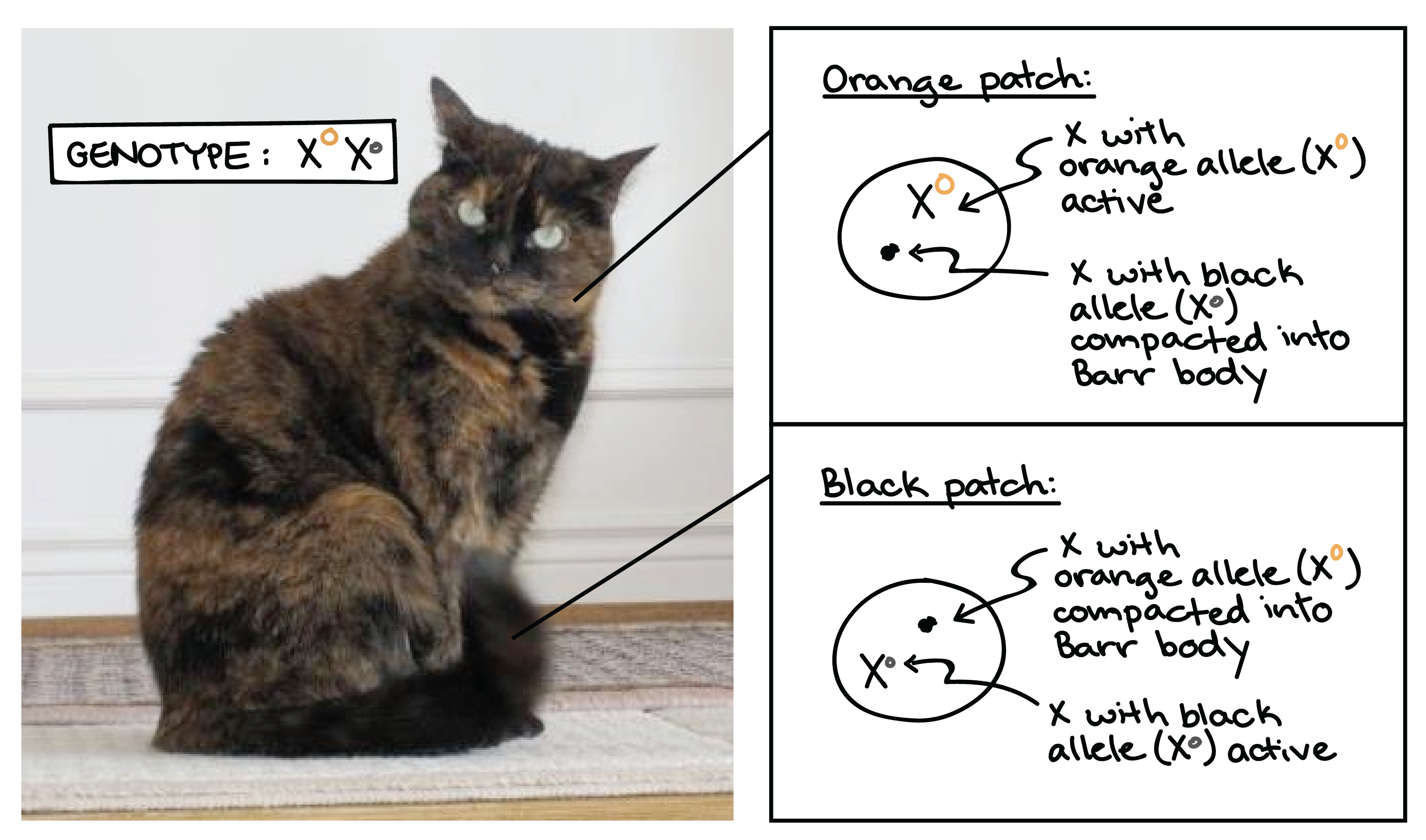 Imagem de um gato malhado, ilustrando o processo de inativação do X que causa as manchas de diferentes cores na sua pelugem. O gato tem uma mistura de manchas pretas e laranjas, algumas menores e outras maiores. O genótipo do gato é XOXostart text, X, end text, start superscript, O, end superscript, start text, X, end text, start superscript, o, end superscript, onde o O grande representa o laranja e o o pequeno representa o preto.
Imagem de um gato malhado, ilustrando o processo de inativação do X que causa as manchas de diferentes cores na sua pelugem. O gato tem uma mistura de manchas pretas e laranjas, algumas menores e outras maiores. O genótipo do gato é XOXostart text, X, end text, start superscript, O, end superscript, start text, X, end text, start superscript, o, end superscript, onde o O grande representa o laranja e o o pequeno representa o preto.- As manchas laranjas são formadas por células que possuem o X com o alelo laranja (XOstart text, X, end text, start superscript, O, end superscript) ativo, enquanto o X com o alelo preto (Xostart text, X, end text, start superscript, o, end superscript) está condensado como corpúsculo de Barr.
- As manchas pretas são formadas por células que possuem o X com o alelo preto (XOstart text, X, end text, start superscript, O, end superscript) ativo, enquanto o X com o alelo laranja (Xostart text, X, end text, start superscript, o, end superscript) está condensado como corpúsculo de Barr.
_Imagem modificada de “6-year old tortoiseshell cat," by Michael Bodega (public domain)._Embora raramente seja tão fácil de se ver como na pelagem dos gatos, fêmeas humanas também são "mosaico" para quaisquer genes que estejam presentes em diferentes alelos de seus dois cromossomos X.SAIBA MAIS:Agora, você pode estar se perguntando por que as mulheres heterozigotas para um alelo recessivo ligado ao X não exibem a condição associada. Afinal, aproximadamente metade das suas células irá inativar o alelo normal, deixando apenas o alelo causador da doença ativo.Em diversos casos, parece que ter 50%50, percent das células com cópia do gene normal é suficiente para apresentar um fenótipo normal ou muito próximo do normal.Um bom exemplo disso vem da condição ligada ao X em que o indivíduo tem deficiência na visão para as cores vermelho e verde (daltonismo). As mulheres que são portadoras dos alelos para essa condição apresentam uma mistura de células fotorreceptoras funcionais e não funcionais em seus olhos . Entretanto, as células funcionais permitem a elas perceberem o vermelho e o verde bem o bastante para visualizarem normalmente no dia a dia. Em testes laboratoriais mais sensíveis, essas mulheres, de fato, apresentam deficiências sutis na visão quando comparadas com mulheres que não apresentam o alelo3cubed.Aneuploidias dos cromossomos sexuais
Quando um organismo tem uma cópia a mais ou faltando de um cromossomo, é considerado aneuploide. Aneuploidias envolvendo autossomos (cromossomos não sexuais), especialmente os grandes, são geralmente tão danosos para o desenvolvimento que um embrião aneuploide não consegue sobreviver ao nascimento.Aneuploidias dos cromossomos X, contudo, tendem a ser muito menos danosas, apesar do fato de o X ser um cromossomo grande. Isso se deve em grande parte à inativação do X. Embora o propósito do sistema de inativação do X seja desligar o segundo X de uma fêmea XX, ele também pode fazer um bom trabalho desligando mais cromossomos X se estiverem presentes.Exemplos de aneuploidias do cromossomo X incluem:- Síndrome do Triplo X, na qual uma mulher tem um genótipo XXX, o qual ocorre em aproximadamente 11 em cada 1,1, comma000000 recém-nascidas4start superscript, 4, end superscript. Mulheres com um genótipo XXX têm características sexuais femininas e são férteis (capazes de ter crianças). Em alguns casos, a síndrome do triplo X pode estar associada a dificuldades de aprendizado, desenvolvimento tardio de habilidades motoras na criança e problemas com tônus muscular4start superscript, 4, end superscript.
- Síndrome de Klinefelter, na qual os homens apresentam um cromossomo X extra, levando a um genótipo XXY. (Em casos raros, a síndrome de Klinefelter pode envolver vários Xs extras, levando a um genótipo XXXY ou XXXXY.) Homens afetados podem ser inférteis ou desenvolver uma menor densidade de pelos corporais e faciais que outros homens. A síndrome de Klinefelter afeta aproximadamente 11 em cada 500500 a 1,1, comma000000 recém-nascidos do gênero masculino5start superscript, 5, end superscript.
Como as mulheres, homens XXY com a síndrome de Klinefelter irão converter um de seus cromossomos X a corpúsculo de Barr em cada célula. Mulheres triplo X (bem como homens Klinefelter com mais de dois cromossomos X) neutralizam seus Xs extra formando corpúsculos de Barr adicionais. Por exemplo, haveria dois corpúsculos de Barr em uma célula de uma mulher XXX ou de um homem XXXY. Diagrama dos cromossomos sexuais e formação do corpúsculo de Barr em indivíduos humanos com diferentes genótipos dos cromossomos sexuais.Feminino XX: um X ativo. Um corpúsculo de Barr. Masculino XY: um X ativo, um Y, nenhum corpúsculo de Barr. Masculino XXY (síndrome de Klinefelter): um X ativo, um Y, um corpúsculo de Barr. Feminino XXX (síndrome de Triplo X): um X ativo, dois corpúsculos de Barr.Na síndrome de Turner, a mulher é desprovida de parte, ou mesmo da totalidade de um de seus cromossomos X (deixando-a com apenas um X funcional). Pessoas com essa desordem se desenvolvem como mulher, mas comumente têm baixa estatura e podem exibir sintomas como infertilidade e dificuldades de aprendizagem. A síndrome de Turner ocorre em aproximadamente em 11 a cada 2.2, point500500 recém-nascidas6start superscript, 6, end superscript. Ela geralmente tem efeitos relativamente amenos pois os humanos normalmente só têm um cromossomo X ativo nas células do corpo de qualquer jeito.
Diagrama dos cromossomos sexuais e formação do corpúsculo de Barr em indivíduos humanos com diferentes genótipos dos cromossomos sexuais.Feminino XX: um X ativo. Um corpúsculo de Barr. Masculino XY: um X ativo, um Y, nenhum corpúsculo de Barr. Masculino XXY (síndrome de Klinefelter): um X ativo, um Y, um corpúsculo de Barr. Feminino XXX (síndrome de Triplo X): um X ativo, dois corpúsculos de Barr.Na síndrome de Turner, a mulher é desprovida de parte, ou mesmo da totalidade de um de seus cromossomos X (deixando-a com apenas um X funcional). Pessoas com essa desordem se desenvolvem como mulher, mas comumente têm baixa estatura e podem exibir sintomas como infertilidade e dificuldades de aprendizagem. A síndrome de Turner ocorre em aproximadamente em 11 a cada 2.2, point500500 recém-nascidas6start superscript, 6, end superscript. Ela geralmente tem efeitos relativamente amenos pois os humanos normalmente só têm um cromossomo X ativo nas células do corpo de qualquer jeito.- Este artigo é uma versão modificada derivada de "Chromosomal basis of inherited disorders," by OpenStax College, Biology, CC BY 4.0. Baixe o artigo original gratuitamente em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@10.53.Este artigo está autorizado sob licença CC BY-NC-SA 4.0.
Referências:
- Mary F. Lyon. (2015, November 22). Acesso em 12 Dezembro, 2015 Disponível em: https://en.wikipedia.org/wiki/Mary_F._Lyon.
- Kimball, J. W. (2015, December 23). Sex chromosomes. Em Kimball's biology pages. Disponível em http://www.biology-pages.info/S/SexChromosomes.html.
- Chen, Shuai. (2010, March 4). Since only one of a woman's X chromosomes works in a cell, why aren't more women colorblind? [answer]. Em Stanford at the Tech: Understanding genetics. Disponível em http://genetics.thetech.org/ask/ask349.
- Triple X syndrome. (2014). Em Genetics home reference. Disponível em: http://ghr.nlm.nih.gov/condition/triple-x-syndrome.
- Klinefelter syndrome. (2013). Em Genetics home reference. Disponível em http://ghr.nlm.nih.gov/condition/klinefelter-syndrome.
- Turner syndrome. (2012). Em Genetics home reference. Disponível em http://ghr.nlm.nih.gov/condition/turner-syndrome.
Outras referências
Bergmann, D. C. (2011). Mutations, aneuploidy and sex determination. Em Genetics lecture notes (pp. 36-44). Biosci41, Stanford University.Chen, Shuai. (2010, March 4). Since only one of a woman's X chromosomes works in a cell, why aren't more women colorblind? [answer]. Em Stanford at the Tech: Understanding genetics. Disponível em http://genetics.thetech.org/ask/ask349.Deeb, S. S. and Motulsky, A. G. (2015, February 5). Red-green color vision defects. In GeneReviews. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK1301/.Genetic factors and hormones that determine gender. (2007, June 27). Em Human embryology: Organogenesis. Disponível em http://www.embryology.ch/anglais/ugenital/molec02.html.Kimball, J. W. (2015, December 23). Sex chromosomes. Em Kimball's biology pages. Disponível em http://www.biology-pages.info/S/SexChromosomes.html.Klinefelter syndrome. (2013). Em Genetics home reference. Disponível em http://ghr.nlm.nih.gov/condition/klinefelter-syndrome.Krempels, D. M. (n.d.). The genetics of calico cats. Disponível em www.bio.miami.edu/dana/dox/calico.html.Mary F. Lyon. (2015, November 22). Acesso em 12 Dezembro, 2015 Disponível em: https://en.wikipedia.org/wiki/Mary_F._Lyon.OpenStax College, Biology. (2015, May 13). Chromosomal theory and genetic linkage. Em OpenStax CNX. Disponível em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@9.85:64/Chromosomal-Theory-and-GeneticO'Neil, Dennis. (2013). Sex chromosome abnormalities. Em Human chromosomal abnormalities. Disponível em: http://anthro.palomar.edu/abnormal/abnormal_5.htm.OPN1LW. (2015). Em Genetics home reference. Disponível em http://ghr.nlm.nih.gov/gene/OPN1LW.Purves, W. K., Sadava, D. E., Orians, G. H., and Heller, H.C. (2003). Sex determination and sex-linked inheritance. Em Life: The science of biology (7th ed., pp. 125-144). Sunderland, MA: Sinauer Associates.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Alterations of chromosome number or structure cause some genetic disorders. In Campbell biology (10th ed., pp. 304-307). San Francisco, CA: Pearson.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Sex-linked genes exhibit unique patterns of inheritance. Em Campbell biology (10th ed., pp. 205-209). San Francisco, CA: Pearson.Testis determining factor. (2015, Novembro 10). Acessado em Dezembro 11, 2015 em Wikipedia: https://en.wikipedia.org/wiki/Testis_determining_factor.Triple X syndrome. (2014). Em Genetics home reference. Disponível em http://ghr.nlm.nih.gov/condition/triple-x-syndrome.Turner syndrome. (2012). Em Genetics home reference. Disponível em http://ghr.nlm.nih.gov/condition/turner-syndrome.XY sex-determination system. (2015, Novembro 19). Acesso em 11 de dezembro 11, 2015 em Wikipedia: https://en.wikipedia.org/wiki/XY_sex-determination_system. Aneuploidia e rearranjos cromossômicos
- Aneuploidia e não disjunção. Síndrome de Down e doenças correlatas. Rearranjos cromossômicos.
Introdução
Algumas coisas funcionam melhor em pares. Exemplos cotidianos incluem sapatos, luvas e fones de ouvido num aparelho de música. Se você perder um membro do par isto será um problema e pode até ser um problema sério (por exemplo, se você já estiver atrasado para a escola!).Pares também são importantes na genética. A maioria de suas células contém 4646 cromossomos, estruturas semelhantes a bastões feitas de DNA e proteína, que formam 2323 pares que se combinam perfeitamente dois a dois. Esses cromossomos possuem dezenas de milhares de genes, que dizem ao seu corpo como se desenvolver e manter-se funcionando a cada momento de sua vida1start superscript, 1, end superscript.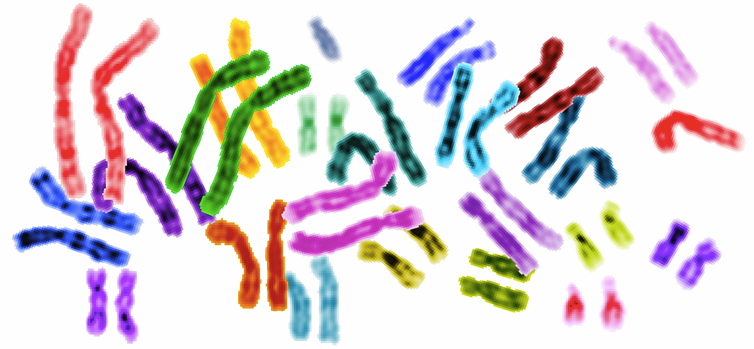 Imagem, colorida artificialmente, dos cromossomos pareados do genoma humano. A imagem ilustra que os cromossomos humanos vêm em pares homólogos, e que cada par é composto de dois cromossomos que se assemelham um ao outro (e têm aparência diferente dos outros cromossomos na célula).Crédito da imagem: "Human genome," by Webridge (CC BY 2.0).Se um par de cromossomos perde ou ganha um membro, ou mesmo parte de um, o equilíbrio delicado do corpo humano pode ser rompido. Neste artigo, iremos examinar como as mudanças no número e na estrutura dos cromossomos acontecem e como elas podem afetar a saúde humana.
Imagem, colorida artificialmente, dos cromossomos pareados do genoma humano. A imagem ilustra que os cromossomos humanos vêm em pares homólogos, e que cada par é composto de dois cromossomos que se assemelham um ao outro (e têm aparência diferente dos outros cromossomos na célula).Crédito da imagem: "Human genome," by Webridge (CC BY 2.0).Se um par de cromossomos perde ou ganha um membro, ou mesmo parte de um, o equilíbrio delicado do corpo humano pode ser rompido. Neste artigo, iremos examinar como as mudanças no número e na estrutura dos cromossomos acontecem e como elas podem afetar a saúde humana.Aneuploidia: cromossomos extra ou faltando
Mudanças no material genético da célula são chamadas de mutações. Em uma forma de mutação as células podem terminar com um cromossomo a mais ou a menos.Cada espécie tem seu número característico de cromossomos, tal como 4646 cromossomos para uma célula típica do corpo humano. Em organismos com dois conjuntos completos de cromossomos, como nos humanos, dá-se a esse número o nome de 2n2, n. Quando um organismo ou uma célula contém 2n2, n cromossomos (ou algum outro múltiplo de nn), ele é chamado de euploide, ou seja, que contém cromossomos corretamente organizados em conjuntos completos (eu- = bom).Se a célula estiver sem um ou mais cromossomos, é chamada de aneuploide (an- = não, "não bom"). Por exemplo, células somáticas humanas com número de cromossomos igual a (2n−1)=45left parenthesis, 2, n, minus, 1, right parenthesis, equals, 45 ou (2n+1)=47left parenthesis, 2, n, plus, 1, right parenthesis, equals, 47 são aneuploides. Da mesma forma, um óvulo ou esperma humano normal tem apenas um conjunto de cromossomos (n=23n, equals, 23). Um óvulo ou esperma com (n−1)=22left parenthesis, n, minus, 1, right parenthesis, equals, 22 ou (n+1)=24left parenthesis, n, plus, 1, right parenthesis, equals, 24 cromossomos é considerado aneuploide.Dois tipos especiais de aneuploidia tem nomes especiais:- Monossomia é quando um organismo tem apenas uma cópia de um cromossomo que deveria estar presente em duas cópias (2n−1)left parenthesis, 2, n, minus, 1, right parenthesis.
- Trissomia é quando um organismo tem uma terceira cópia de um cromossomo que deveria estar presente em duas cópias (2n+1)left parenthesis, 2, n, plus, 1, right parenthesis.
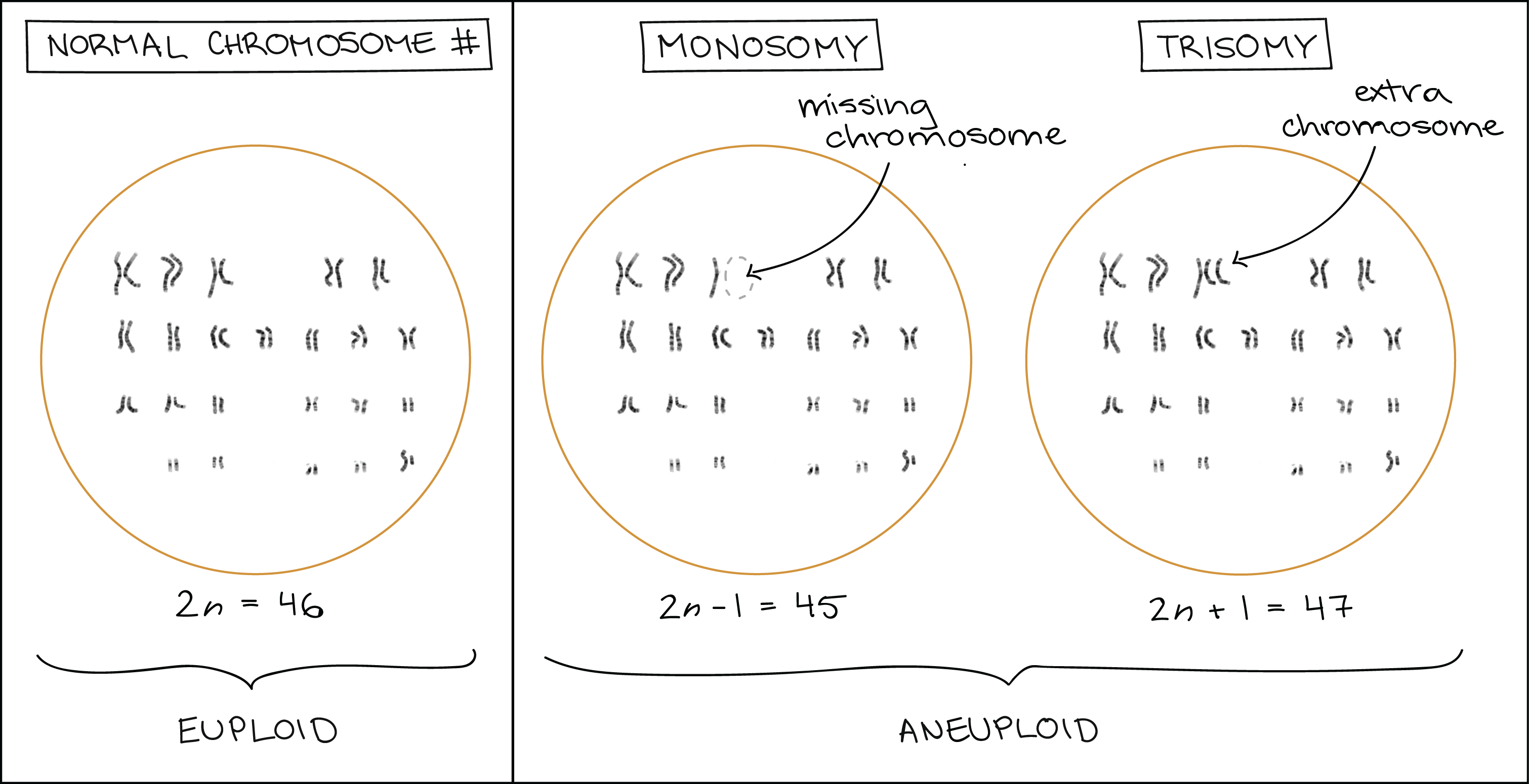 Diagrama ilustrando euploidia e aneuploidia.Célula euploide: uma célula humana com número normal de cromossomos, 2n = 46. Os cromossomos estão organizados em 23 pares.Célula aneuploide, exemplo 1: monossomia. Uma célula humana com um cromossomo a menos, neste caso, o cromossomo 3. Todos os outros cromossomos ainda estão organizados em pares de dois, mas há apenas uma cópia do cromossomo 3. O número de cromossomos desta célula é 2n-1 = 45.Célula aneuploide, exemplo 2: trissomia. Uma célula humana com um cromossomo extra, neste caso uma cópia extra do cromossomo 3. Todos os outros cromossomos continuam organizados em pares de dois, mas há três cópias do cromossomo. O número de cromossomos desta célula é 2n+1 = 47.Imagem modificada de "NHGRI human male karyotype," pelo National Human Genome Research Institute (domínio público).Aneuploidia também inclui casos onde uma célula tem um número maior de cromossomos extras ou ausentes, como em (2n−2),(2n+3)left parenthesis, 2, n, minus, 2, right parenthesis, comma, left parenthesis, 2, n, plus, 3, right parenthesis, etc. No entanto, se houver um conjunto completo de cromossomos extras ou ausentes (p.e., 3n3, n), isto não é formalmente considerado aneuploidia, mesmo que ainda possa ser ruim para a célula ou para o organismo. Os organismos com mais de um conjunto completo de cromossomos são chamados de poliploides.
Diagrama ilustrando euploidia e aneuploidia.Célula euploide: uma célula humana com número normal de cromossomos, 2n = 46. Os cromossomos estão organizados em 23 pares.Célula aneuploide, exemplo 1: monossomia. Uma célula humana com um cromossomo a menos, neste caso, o cromossomo 3. Todos os outros cromossomos ainda estão organizados em pares de dois, mas há apenas uma cópia do cromossomo 3. O número de cromossomos desta célula é 2n-1 = 45.Célula aneuploide, exemplo 2: trissomia. Uma célula humana com um cromossomo extra, neste caso uma cópia extra do cromossomo 3. Todos os outros cromossomos continuam organizados em pares de dois, mas há três cópias do cromossomo. O número de cromossomos desta célula é 2n+1 = 47.Imagem modificada de "NHGRI human male karyotype," pelo National Human Genome Research Institute (domínio público).Aneuploidia também inclui casos onde uma célula tem um número maior de cromossomos extras ou ausentes, como em (2n−2),(2n+3)left parenthesis, 2, n, minus, 2, right parenthesis, comma, left parenthesis, 2, n, plus, 3, right parenthesis, etc. No entanto, se houver um conjunto completo de cromossomos extras ou ausentes (p.e., 3n3, n), isto não é formalmente considerado aneuploidia, mesmo que ainda possa ser ruim para a célula ou para o organismo. Os organismos com mais de um conjunto completo de cromossomos são chamados de poliploides.A não disjunção dos cromossomos
Anomalias nos números de cromossomos são causadas pela não disjunção, que ocorre quando os pares de cromossomos homólogos ou cromátides irmãs não conseguem separar-se durante a meiose I ou II (ou durante a mitose).Meiose I. O diagrama abaixo mostra como a não disjunção pode ocorrer durante a meiose I se os homólogos não se separarem e como isto pode levar à produção de gametas aneuploides (óvulos ou esperma):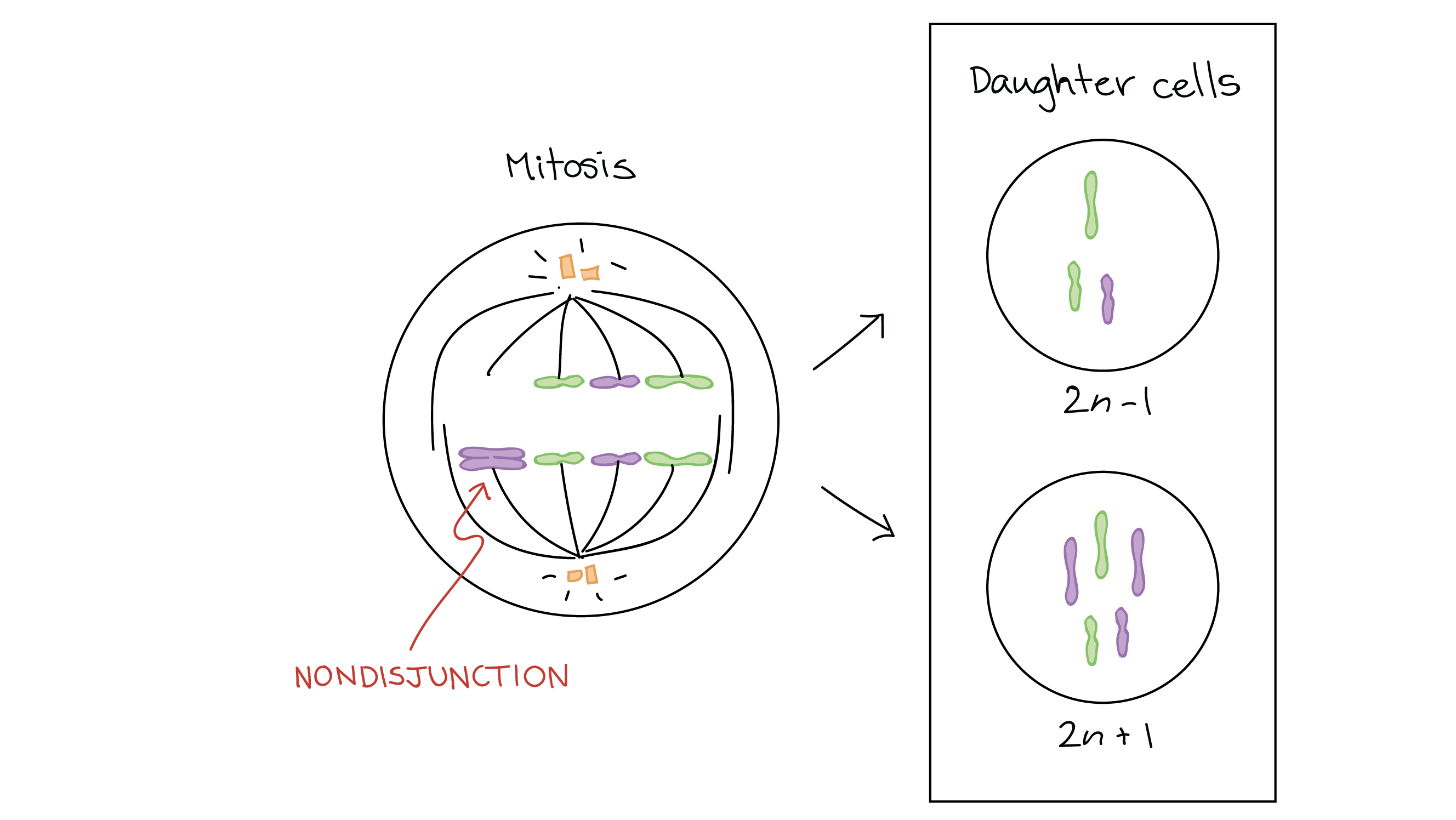 Imagem representando a não disjunção na meiose I. Um par de cromossomos homólogos não se separa durante a meiose I, levando a duas células anormais como produtos da meiose I: uma célula com um cromossomo extra e outra com um cromossomo faltando. Na meiose II, as cromátides irmãs dos cromossomos se separam normalmente. Isto leva à produção de dois gametas com um cromossomo extra (gametas n+1) e dois gametas com um cromossomo ausente (gametas n-1).Meiose II. A não disjunção também pode ocorrer na meiose II, quando as cromátides irmãs (ao invés dos cromossomos homólogos) não se separam. Novamente, alguns gametas irão conter cromossomos a mais ou a menos:
Imagem representando a não disjunção na meiose I. Um par de cromossomos homólogos não se separa durante a meiose I, levando a duas células anormais como produtos da meiose I: uma célula com um cromossomo extra e outra com um cromossomo faltando. Na meiose II, as cromátides irmãs dos cromossomos se separam normalmente. Isto leva à produção de dois gametas com um cromossomo extra (gametas n+1) e dois gametas com um cromossomo ausente (gametas n-1).Meiose II. A não disjunção também pode ocorrer na meiose II, quando as cromátides irmãs (ao invés dos cromossomos homólogos) não se separam. Novamente, alguns gametas irão conter cromossomos a mais ou a menos: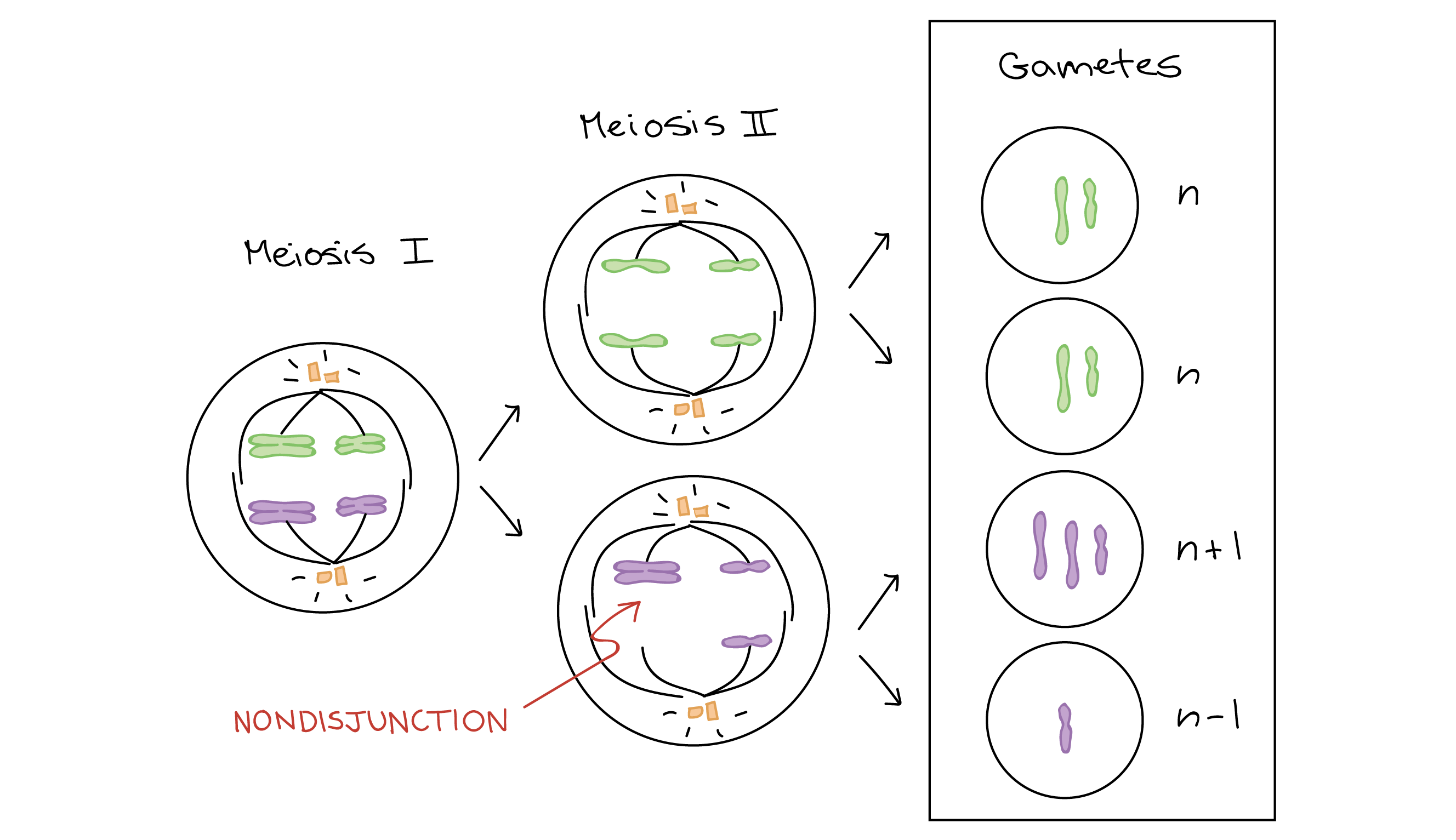 Diagrama representando não disjunção na meiose II. Os cromossomos homólogos se separam normalmente durante a meiose I. No entanto, as cromátides irmãs de um cromossomo não se separam durante a meiose II e, em vez disso, migram para o mesmo pólo da célula e são segregadas para o mesmo gameta. Neste caso, os produtos da meiose são dois gametas normais, euploides (n), um gameta com um cromossomo extra (n+1) e um gameta com um cromossomo ausente (n-1).Mitose. A não disjunção também pode acontecer durante a mitose. Em seres humanos, alterações cromossômicas devido à não disjunção durante a mitose em células do corpo não serão passadas para os filhos (pois estas células não produzem espermatozoides e óvulos). Mas a não disjunção mitótica pode causar outros problemas: células cancerosas geralmente têm número anormal de cromossomos2squared.Quando um espermatozoide ou óvulo aneuploide combina-se com um óvulo ou espermatozoide normal, durante a fertilização, produz um zigoto que também é aneuploide. Por exemplo, se um espermatozoide com um cromossomo extra (n+1n, plus, 1) combinar-se com um óvulo normal (nn), o zigoto, ou embrião unicelular, resultante terá um número de cromossomos de 2n+12, n, plus, 1.
Diagrama representando não disjunção na meiose II. Os cromossomos homólogos se separam normalmente durante a meiose I. No entanto, as cromátides irmãs de um cromossomo não se separam durante a meiose II e, em vez disso, migram para o mesmo pólo da célula e são segregadas para o mesmo gameta. Neste caso, os produtos da meiose são dois gametas normais, euploides (n), um gameta com um cromossomo extra (n+1) e um gameta com um cromossomo ausente (n-1).Mitose. A não disjunção também pode acontecer durante a mitose. Em seres humanos, alterações cromossômicas devido à não disjunção durante a mitose em células do corpo não serão passadas para os filhos (pois estas células não produzem espermatozoides e óvulos). Mas a não disjunção mitótica pode causar outros problemas: células cancerosas geralmente têm número anormal de cromossomos2squared.Quando um espermatozoide ou óvulo aneuploide combina-se com um óvulo ou espermatozoide normal, durante a fertilização, produz um zigoto que também é aneuploide. Por exemplo, se um espermatozoide com um cromossomo extra (n+1n, plus, 1) combinar-se com um óvulo normal (nn), o zigoto, ou embrião unicelular, resultante terá um número de cromossomos de 2n+12, n, plus, 1.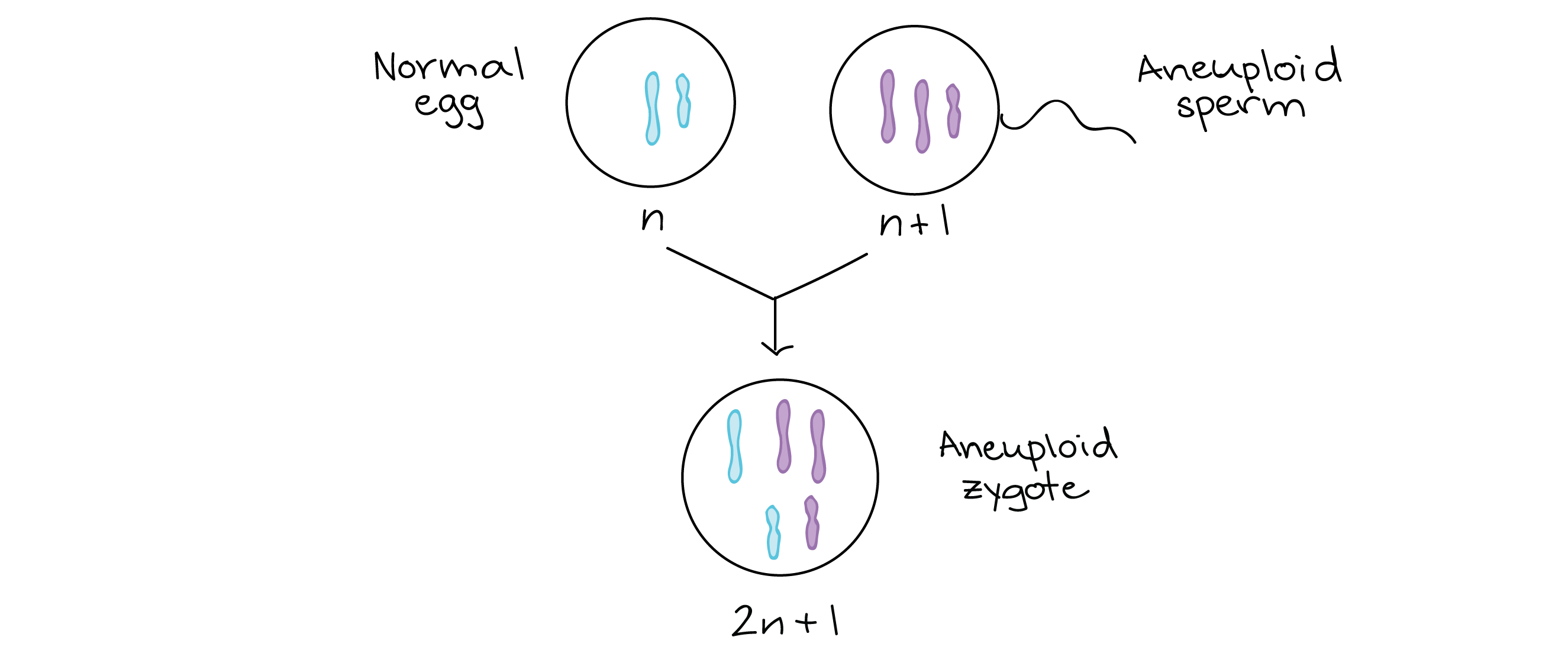 Diagrama de um evento de fertilização onde um óvulo normal (n) se combina com um espermatozoide aneuploide (n + 1). O zigoto formado pela fertilização é aneuploide (2n + 1).
Diagrama de um evento de fertilização onde um óvulo normal (n) se combina com um espermatozoide aneuploide (n + 1). O zigoto formado pela fertilização é aneuploide (2n + 1).Distúrbios genéticos causados por aneuploidia
Embriões humanos nos quais falta uma cópia de qualquer autossomo (cromossomo não sexual) não se desenvolvem até o nascimento. Em outras palavras, monossomias autossômicas humanas são sempre letais. Isto porque os embriões têm uma "dose" muito baixa de proteínas e de outros produtos que são codificados pelos genes do cromossomo que falta3cubed.A maioria das trissomias autossômicas também impedem que um embrião se desenvolva até o nascimento. Mas uma cópia extra de algum dos cromossomos menores (13, 15, 18, 21, ou 22) pode permitir que o indivíduo afetado sobreviva por um curto espaço de tempo após o nascimento, ou em alguns casos, por muitos anos. Quando um cromossomo extra está presente, ele pode causar problemas no desenvolvimento devido ao desequilíbrio entre os produtos dos genes do cromossomo duplicado e os produtos dos genes de outros cromossomos3cubed.A trissomia mais comum entre os embriões que sobrevivem ao nascimento é a síndrome de Down, ou trissomia 21. Pessoas com este distúrbio hereditário têm estatura baixa e dedos curtos, distinções faciais que incluem um crânio largo, língua grande e atrasos no desenvolvimento. Aqui está um cariótipo, ou imagem dos cromossomos, de uma pessoa com síndrome de Down, mostrando as características três cópias do cromossomo 21: Cariótipo de um ser humano do sexo masculino com síndrome de Down. A maioria dos pares autossômicos e o par de cromossomos sexuais X-Y são normais. Contudo, o cromossomo 21 está presente em três cópias.Crédito da imagem: "Trissomia do 21 - síndrome de Down," do U.S. Department of Energy Human Genome Program (domínio público).Cerca de 11 a cada 800800 recém-nascidos nasce com síndrome de Down 4start superscript, 4, end superscript. Mas, a probabilidade de que a gravidez resulte em um embrião com a síndrome de Down aumenta com a idade da mulher, particularmente acima de 4040 anos5,6start superscript, 5, comma, 6, end superscript. Isto provavelmente se deve à maior frequência de não disjunções no desenvolvimento dos óvulos das mulheres mais velhas.SAIBA MAIS:
Cariótipo de um ser humano do sexo masculino com síndrome de Down. A maioria dos pares autossômicos e o par de cromossomos sexuais X-Y são normais. Contudo, o cromossomo 21 está presente em três cópias.Crédito da imagem: "Trissomia do 21 - síndrome de Down," do U.S. Department of Energy Human Genome Program (domínio público).Cerca de 11 a cada 800800 recém-nascidos nasce com síndrome de Down 4start superscript, 4, end superscript. Mas, a probabilidade de que a gravidez resulte em um embrião com a síndrome de Down aumenta com a idade da mulher, particularmente acima de 4040 anos5,6start superscript, 5, comma, 6, end superscript. Isto provavelmente se deve à maior frequência de não disjunções no desenvolvimento dos óvulos das mulheres mais velhas.SAIBA MAIS:- Em mulheres, os precursores dos óvulos (chamados de oócitos primários) se formam muito precocemente, quando a mulher é ainda um embrião. Apesar de os oócitos primários começarem a meiose nesta fase inicial, eles não a completam. Em vez disso, eles fazem uma pausa na prófase I, onde permanecem por muitos anos.
- Quando o corpo da mulher se prepara para a ovulação durante o ciclo menstrual, um dos oócitos primários completará a meiose para produzir um óvulo. Os oócitos primários que ficaram parados na prófase I por um longo período (por exemplo, em uma mulher de 4040 anos), têm um risco maior de ter uma segregação cromossômica anormal (não disjunção), quando eles reiniciarem a meiose, que os oócitos primários que ficaram parados na prófase I por um período de tempo menor (por exemplo, numa mulher de 2020 anos)7start superscript, 7, end superscript.Os distúrbios genéticos humanos também podem ser causados por aneuploidias envolvendo os cromossomos sexuais. Estas aneuploidias são melhor toleradas que as autossômicas porque as células humanas têm capacidade para desligar os cromossomos X extras, em um processo chamado de inativação do X. Você pode aprender mais sobre isto no artigo sobre Inativação do cromossomo X(Abre em uma nova janela)(Abre em uma nova janela)(Abre em uma nova janela).
Rearranjos cromossômicos
Em outra classe de mutações de larga escala, grandes pedaços de cromossomos (mas não cromossomos inteiros) são afetados. Tais mudanças são chamadas rearranjos cromossômicos. Elas incluem:- Uma duplicação, onde parte de um cromossomo é copiada.
- Uma deleção, onde parte de um cromossomo é removida.
- Uma inversão, onde uma região cromossômica é invertida apontando assim, para a direção oposta.
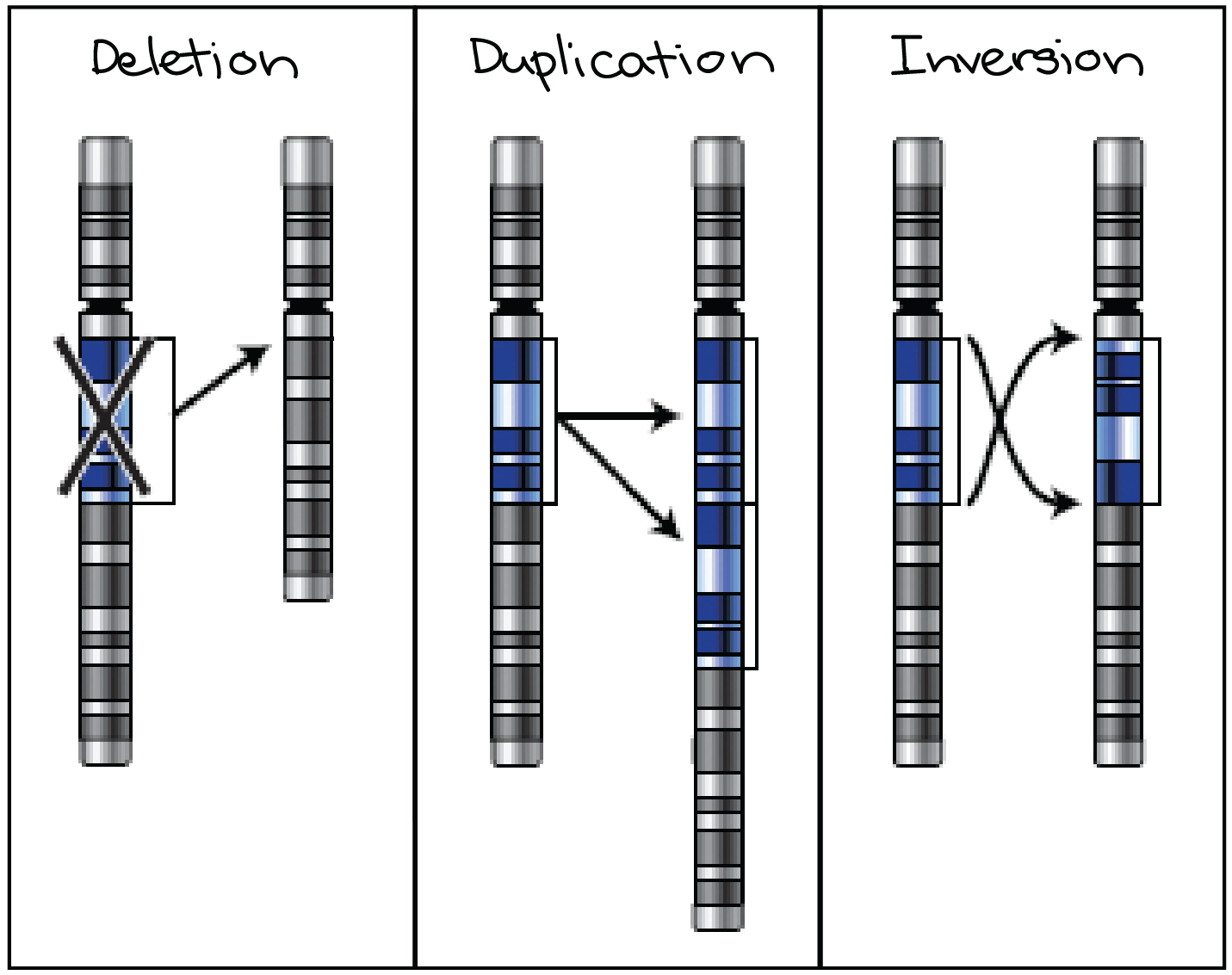 Diagrama representando esquematicamente a deleção, a duplicação e a inversão.Deleção: uma região do cromossomo original é removida, levando a um cromossomo mais curto em que falta uma seção.Duplicação: uma região do cromossomo original é duplicada, levando a um cromossomo mais longo com uma cópia a mais de uma seção em particular.Inversão: uma região do cromossomo original se separa do restante do cromossomo e é recolocada em seu lugar original, mas na orientação oposta.Imagem modificada de "Chromosomenmutation," por Deitzel66, modificada de NIH Talking Glossary of Genetics (domínio público).
Diagrama representando esquematicamente a deleção, a duplicação e a inversão.Deleção: uma região do cromossomo original é removida, levando a um cromossomo mais curto em que falta uma seção.Duplicação: uma região do cromossomo original é duplicada, levando a um cromossomo mais longo com uma cópia a mais de uma seção em particular.Inversão: uma região do cromossomo original se separa do restante do cromossomo e é recolocada em seu lugar original, mas na orientação oposta.Imagem modificada de "Chromosomenmutation," por Deitzel66, modificada de NIH Talking Glossary of Genetics (domínio público). - Uma translocação, onde uma parte de um cromossomo fica aderida a outro cromossomo. Uma translocação recíproca envolve a troca de segmentos entre dois cromossomos; uma translocação não recíproca significa que um segmento de um cromossomo se move para o outro.
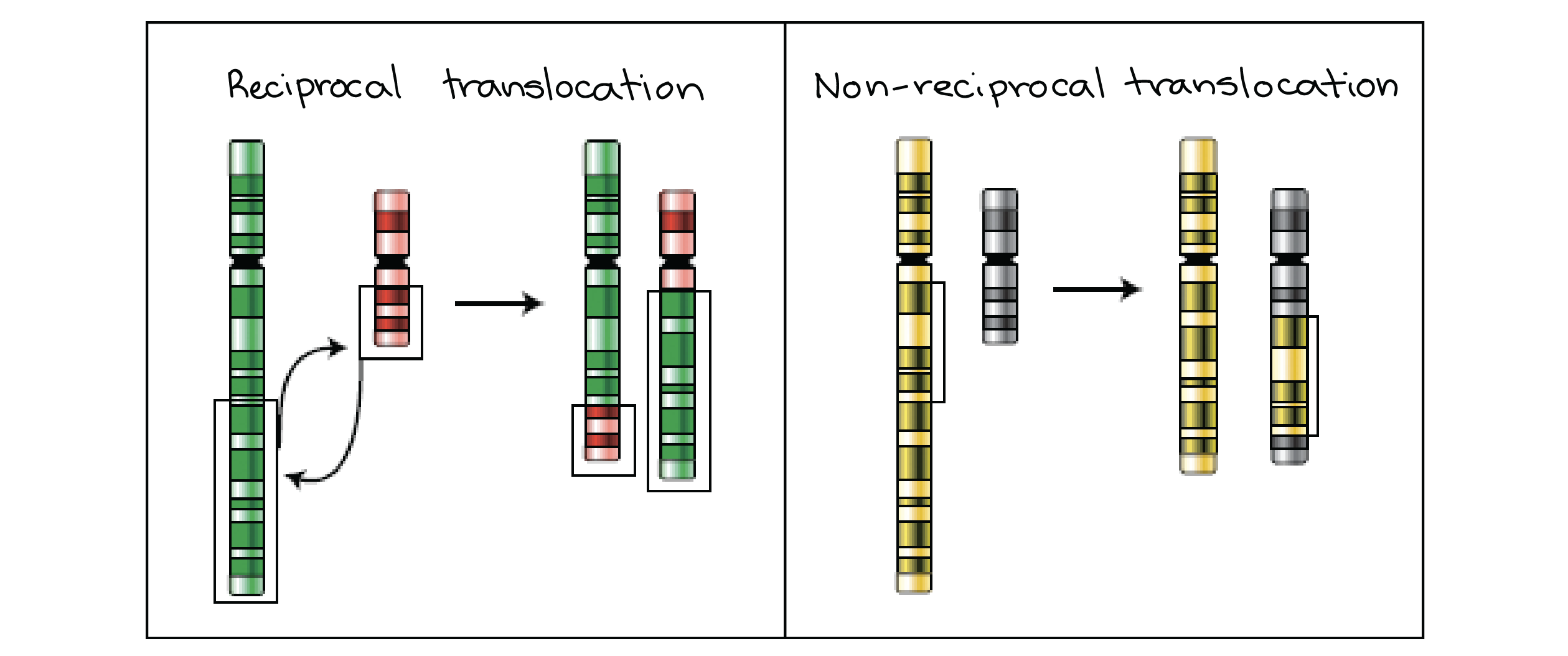 Diagrama da representação esquemática das translocações recíprocas e não recíprocas.Translocação recíproca: troca de fragmentos entre dois cromossomos não homólogos. Nenhum material genético se perde, mas os cromossomos resultantes são híbridos, cada um contendo segmentos encontrados normalmente em um cromossomo diferente.Translocação não recíproca: um fragmento é removido de um cromossomo doador e inserido em um cromossomo receptor. O cromossomo doador perde uma região, enquanto que o cromossomo receptor ganha uma região normalmente não encontrada nesse cromossomo.Imagem modificada de "Chromosomenmutation," por Deitzel66, modificada de NIH Talking Glossary of Genetics (domínio público).
Diagrama da representação esquemática das translocações recíprocas e não recíprocas.Translocação recíproca: troca de fragmentos entre dois cromossomos não homólogos. Nenhum material genético se perde, mas os cromossomos resultantes são híbridos, cada um contendo segmentos encontrados normalmente em um cromossomo diferente.Translocação não recíproca: um fragmento é removido de um cromossomo doador e inserido em um cromossomo receptor. O cromossomo doador perde uma região, enquanto que o cromossomo receptor ganha uma região normalmente não encontrada nesse cromossomo.Imagem modificada de "Chromosomenmutation," por Deitzel66, modificada de NIH Talking Glossary of Genetics (domínio público).
Em alguns casos, um rearranjo cromossômico causa sintomas semelhantes aos da perda ou ganho de um cromossomo inteiro. Por exemplo, a síndrome de Down é geralmente causada por uma terceira cópia do cromossomo 21, mas também pode ocorrer quando um segmento grande do cromossomo 21 se move para outro cromossomo (e é passado para a prole, junto com um cromossomo 21 normal)4start superscript, 4, end superscript. Em outros casos, os rearranjos causam distúrbios específicos, que não estão associados com a aneuploidia.Créditos:
Este artigo é uma versão modificada derivada de "Chromosomal basis of inherited disorders," by OpenStax College, Biology, CC BY 4.0. Baixe o artigo original gratuitamente em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@10.53.Este artigo está autorizado sob licença CC BY-NC-SA 4.0.Referências:
- Madhusoodanan, J. (2014, July 8). Human gene set shrinks again. In The scientist. Disponível em: http://www.the-scientist.com/?articles.view/articleNo/40441/title/Human-Gene-Set-Shrinks-Again/.
- Hanahan, D. and Weinberg, R. A. (2011). Hallmarks of cancer: the next generation. Cell, 144(5), 646-674. http://dx.doi.org/10.1016/j.cell.2011.02.013.
- Torres, E. M., Williams, B. R., and Amon, A. (2008). Aneuploidy: Cells losing their balance. Genetics, 179(2), 737-746. http://dx.doi.org/10.1534/genetics.108.090878. Disponível em http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2429870/.
- Down syndrome. (2012). In Genetics home reference. Disponível em http://ghr.nlm.nih.gov/condition/down-syndrome.
- Leshin, L. (n.d.) Frequency of Down syndrome per maternal age. In Down syndrome: Health issues. Disponível em http://www.ds-health.com/risk.htm.
- National Down Syndrome Society. (2012). What is Down syndrome? Disponível em http://www.ndss.org/Down-Syndrome/What-Is-Down-Syndrome/.
- Rodriguez, Monica. (2007, October 4). "Extra or missing chromosomes." In Stanford at the Tech: Understanding genetics. Acesso em 27 de julho, 2016. Disponível em http://genetics.thetech.org/ask/ask234.
Referências:
16p11.2 deletion syndrome. (2014). In Genetics home reference. Disponível em: http://ghr.nlm.nih.gov/condition/16p112-deletion-syndrome.Champaigne, N. (2008). Abnormalities of chromosomes number (aneuploidy). In Medical genetics and dysmorphology. Disponível em http://www.utmb.edu/pedi_ed/CORE/MedicalGenetics/page_11.htm.Cri-du-chat syndrome. (2014). In Genetics home reference. Disponível em: http://ghr.nlm.nih.gov/condition/cri-du-chat-syndrome.Down syndrome. (2015, May 28). In Online Mendelian inheritance in man. Disponível em: http://omim.org/entry/190685.Griffiths, A. J. F., Gelbart, W. M., Miller, J. H., and Lewontin, R. C. (1999). Chromosomal rearrangements. In Modern genetic analysis. New York, NY: W. H. Freeman. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK21367/.Hanahan, D. and Weinberg, R. A. (2011). Hallmarks of cancer: the next generation. Cell, 144(5), 646-674. http://dx.doi.org/10.1016/j.cell.2011.02.013.Hill, M.A. (2015). Oocyte development. In Embryology. Acesso em 17 de dezembro, 2015. Disponível em: https://embryology.med.unsw.edu.au/embryology/index.php/Oocyte_Development.Moran, L. A. (2007, March 24). Summary of genes on human chromosomes [Web log post]. Disponível em: http://sandwalk.blogspot.com/2007/03/summary-of-genes-on-human-chromosomes.html.Mutation. (2015, December 15). Acesso em 17 de dezembro, 2015. Disponível em: Wikipedia: https://en.wikipedia.org/wiki/Mutation.O'Neil, Dennis. (2013). Sex chromosome abnormalities. Em Human chromosomal abnormalities. Disponível em: http://anthro.palomar.edu/abnormal/abnormal_5.htm.Oogenesis. (2015, December 13). Acesso em 17 de dezembro, 2015. Disponível em: Wikipedia: https://en.wikipedia.org/wiki/Oogenes.Oogenesis. (n.d.). In Embryogenesis. Disponível em: http://www.embryology.ch/anglais/cgametogen/oogenese01.html.Philadelphia chromosome. (2013). In Genetics home reference. Disponível em: http://ghr.nlm.nih.gov/condition/pdgfrb-associated-chronic-eosinophilic-leukemia.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Alterations of chromosome number or structure cause some genetic disorders. In Campbell biology (10th ed., pp. 304-307). San Francisco, CA: Pearson.Rodriguez, Monica. (2007, October 4). "Extra or missing chromosomes." In Stanford at the Tech: Understanding genetics. Acesso em 27 de julho, 2016. Disponível em http://genetics.thetech.org/ask/ask234.Szauter, P. (2005, July 18). Why does rate of Down syndrome increase with maternal age? [answer]. In MadSci network: Genetics. Disponível em: http://www.madsci.org/posts/archives/2005-07/1121714807.Ge.r.html.Trisomy. (2015, November 26). Acesso em 17 de dezembro, 2015. Disponível em: Wikipedia: https://en.wikipedia.org/wiki/Trisomy.Herança do DNA mitocondrial e cloroplástico
- DNA mitocondrial e cloroplástico e porque sua herança não segue padrões mendelianos.
Introdução
Se pedissem a você para nomear a organela que contém DNA, o que você diria? Se você respondesse o núcleo, você definitivamente marcaria pontos, mas o núcleo não é a única fonte de DNA na maioria das células.Ao invés disso, o DNA também é encontrado nas mitocôndrias presentes na maioria das células animais e vegetais, assim como nos cloroplastos das células vegetais. Aqui, exploraremos como o DNA mitocondrial e dos cloroplastos são herdados.DNA mitocondrial e cloroplástico
As moléculas de DNA encontradas nas mitocôndrias e cloroplastos são pequenas e circulares, muito parecidas com o DNA de uma bactéria típica. Geralmente existem muitas cópias de DNA em uma única mitocôndria ou cloroplasto.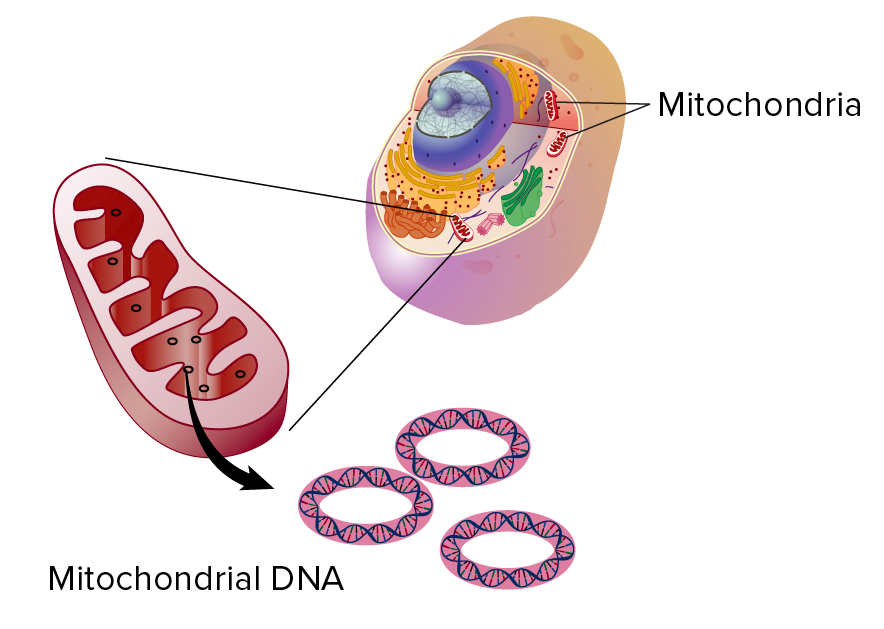 Diagrama de uma célula eucariótica, mostrando a localização da mitocôndria (flutuando no citosol; fora e separada do núcleo) e a ampliação na mitocôndria para mostrar as moléculas de DNA circular em seu interior.Imagem modificada de "DNA Mitocondrial," NIH Talking Glossary of Genetic Terms, NHGRI (domínio público).As semelhanças entre o DNA das mitocôndrias e cloroplastos e o DNA das bactérias são uma importante linha de evidência apoiando a teoria endossimbiótica, que sugere que as mitocôndrias e cloroplastos eram originalmente células procariontes de vida livre.
Diagrama de uma célula eucariótica, mostrando a localização da mitocôndria (flutuando no citosol; fora e separada do núcleo) e a ampliação na mitocôndria para mostrar as moléculas de DNA circular em seu interior.Imagem modificada de "DNA Mitocondrial," NIH Talking Glossary of Genetic Terms, NHGRI (domínio público).As semelhanças entre o DNA das mitocôndrias e cloroplastos e o DNA das bactérias são uma importante linha de evidência apoiando a teoria endossimbiótica, que sugere que as mitocôndrias e cloroplastos eram originalmente células procariontes de vida livre.Como é herdado o DNA não nuclear?
Estes são alguns modos em que o DNA dos cloroplastos e das mitocôndrias diferem do DNA encontrado no núcleo:- Elevado número de cópias. Uma mitocôndria ou cloroplasto apresenta múltiplas cópias do seu DNA, e uma célula típica apresenta muitas mitocôndrias (no caso de uma célula de planta, cloroplastos). Como resultado, as células geralmente apresentam muitas cópias – com frequência, milhares – de DNA mitocondrial e cloroplástico.
- SAIBA MAIS:
- Não necessariamente. Há boas evidências de que as mitocôndrias individuais podem se fundir e trocar material genético umas com as outras, significando que nem todas as cópias de DNA em uma mitocôndria são necessariamente idênticas1start superscript, 1, end superscript. Nas plantas, a fusão de cloroplastos parece menos comum, assim os cloroplastos individuais tendem a ser geneticamente homogêneos (apesar de que vários cloroplastos em uma célula podem ainda ser diferentes uns dos outros)2squared. Naturalmente, também é possível que as cópias de DNA em uma organela geneticamente homogênea tornem-se não idênticas se uma mutação aleatória ocorrer em uma delas.
- Segregação aleatória. As mitocôndrias e os cloroplastos (e os genes que eles carregam) tendem a ser distribuídos aleatoriamente nas células-filhas durante a mitose e meiose. Quando ocorre a citocinese, as organelas que estão em lados opostos do sulco de clivagem terminam em células-filhas diferentes3cubed.
- Herança monoparental. O DNA não nuclear é muitas vezes herdado uniparentalmente, significando que a prole obtém o DNA apenas do progenitor ou da progenitora, não de ambos4start superscript, 4, end superscript. Em humanos, por exemplo, as crianças obtêm DNA mitocondrial de sua mãe (mas não do pai).
Herança cloroplástica: as primeiras experiências
No começo do século XX, Carl Correns, um botânico alemão, realizou uma série de experiências genéticas utilizando a planta maravilha (Mirabilis jalapa) ('four o'clock plants', em inglês). Hoje sabemos que seu trabalho demonstrou como o DNA do cloroplasto é passado de célula para célula, e dos pais para os descendentes - embora o próprio Correns não soubesse disto naquela época5start superscript, 5, end superscript!Experimentos de Correns
As plantas de Mirabilis com as quais Correns trabalhou resultaram em três tipos: verde puro, branco puro ou variegada (malhada de verde e branco). Ramos verdes e brancos poderiam aparecer em plantas variegadas, mas ramos variegados não apareceram em plantas verdes ou brancas6start superscript, 6, end superscript.Correns ficou curioso a respeito dessa característica de coloração e realizou uma série de cruzamentos entre plantas de cores diferentes. Ele descobriu que6start superscript, 6, end superscript:- A coloração do ramo do óvulo doador (progenitora) ditou a coloração do descendente.
- Os ramos maternos puramente brancos ou verdes somente produziram descendentes puramente brancos e verdes, respectivamente.
- Ramos variegados da progenitora poderiam produzir todos os três tipos de descendência, mas não em qualquer proporção previsível.
Correns especulou que algum fator no citoplasma do óvulo deve determinar a cor dos descendentes. Foi, na verdade, um outro botânico alemão, Erwin Baur, que sugeriu que os cloroplastos no citoplasma podem carregar fatores hereditários (genes)5start superscript, 5, end superscript.Baur pensou que, em plantas variegadas, alguns dos cloroplastos devem ter mutações que os torna incapazes de ficarem verdes (produzir o pigmento). Hoje, sabemos que esta hipótese estava perfeitamente correta!Explicando os resultados de Correns
Como é que a ideia de herança do cloroplasto pode deixar variegadas as plantas variegadas? Vamos seguir um zigoto (embrião unicelular) com a mistura de cloroplastos herdada do óvulo. Alguns dos cloroplastos são verdes, enquanto outros são brancos. Como o zigoto sofre muitos ciclos de mitose para formar um embrião e, em seguida, uma planta, os cloroplastos também se dividem e são distribuídos aleatoriamente em células-filhas em cada divisão.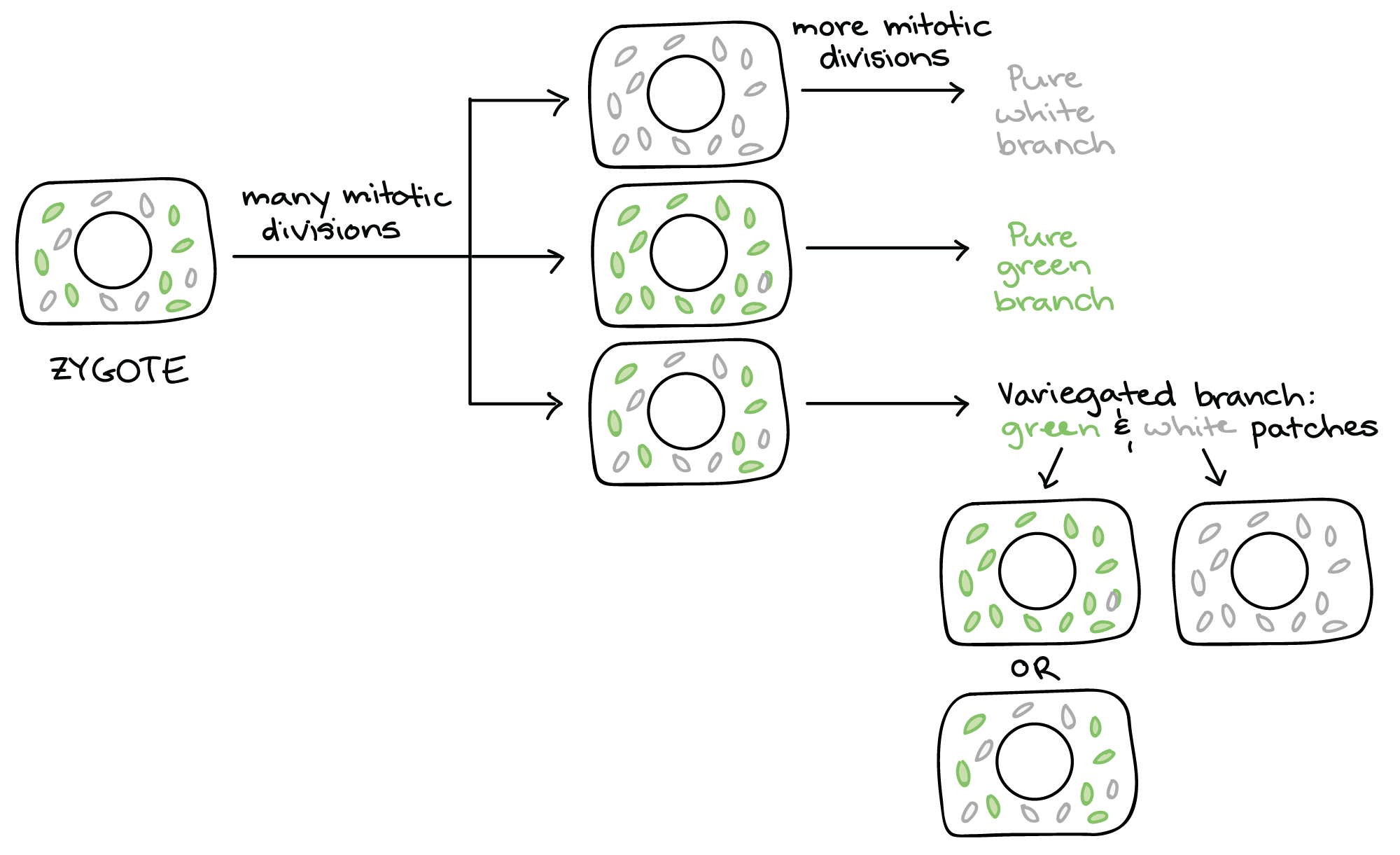 Imagem mostrando segregação citoplasmática de cloroplastos em uma planta originária de um zigoto com uma mistura de cloroplastos brancos (não funcionais, mutantes) e cloroplastos verdes (funcionais, normais). Depois de muitas divisões mitóticas, nas quais os cloroplastos se replicam e são distribuídos aleatoriamente, algumas células terão apenas cloroplastos verdes, outras terão apenas cloroplastos brancos e outras continuarão a ter uma mistura. As células com apenas cloroplastos brancos darão origem a ramos puramente brancos e as células com apenas cloroplastos verdes darão origem a ramos puramente verdes. As células com uma mistura de cloroplastos darão origem a ramos mesclados, nos quais a contínua segregação aleatória dos cloroplastos produzirá setores brancos (descendência das células com apenas cloroplastos brancos) e setores verdes (descendência das células com cloroplastos mistos ou apenas verde). As células verdes que contêm uma mistura de cloroplastos continuarão a produzir ocasionalmente setores puramente brancos e puramente verdes conforme elas se dividem.Baseado em diagrama similar encontrado em Griffiths et al.7start superscript, 7, end superscript.Durante as muitas divisões celulares, algumas células terminarão com um simples conjunto de cloroplastos normais (ganhando manchas verdes). Outros receberão um conjunto simples de cloroplastos não funcionais (ganhando manchas brancas). Outros, ainda, terão uma mistura de cloroplastos normais e não funcionais, produzindo manchas verdes que podem dar origem a setores de verde puro ou branco puro7start superscript, 7, end superscript.E quanto ao padrão materno de herança? As plantas produzem células germinativas tardiamente no seu desenvolvimento, convertendo as células na ponta de um ramo em células produtoras de gametas. Um ramo verde puro produzirá óvulos com cloroplastos verdes que darão origem a uma prole de verde puro. Da mesma forma, um ramo branco puro produzirá óvulos que contenham apenas cloroplastos brancos e darão origem a uma prole de branco puro.Se um ramo for variegado, ele possui uma mistura de células, algumas somente com cloroplastos funcionais, outras somente com cloroplastos não funcionais e outras com uma mistura de cloroplastos. Todos estes três tipos celulares podem dar origem a óvulos, originando uma prole verde, uma prole branca e uma prole variegada em proporções não previsíveis6,7start superscript, 6, comma, 7, end superscript.
Imagem mostrando segregação citoplasmática de cloroplastos em uma planta originária de um zigoto com uma mistura de cloroplastos brancos (não funcionais, mutantes) e cloroplastos verdes (funcionais, normais). Depois de muitas divisões mitóticas, nas quais os cloroplastos se replicam e são distribuídos aleatoriamente, algumas células terão apenas cloroplastos verdes, outras terão apenas cloroplastos brancos e outras continuarão a ter uma mistura. As células com apenas cloroplastos brancos darão origem a ramos puramente brancos e as células com apenas cloroplastos verdes darão origem a ramos puramente verdes. As células com uma mistura de cloroplastos darão origem a ramos mesclados, nos quais a contínua segregação aleatória dos cloroplastos produzirá setores brancos (descendência das células com apenas cloroplastos brancos) e setores verdes (descendência das células com cloroplastos mistos ou apenas verde). As células verdes que contêm uma mistura de cloroplastos continuarão a produzir ocasionalmente setores puramente brancos e puramente verdes conforme elas se dividem.Baseado em diagrama similar encontrado em Griffiths et al.7start superscript, 7, end superscript.Durante as muitas divisões celulares, algumas células terminarão com um simples conjunto de cloroplastos normais (ganhando manchas verdes). Outros receberão um conjunto simples de cloroplastos não funcionais (ganhando manchas brancas). Outros, ainda, terão uma mistura de cloroplastos normais e não funcionais, produzindo manchas verdes que podem dar origem a setores de verde puro ou branco puro7start superscript, 7, end superscript.E quanto ao padrão materno de herança? As plantas produzem células germinativas tardiamente no seu desenvolvimento, convertendo as células na ponta de um ramo em células produtoras de gametas. Um ramo verde puro produzirá óvulos com cloroplastos verdes que darão origem a uma prole de verde puro. Da mesma forma, um ramo branco puro produzirá óvulos que contenham apenas cloroplastos brancos e darão origem a uma prole de branco puro.Se um ramo for variegado, ele possui uma mistura de células, algumas somente com cloroplastos funcionais, outras somente com cloroplastos não funcionais e outras com uma mistura de cloroplastos. Todos estes três tipos celulares podem dar origem a óvulos, originando uma prole verde, uma prole branca e uma prole variegada em proporções não previsíveis6,7start superscript, 6, comma, 7, end superscript.- Correns especulou que algum fator no citoplasma do óvulo deve determinar a cor dos descendentes. Foi, na verdade, um outro botânico alemão, Erwin Baur, que sugeriu que os cloroplastos no citoplasma podem carregar fatores hereditários (genes)5start superscript, 5, end superscript.Baur pensou que, em plantas variegadas, alguns dos cloroplastos devem ter mutações que os torna incapazes de ficarem verdes (produzir o pigmento). Hoje, sabemos que esta hipótese estava perfeitamente correta!
- Herança mitocondrial
- As mitocôndrias, como os cloroplastos, tendem a ser herdados somente de um dos genitores (ou, pelo menos, serem desigualmente herdados dos dois genitores)4start superscript, 4, end superscript. No caso dos seres humanos, é a mãe que contribui com as mitocôndrias para o zigoto, ou embrião unicelular, através do citoplasma do óvulo. Os espermatozoides contêm mitocôndrias, mas eles normalmente não são herdados pelo zigoto. Houve o relato de um caso de herança paterna de mitocôndrias em um ser humano, mas isto é extremamente raro8start superscript, 8, end superscript.
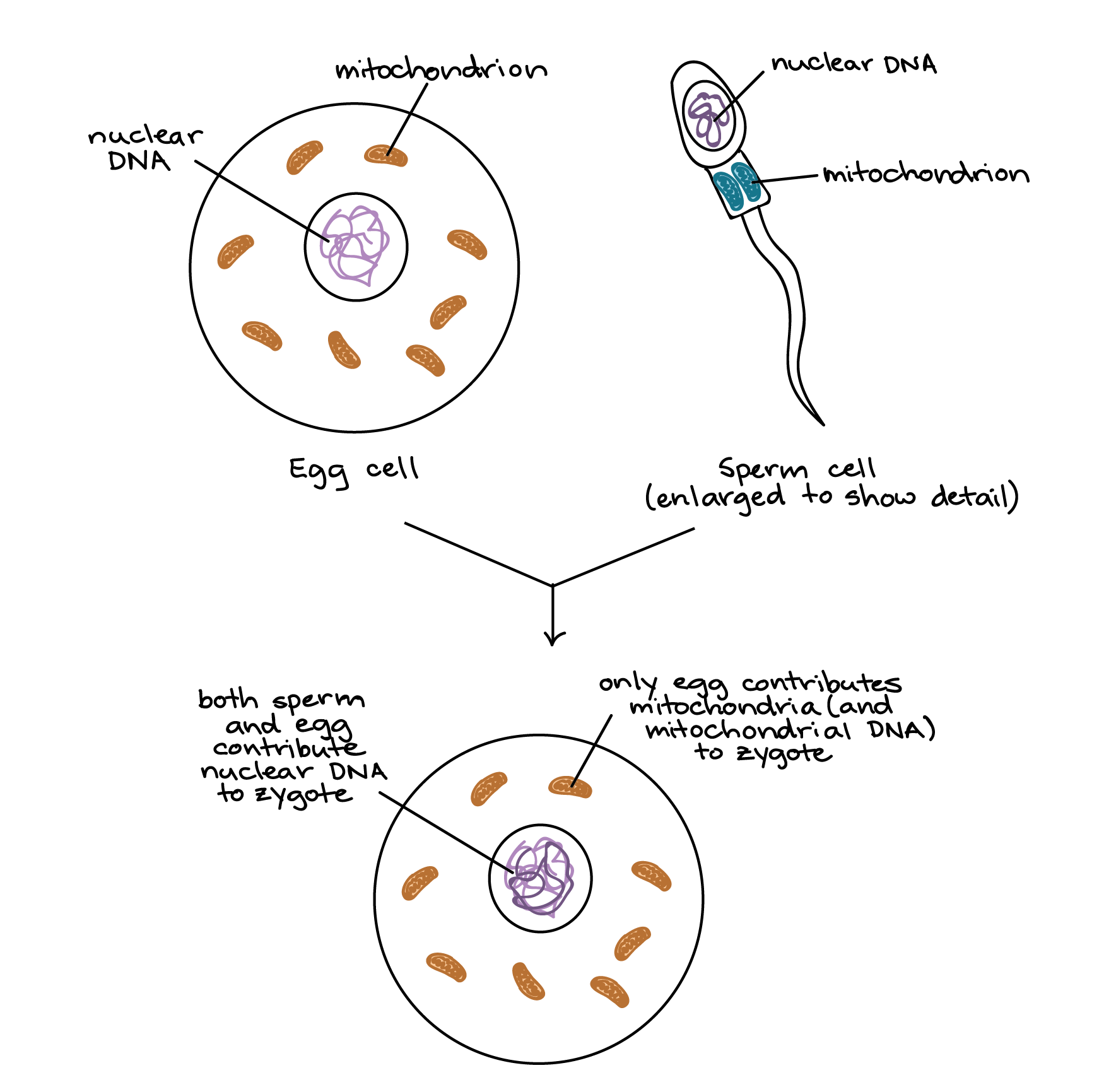 Tanto o espermatozoide quanto o óvulo contêm mitocôndrias e DNA nuclear. Quando se combinam na fecundação, o zigoto resultante conterá DNA nuclear de ambos os pais, mas irá possuir apenas as mitocôndrias (e, portanto, o DNA mitocondrial) do óvulo.
Tanto o espermatozoide quanto o óvulo contêm mitocôndrias e DNA nuclear. Quando se combinam na fecundação, o zigoto resultante conterá DNA nuclear de ambos os pais, mas irá possuir apenas as mitocôndrias (e, portanto, o DNA mitocondrial) do óvulo.Herança maternal de mitocôndrias nos seres humanos
Uma vez que as mitocôndrias são herdadas da mãe, elas fornecem uma forma de rastrear a linhagem matrilinear (linha de descendência através de uma cadeia intacta de ancestrais femininos)Para entender como as mitocôndrias ligam você às antepassadas de sua mãe, considere de onde suas mitocôndrias vieram. Elas foram recebidas de sua mãe, no citoplasma do óvulo que deu origem a você. Onde sua mãe conseguiu suas próprias mitocôndrias? Da mãe dela , que é sua avó materna9start superscript, 9, end superscript.Se você continuar repetindo esta pergunta, você pode voltar no tempo ao longo de sua árvore genealógica, seguindo suas ancestrais maternas e rastreando a rota de transmissão de seu DNA mitocondrial.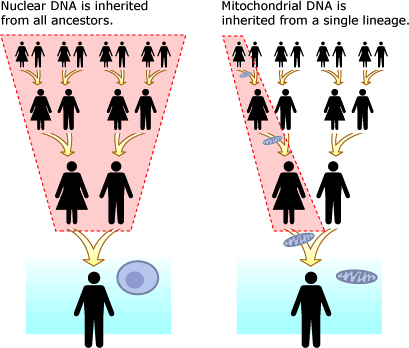 O DNA nuclear é herdado de todos os ancestrais. Ao longo de três gerações, os casais de antepassados tiveram filhos até chegar a uma única pessoa atual que contém o DNA nuclear de oito ancestrais da geração dos bisavós, quatro ancestrais da geração dos avós e dois ancestrais da geração dos pais.O DNA mitocondrial é herdado de uma única linhagem. Ao longo de três gerações, os casais ancestrais tiveram filhos até chegar a uma única pessoa atual que contém o DNA nuclear de oito ancestrais da geração dos bisavós, quatro ancestrais da geração dos avós e dois ancestrais da geração dos pais. Apenas uma mulher em cada geração é o ancestral mitocondrial da pessoa da atualidade: sua mãe (geração dos pais), a mãe de sua mãe (geração dos avós) e a mãe da mãe de sua mãe (geração dos bisavós)._Credito da imagem: "DNA mitocondrial x DNA nuclear," por University of California Museum of Paleontology (CC BY-SA 3.0)._Como mostrado no diagrama acima, o padrão de herança do DNA mitocondrial é diferente daquele do DNA nuclear. O DNA nuclear de uma pessoa é uma "colcha de retalhos" de segmentos herdados de muitos ancestrais diferentes, enquanto o DNA mitocondrial é herdado através de uma única linhagem contínua de ancestrais femininos9,10start superscript, 9, comma, 10, end superscript.
O DNA nuclear é herdado de todos os ancestrais. Ao longo de três gerações, os casais de antepassados tiveram filhos até chegar a uma única pessoa atual que contém o DNA nuclear de oito ancestrais da geração dos bisavós, quatro ancestrais da geração dos avós e dois ancestrais da geração dos pais.O DNA mitocondrial é herdado de uma única linhagem. Ao longo de três gerações, os casais ancestrais tiveram filhos até chegar a uma única pessoa atual que contém o DNA nuclear de oito ancestrais da geração dos bisavós, quatro ancestrais da geração dos avós e dois ancestrais da geração dos pais. Apenas uma mulher em cada geração é o ancestral mitocondrial da pessoa da atualidade: sua mãe (geração dos pais), a mãe de sua mãe (geração dos avós) e a mãe da mãe de sua mãe (geração dos bisavós)._Credito da imagem: "DNA mitocondrial x DNA nuclear," por University of California Museum of Paleontology (CC BY-SA 3.0)._Como mostrado no diagrama acima, o padrão de herança do DNA mitocondrial é diferente daquele do DNA nuclear. O DNA nuclear de uma pessoa é uma "colcha de retalhos" de segmentos herdados de muitos ancestrais diferentes, enquanto o DNA mitocondrial é herdado através de uma única linhagem contínua de ancestrais femininos9,10start superscript, 9, comma, 10, end superscript.Mutações mitocondriais e doenças humanas
Mutações no DNA mitocondrial podem levar a distúrbios genéticos humanos. Por exemplo, grandes deleções no DNA mitocondrial causam uma condição chamada de síndrome de Kearns-Sayre. Estas deleções impedem as mitocôndrias de realizar seu trabalho de extração de energia. A síndrome de Kearns-Sayre pode causar sintomas tais como fraqueza dos músculos, incluindo aqueles que controlam o movimento dos olhos e pálpebras, bem como degeneração da retina e desenvolvimento de doença cardíaca11,12start superscript, 11, comma, 12, end superscript.Distúrbios genéticos causados por mutações mitocondriais não são transmitidos de pais para filhos, visto que as mitocôndrias são fornecidas somente pela mãe. Ao contrário, elas são transmitidas das mães para os filhos das seguintes maneiras13start superscript, 13, end superscript: - Uma pessoa com uma doença causada por uma mutação mitocondrial pode não ter mitocôndrias normais (e ter somente mitocôndrias anormais, portadoras de mutação). Neste caso, uma mãe afetada sempre passará mitocôndrias portadoras de mutação para seus filhos.
- Uma disfunção mitocondrial pode ocorrer quando uma pessoa tem uma mistura de mitocôndrias normais e anormais no seu corpo. Neste caso, as mitocôndrias normais e as mitocôndrias portadoras de mutação são distribuídas aleatoriamente aos óvulos durante a meiose. Filhos que recebem uma grande proporção de mitocôndrias mutantes podem apresentar uma forma severa da doença, enquanto aqueles com poucas mitocôndrias mutantes podem ter uma forma leve ou não apresentar a doença13start superscript, 13, end superscript.
 Diagrama mostrando os padrões de herança de distúrbios causados por mutações no DNA mitocondrial.Pai afetado e mãe não afetada produzem somente filhos não afetadosPai não afetado e mãe afetada, com mitocôndrias uniformemente anormais (portadoras de mutação), produzem somente filhos afetados (presumindo uma completa penetração do distúrbio).Pai não afetado e mãe afetada, com uma mistura de mitocôndrias anormais (portadoras de mutação) e normais, podem produzir filhos com uma variedade de fenótipos, desde não afetados a levemente afetados e, ainda, severamente afetados. Estes fenótipos diferentes refletem a herança de proporções variáveis de mitocôndrias normais e anormais.Imagem modificada de "Mitocondrial," por National Institutes of Health (domínio público).
Diagrama mostrando os padrões de herança de distúrbios causados por mutações no DNA mitocondrial.Pai afetado e mãe não afetada produzem somente filhos não afetadosPai não afetado e mãe afetada, com mitocôndrias uniformemente anormais (portadoras de mutação), produzem somente filhos afetados (presumindo uma completa penetração do distúrbio).Pai não afetado e mãe afetada, com uma mistura de mitocôndrias anormais (portadoras de mutação) e normais, podem produzir filhos com uma variedade de fenótipos, desde não afetados a levemente afetados e, ainda, severamente afetados. Estes fenótipos diferentes refletem a herança de proporções variáveis de mitocôndrias normais e anormais.Imagem modificada de "Mitocondrial," por National Institutes of Health (domínio público).Referências:
- Mears, J. A. (2014). Mitochondrial biogenesis and quality control. In M. F. Hohmann-Marriott, Ed., The structural basis of biological energy generation (pp. 451-476). New York, NY: Springer.
- Lasbury, M. (13 de junho de 2012). Biological fusion energy [Web log post]. Disponível em http://biologicalexceptions.blogspot.com/2012/06/biological-fusion-energy.html.
- Birky, C. W. (2001). The inheritance of genes in mitochondria and chloroplasts: Laws, mechanisms, and models. Annu. Rev. Genet. 35, 128.
- Birky, C. W. (2001). The inheritance of genes in mitochondria and chloroplasts: Laws, mechanisms, and models. Annu. Rev. Genet. 35, 18.
- Hagemann, R. (2000). Erwin Baur or Carl Correns: Who really created the theory of plastid inheritance? The Journal of Heredity, 91(6), 435-440. Disponível em http://www.math.uci.edu/~brusso/BaurCorrens[32,33]sortof.pdf.
- Miko, I. (2008). Non-nuclear genes and their inheritance. Nature Education, 1(1), 135. Disponível em http://www.nature.com/scitable/topicpage/non-nuclear-genes-and-their-inheritance-589.
- Griffiths, A. J. F., Gelbart, W. M., Miller, J. H., e Lewontin, R. C. (1999). Inheritance of organelle genes. In Modern genetic analysis. New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21224/.
- Schwartz, M. and Vissing, J. (2002). Paternal inheritance of mitochondrial DNA. New England Journal of Medicine, 347, 576-580. http://dx.doi.org/10.1056/NEJMoa020350.
- Genealogy enthusiasts mine DNA for clues to evolutionary history. (2007). In Understanding evolution. Disponível em http://evolution.berkeley.edu/evolibrary/news/071101_genealogy.
- Mitochondrial Eve. (19 de dezembro de 2015). Acesso em 20 de dezembro de 2015 em Wikipedia: https://en.wikipedia.org/wiki/Mitochondrial_Eve.
- National Organization for Rare Disorders. (2013). Kearns Sayre syndrome. In Rare disease information. Disponível em https://rarediseases.org/rare-diseases/kearns-sayre-syndrome/.
- Kearns-Sayre syndrome. (2011). In Genetics home reference. Disponível em http://ghr.nlm.nih.gov/condition/kearns-sayre-syndrome.
- Taylor, R. W. and Turnbull, D. M. (2005). Mitochondrial DNA mutations in human disease. Nature Reviews Genetics, 6(5), 389-402. http://dx.doi.org/10.1038/nrg1606. Disponível em http://www.ncbi.nlm.nih.gov/pmc/articles/PMC1762815/.
Outras referências
Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., and Walter, P. (2002). The genetic systems of mitochondria and plastids. In Molecular biology of the cell (4th ed.). New York, NY: Garland Science. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21054/.Birky, C. W. (1983). Nonrandom partitioning of organelle genes. In G. H. Bourne and J. F. Danielli, Eds., Aspects of cell regulation (pp. 84-86). New York, NY: Academic Press.Birky, C. W. (1995). Uniparental inheritance of mitochondrial and chloroplast genes: Mechanisms and evolution. PNAS, 92(25), 11331-11338. Disponível em http://www.pnas.org/content/92/25/11331.abstract.Birky, C. W. (2001). The inheritance of genes in mitochondria and chloroplasts: Laws, mechanisms, and models. Annu. Rev. Genet. 35, 125-48.BluWasabi. (20 de março de 2015). Why do mitochondria fuse together? In Biology stack exchange. Disponível em http://biology.stackexchange.com/questions/30606/why-do-mitochondria-fuse-together.Cann, R. L., Stoneking, M., and Wilson, A. C. (1987). Mitochondrial DNA and human evolution. Nature, 325, 31-36. http://dx.doi.org/10.1038/325031a0. Disponível em http://www.nature.com/scitable/content/Mitochondrial-DNA-and-human-evolution-11488.Chiai, H. and Craig, J. (2008). mtDNA and mitochondrial diseases. Nature Education, 1(1), 217. Disponível em http://www.nature.com/scitable/topicpage/mtdna-and-mitochondrial-diseases-903.Chloroplast DNA. (2 de dezembro de 2015). Disponível em Wikipedia: https://en.wikipedia.org/wiki/Chloroplast_DNA.Hartsock, A. (2015). Bacterial plasmids: Definition, function, & uses. In Study.com. Disponível em http://study.com/academy/lesson/bacterial-plasmids-definition-function-uses.html.Jones, R. E. and Lopez, K. H. (2014). The process of fertilization. In Human reproductive biology (4th ed., pp. 163-169). New York, NY: Academic Press.Khrapko, K. (2008). Two ways to make a mtDNA bottleneck. Nature Genetics, 40(2), 134-135. http://dx.doi.org/10.1038/ng0208-134. Disponível em http://www.ncbi.nlm.nih.gov/pmc/articles/PMC3717270/.Lasbury, M. (13 de junho de 2012). Biological fusion energy [Web log post]. Disponível em http://biologicalexceptions.blogspot.com/2012/06/biological-fusion-energy.html.Miko, I. (2008). Non-nuclear genes and their inheritance. Nature Education, 1(1), 135. Disponível em http://www.nature.com/scitable/topicpage/non-nuclear-genes-and-their-inheritance-589.Mitochondrial DNA. (2014). In Genetics home reference. Disponível em http://ghr.nlm.nih.gov/mitochondrial-dna.Mitochondrial Eve. (19 de dezembro de 2015). Disponível em Wikipedia: https://en.wikipedia.org/wiki/Mitochondrial_Eve.Paternal mtDNA transmission. (29 de outubro de 2015). Disponível em Wikipedia: https://en.wikipedia.org/wiki/Paternal_mtDNA_transmission. Acesso em 20/12/2015.Phillips, R. (2012). How large are chloroplasts? In Physical biology of the cell. Disponível em http://www.rpgroup.caltech.edu/courses/aph161/2012/files_2012/homework/CBN-2012-Binder2.pdf.Pierce, B. A. (2009). Cytoplasmic inheritance. In Transmission and population genetics (3rd ed., pp. 117-119). New York, NY: W. H. Freeman and Company.Poznik, G. D., Henn, B. M., Yee, M.-C., Sliwerska, E., Euskirchen, G. M., Lin, A. A., Snyder, M., Quintana-Murci, L., Kidd, J. M., Underhill, P. A., and Bustamante, C. D. (2013). Sequencing Y chromosomes resolves discrepancy in time to common ancestor of males versus females. Science, 341, 562-565. http://dx.doi.org/10.1126/science.1237619. Disponível em http://www.ncbi.nlm.nih.gov/pmc/articles/PMC4032117/.Purves, W. K., Sadava, D. E., Orians, G. H., and Heller, H.C. (2004). Non-nuclear inheritance. In Life: the science of biology (7th ed., p. 209). Sunderland, MA: Sinauer Associates.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Inheritance of organelle genes. In Campbell biology (10th ed., p. 309). San Francisco, CA: Pearson.Tychonievich, J. (24 de julho de 2011). Sciency answers: Variegation part 2. Stripes and splotches! [Web log post]. Disponível em http://blog.arrowheadalpines.com/2011/07/sciency-answers-variegation-part-2.html.Wallace, D. C. and Chalkia, D. (2013). Mitochondrial DNA genetics and the heteroplasmy conundrum in evolution and disease. Cold Spring Harbor Perspectives in Biology. Disponível em http://dx.doi.org/10.1101/cshperspect.a021220.Ymar. (19 de agosto de 2012). Does every mitochondrion in a cell contain the same DNA? In Biology stack exchange. Disponível em http://biology.stackexchange.com/questions/3245/does-every-mitochondrion-in-a-cell-contain-the-same-dna(Abre em uma nova janela).- https://youtu.be/u5SGwPEMh9c
- https://youtu.be/YWNB_yVySxQ
- Exercícios de Fixação
- 1- A síndrome de Down é causada por uma anomalia cromossômica em que o portador apresenta células com três cromossomos do número 21. Os afetados apresentam geralmente baixa estatura, pescoço relativamente curto, olhos oblíquos, mãos curtas e largas, entre outras características. Essa síndrome é um exemplo de:
a) Euploidia.
b) Aneuploidia.
c) Deleção.
d) Duplicação.
e) Translocação.
2- Sabemos que as aberrações cromossômicas podem ser numéricas ou estruturais. Marque a alternativa que indica corretamente os processos que levam a alterações estruturais:a) Euploidia, aneuploidia e duplicação.
b) Duplicação, transcrição e tradução.
c) Deleção, translocação e transcrição.
d) Deleção, inversão e translocação.
e) Inversão, deleção e transcrição.
3- Nas aneuploidias, um tipo de aberração cromossômica numérica, ocorrem alterações no número de um ou mais pares de cromossomos. Um exemplo de aneuploidia é a síndrome de Turner, em que o indivíduo apresenta apenas um cromossomo X, sendo, portanto, sempre do sexo feminino. Nesse caso ocorre uma pessoa com baixa estatura, ovários atrofiados, pescoço alado, deficiência hormonal, entre outras características. Esse tipo de aneuploidia recebe o nome de:a) Nulissomia.
b) Monossomia.
c) Trissomia.
d) Tetrassomia.
I – As mutações numéricas podem ser classificadas em dois tipos: euploidias e aneuploidias.
II – Euploidia é quando ocorre perda ou acréscimo de um ou alguns cromossomos. Aneuploidia é quando ocorre perda ou acréscimo de genomas (formando-se células 3n, 4n e assim por diante).
III – Euploidia é quando ocorre perda ou acréscimo de genomas. Aneuploidia é quando ocorre perda ou acréscimo de um ou alguns cromossomos.
IV – A síndrome de Down e a síndrome de Turner são alguns exemplos de aneuploidias.
Estão corretas:
a) I e II, apenas
b) I e III, apenas
c) I, II e IV, apenas
d) I, III e IV, apenas
e) III e IV, apenas
