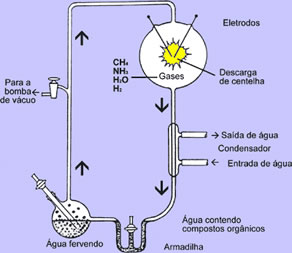
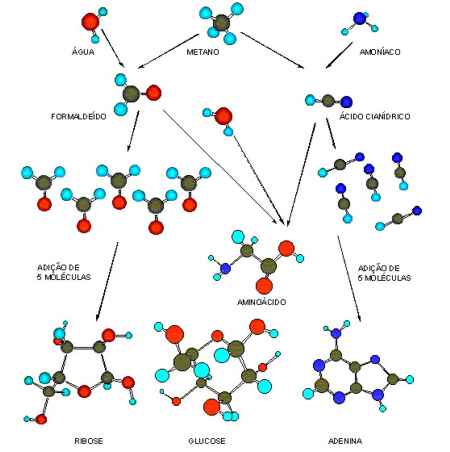
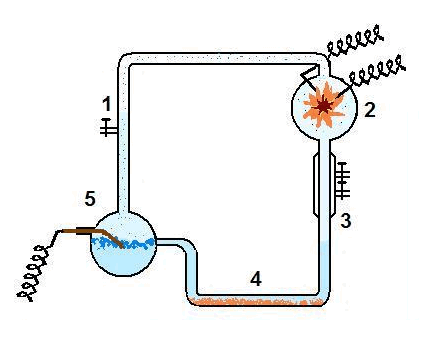
2° Ano E Biologia Profa Solange
Período para entrega: Até 23/09/2020
Unidade Temática: DNA como material genético- DNA- Receita da Vida e seu Código.
Instruções:
Retomando Conceitos- DNA Receita da Vida e seu Código
1- Assistir os vídeos (Khan Academy) links disponibilizados abaixo.
2- Fazer a Leitura e Interpretação de Texto.
3- Responder a atividade: Exercícios de Fixação.
4- Postar no Blogger e enviar para o e-mail da professora: solangestandbyme@gmail.com
https://youtu.be/U1hnrmyxk1M
https://youtu.be/Qen2RWLvCMc
https://youtu.be/U1hnrmyxk1M
https://youtu.be/AKngKsb-bZw
https://youtu.be/gC-w8UYcayA
https://youtu.be/U1hnrmyxk1M
https://youtu.be/cSCQ1NW3DrI
Introdução à expressão dos genes (dogma central)
Resumo: a expressão gênica
Genes especificam produtos funcionais (como proteínas)

Como a sequência do DNA de um gene especifica uma determinada proteína?
- Na transcrição, a sequência de DNA de um gene é copiada para fazer uma molécula de RNA. Essa etapa é chamada de transcrição pois envolve reescrever, ou transcrever, a sequência de DNA num "alfabeto" similar de RNA. Nos eucariontes, a molécula de RNA deve passar por um processamento para se tornar um RNA mensageiro (RNAm) maduro.
- Na tradução, a sequência de RNAm é decodificada para determinar a sequência de aminoácidos de um polipeptídeo. O nome tradução significa que a sequência do RNAm precisa ser traduzida numa "linguagem" de aminoácidos completamente diferente.
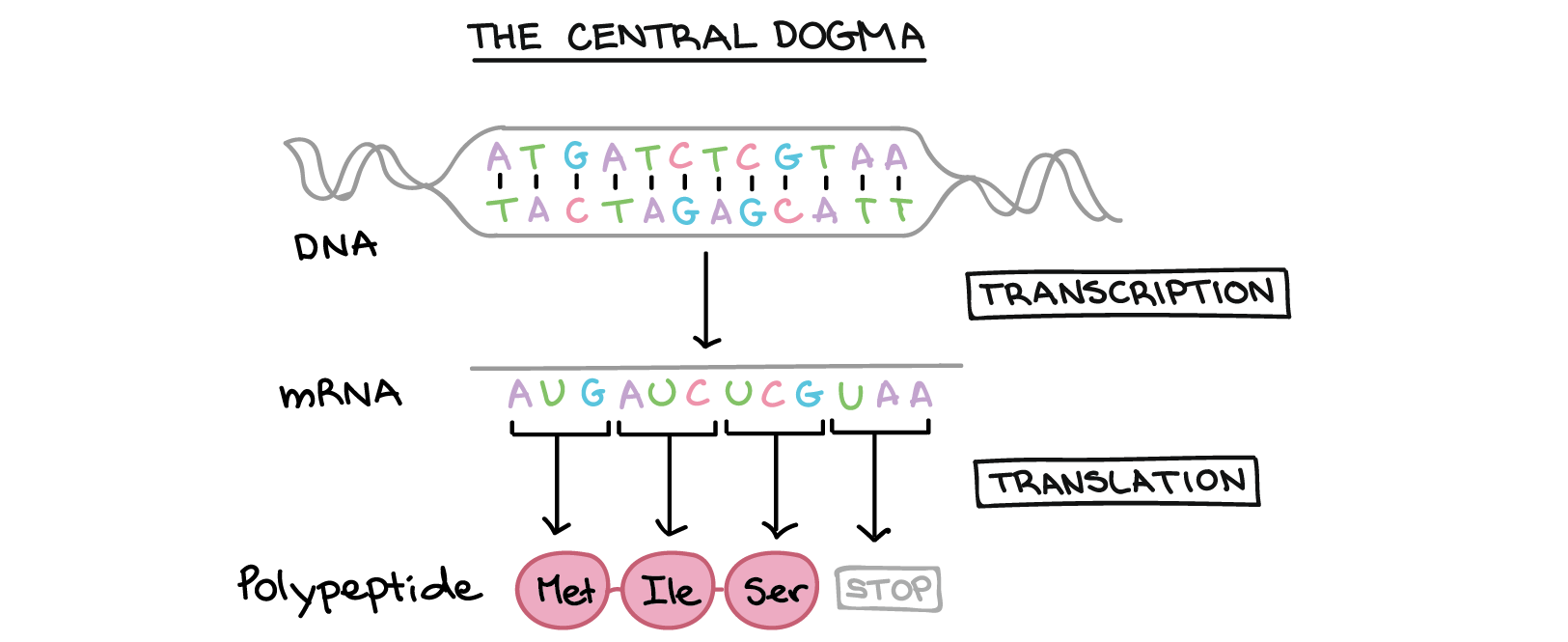
Assim, durante a expressão de um gene codificante de proteína, a informação flui do DNA →right arrow RNA →right arrow proteína. Esse fluxo direcional de informação é conhecido como o dogma central da biologia molecular. Genes não codificantes de proteína (genes que especificam RNAs funcionais) ainda são transcritos para produzir um RNA, mas esse RNA não é traduzido em um polipeptídeo. Tanto para um quanto para outro tipo de gene, o processo de ir de um DNA para um produto funcional é conhecido como expressão gênica.
Saiba mais:
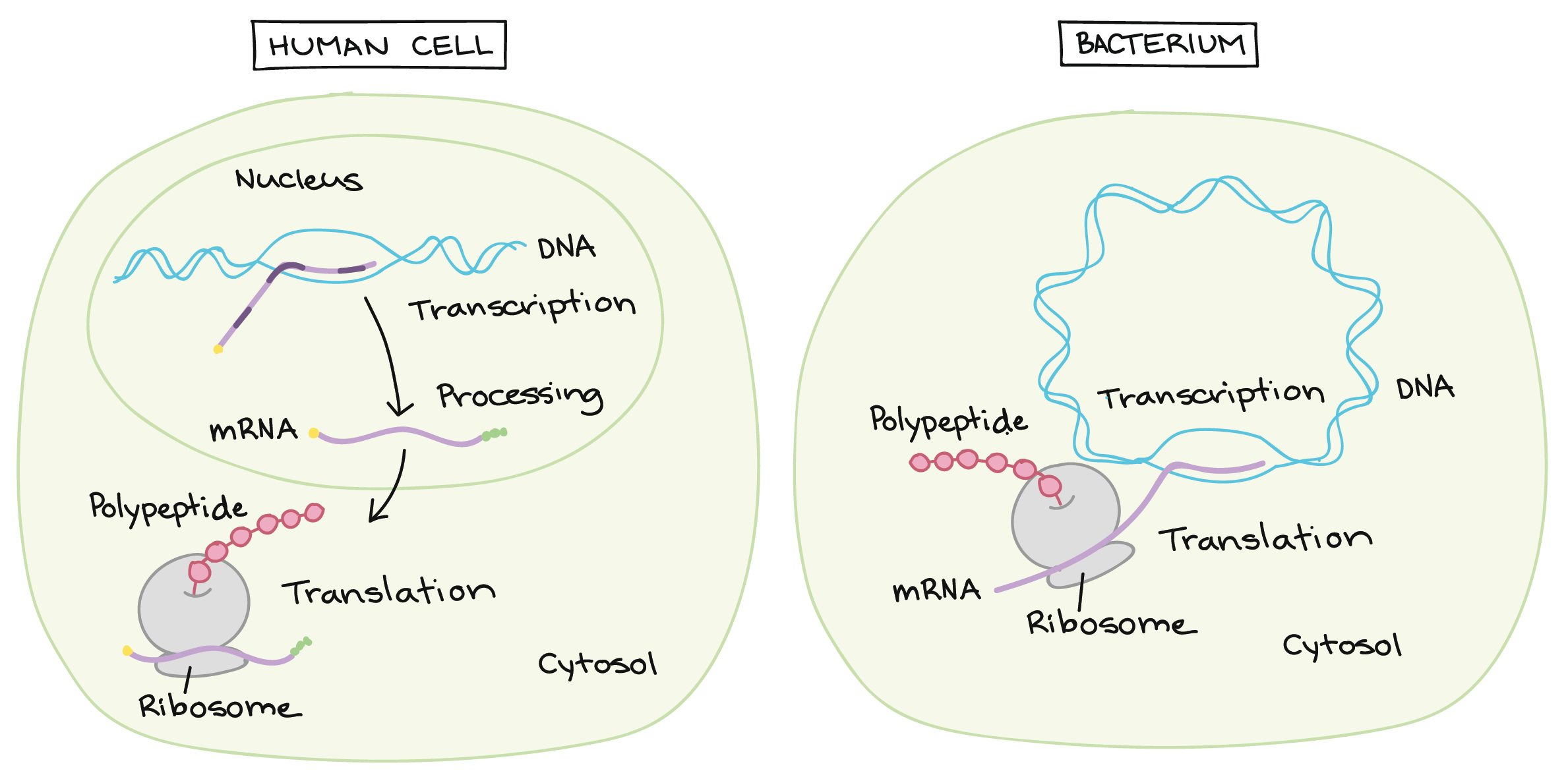
- Uma razão simples relaciona-se à localização. Em uma célula eucariótica, o DNA está fechado no núcleo, enquanto os ribossomos - máquinas moleculares usadas para fazer proteínas - estão no citoplasma. Assim, é preciso usar um "mensageiro" que transporte a informação do DNA para fora do núcleo para os ribossomos. Os RNAs mensageiros desempenham este papel.
- A transcrição também fornece um ponto de controle importante no qual as células regulam quanto de um polipeptídeo é produzido. Embora outras etapas da expressão gênica possam também ser reguladas, o controle da transcrição é a forma mais comum da regulação gênica. Se a etapa da transcrição fosse de alguma maneira removida, as células perderiam muito de seu controle sobre quais polipeptídeos seriam produzidos e quando.
Transcrição
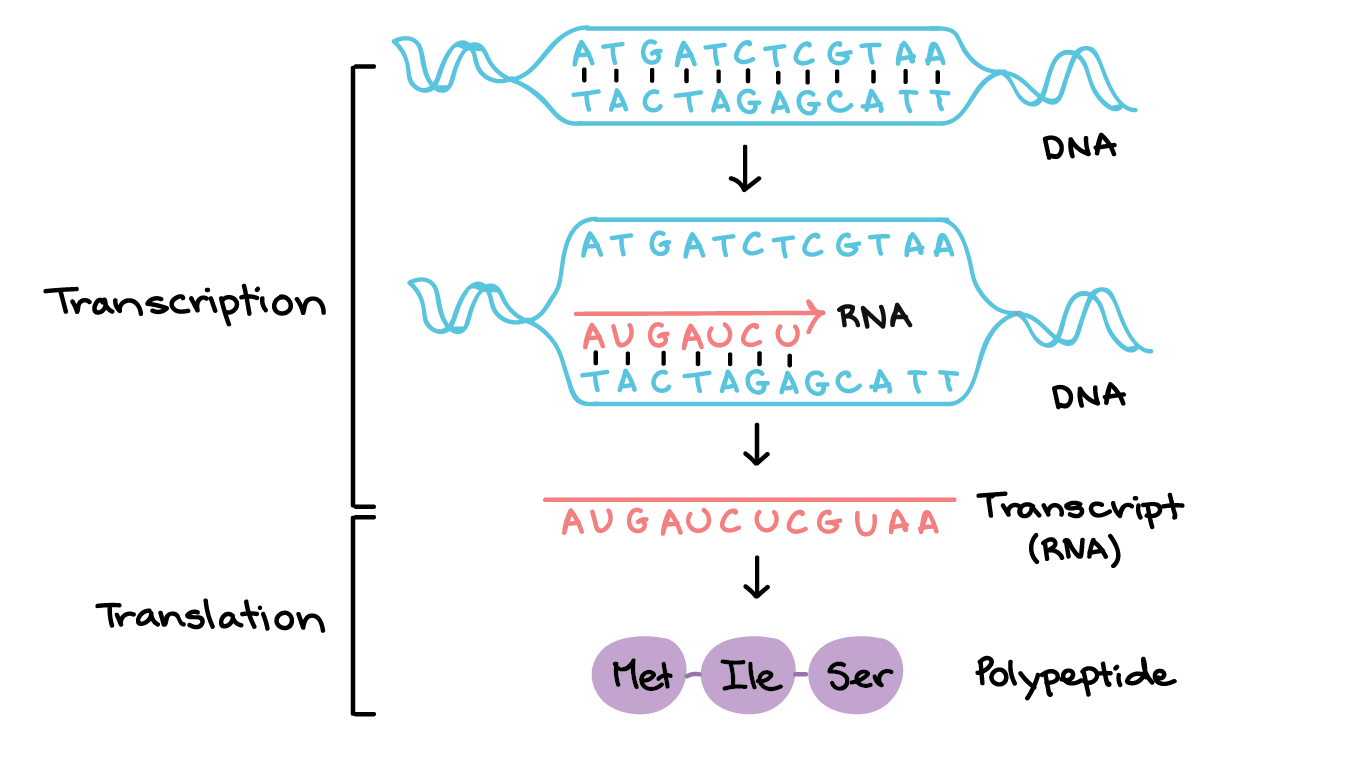
Na transcrição, uma fita de DNA que compõe um gene, chamada de fita não codificante, age como molde para a síntese de uma fita correspondente (complementar) de RNA por uma enzima chamada RNA polimerase. Essa fita de RNA é o transcrito primário.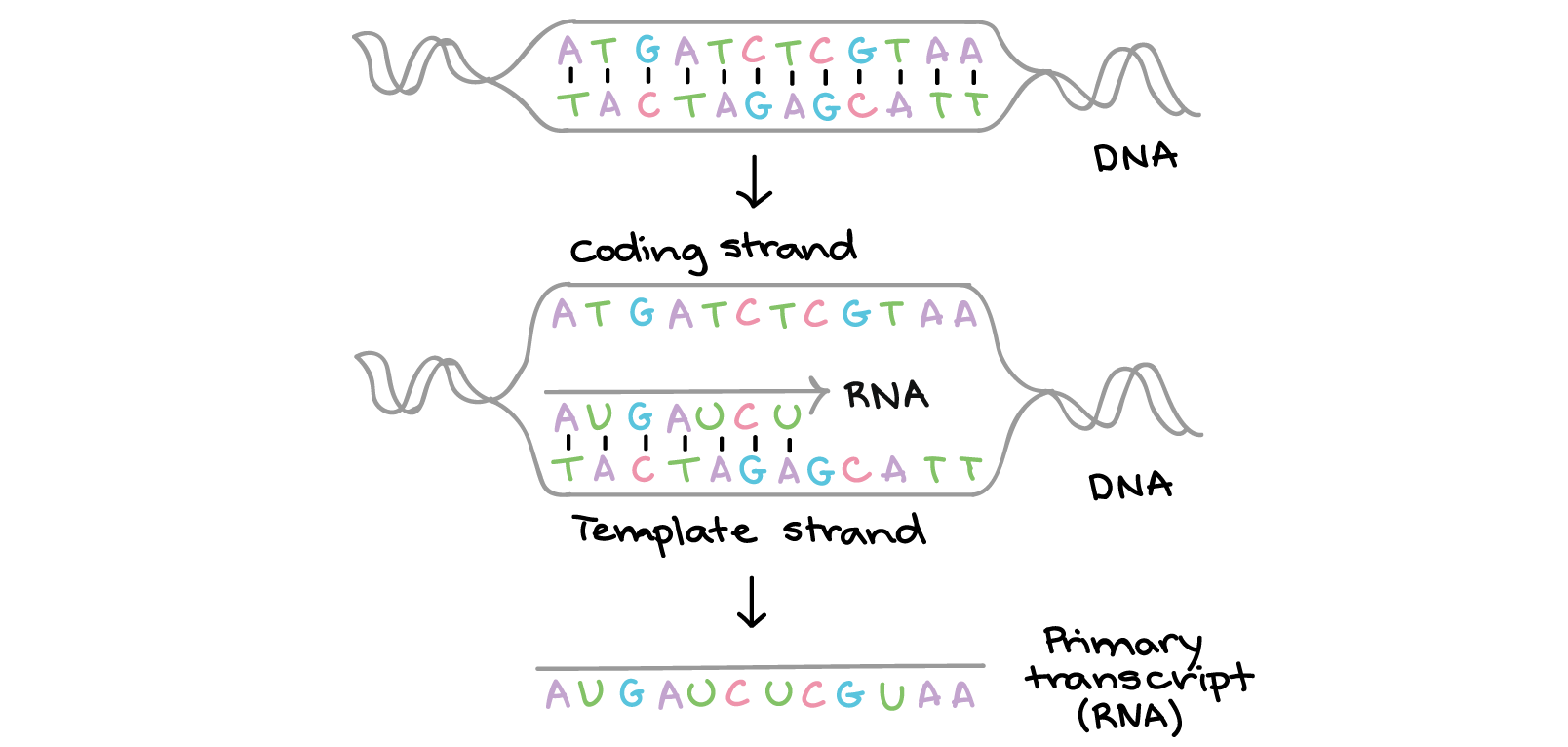 As duas fitas de DNA têm as seguintes sequências:5'-ATGATCTCGTAA-3' 3'-TACTAGAGCATT-5'O DNA se abre para formar um bolha, e a fita inferior serve de molde para a síntese de uma fita complementar de RNA. Essa fita é chamada de fita molde. A transcrição da fita molde produz um RNAm quase igual a outra fita (fita codificante) do DNA em sequência. Contudo, devido a diferenças bioquímicas entre DNA e RNA, os Ts do DNA são substituídos por Us no RNAm. A sequência do RNAm é:5'-AUGAUCUCGUAA-5'O transcrito primário carrega a mesma sequência de informação que a fita de DNA não transcrita, algumas vezes chamada de fita codificante. Contudo, o transcrito primário e a fita codificante de DNA não são idênticos, graças a algumas diferenças bioquímicas entre DNA e RNA. Uma diferença importante é que as moléculas de RNA não incluem a base timina (T). Ao invés disso, elas têm uma base similar uracila (U). Como a timina, a uracila pareia com adenina.
As duas fitas de DNA têm as seguintes sequências:5'-ATGATCTCGTAA-3' 3'-TACTAGAGCATT-5'O DNA se abre para formar um bolha, e a fita inferior serve de molde para a síntese de uma fita complementar de RNA. Essa fita é chamada de fita molde. A transcrição da fita molde produz um RNAm quase igual a outra fita (fita codificante) do DNA em sequência. Contudo, devido a diferenças bioquímicas entre DNA e RNA, os Ts do DNA são substituídos por Us no RNAm. A sequência do RNAm é:5'-AUGAUCUCGUAA-5'O transcrito primário carrega a mesma sequência de informação que a fita de DNA não transcrita, algumas vezes chamada de fita codificante. Contudo, o transcrito primário e a fita codificante de DNA não são idênticos, graças a algumas diferenças bioquímicas entre DNA e RNA. Uma diferença importante é que as moléculas de RNA não incluem a base timina (T). Ao invés disso, elas têm uma base similar uracila (U). Como a timina, a uracila pareia com adenina.
Transcrição e processamento de RNA: Eucariontes vs. bactérias
Tradução
O código genético
Etapas da tradução
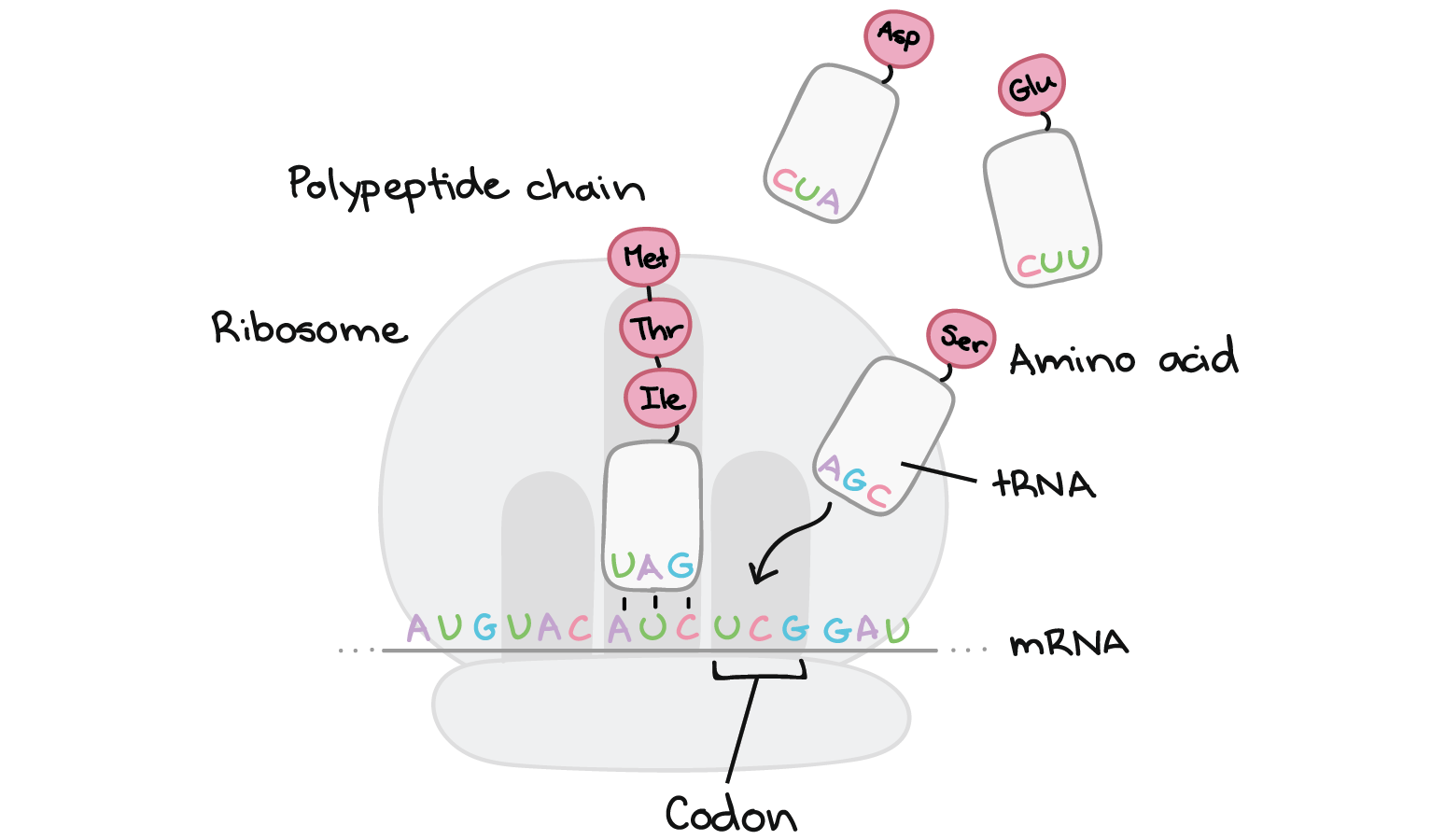
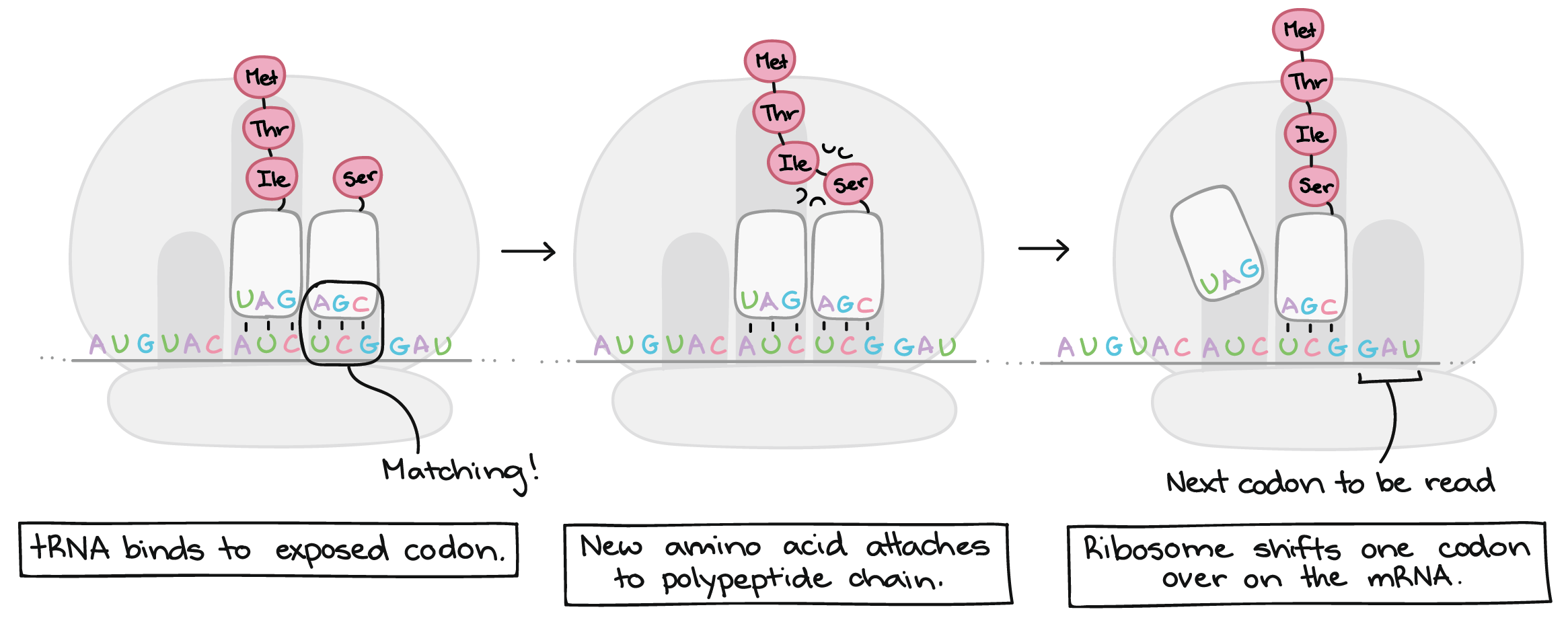
- O tRNA correspondente se liga ao códon exposto no encaixe mais à direita do ribossomo.
- A cadeia de aminoácidos é transferida do RNAt no meio do encaixe do ribossomo para o aminoácido do RNAt na extrema direita do encaixe. Isto tem o efeito de adicionar o aminoácido no final da cadeia de aminoácido.
- O ribossomo desloca-se sobre um códon. O RNAt anteriormente no encaixe do meio, move-se para o encaixe mais à esquerda e sai do ribossomo.O RNAt que estava no encaixe da direita move-se para o encaixe do meio e continua preso à cadeia de aminoácido. Um novo códon fica exposto no encaixe mais à direita permitindo sua união com um novo RNAt .
O que acontece em seguida?
Resumo:
- O DNA é dividido em unidades funcionais chamadas genes, que podem especificar polipeptídeos (proteínas e subunidades de proteínas) ou RNAs funcionais (como RNAt e RNAr).
- Informação de um gene é usada para construir um produto funcional em um processo chamado expressão gênica.
- Um gene que codifica um polipeptídeo é expresso em duas etapas. Nesse processo, a informação flui do DNA →right arrow RNA →right arrow proteína, uma relação direcional conhecida como dogma central da biologia molecular.
- Transcrição: Uma fita do DNA do gene é copiada para RNA. Nos eucariontes, o transcrito de RNA deve passar por etapas adicionais de processamento para se tornar um RNA mensageiro (RNAm) maduro.
- Tradução: A sequência de nucleotídeos do RNAm é decodificada para especificar a sequência de aminoácidos de um polipeptídeo. Este processo ocorre dentro de um ribossomo e requer moléculas adaptadoras chamadas de RNAt.
- Durante a tradução, os nucleotídeos do RNAm são lidos em grupos de três, chamados códons. Cada códon especifica um aminoácido em particular ou um sinal de parada. Esse conjunto de relações é conhecido como o código genético.
Este artigo está autorizado sob licença CC BY-NC-SA 4.0.Referências:
- Hellens, R. P., Moreau, C., Lin-Wang, K., Schwinn, K. E., Thomson, S. J., Fiers, M. W. E. J., . . . Noel Ellis, T. H. (2010, October 11). Identification of Mendel's white flower character. PLOS ONE. http://dx.doi.org/10.1371/journal.pone.0013230.
- Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Figura 14.4. Alleles, alternative versions of a gene. Em Campbell biology (10th ed., p. 271). San Francisco, CA: Pearson.
- CyberBridge. (2007). RNA structure. Em Structure of DNA. Disponível em http://cyberbridge.mcb.harvard.edu/dna_3.html.
Referências:
Hellens, R. P., Moreau, C., Lin-Wang, K., Schwinn, K. E., Thomson, S. J., Fiers, M. W. E. J., . . . Noel Ellis, T. H. (2010, October 11). Identification of Mendel's white flower character. PLOS ONE. http://dx.doi.org/10.1371/journal.pone.0013230.OpenStax College, Biology. (2015, December 29). The genetic code. Em OpenStax CNX. Disponível em http://cnx.org/contents/GFy_h8cu@9.87:QEibhJMi@8/The-Genetic-Code.Purves, W. K., Sadava, D. E., Orians, G. H., and Heller, H.C. (2004). DNA, RNA, and the flow of information. Em Life: the science of biology (7th ed., pp. 236-237). Sunderland, MA: Sinauer Associates.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Genes specify proteins via transcription and translatioin. Em Campbell biology (10th ed., pp. 334-340). San Francisco, CA: Pearson.Agradecimentos:
Agradecimentos a Willy McAllister pelos relevantes comentários sobre este artigo.O código genético
Introdução
Você já escreveu uma mensagem secreta para algum dos seus amigos? Em caso positivo, você pode ter usado um código para manter a mensagem secreta. Por exemplo, você pode ter substituído as letras de uma palavra por números ou símbolos, seguindo um conjunto particular de regras. Para que seu amigo entenda a mensagem, ele precisa conhecer o código e aplicar o mesmo conjunto de regras para decodificar a mensagem.Decifrar mensagens também é um passo chave na expressão genética, na qual a informação de um gene é lida para construir uma proteína. Neste artigo, olharemos mais de perto o código genético, que permite que as sequências de DNA e RNA sejam "decodificadas" em aminoácidos de uma proteína.Contexto: Fazendo uma proteína
Os genes que fornecem instruções para proteínas são expressos num processo de duas etapas.- Na transcrição, a sequência de DNA de um gene é "reescrita" em RNA. Nos eucariontes, o RNA deve passar por etapas adicionais de processamento para se tornar um RNA mensageiro ou RNAm.
- Na tradução , a sequência de nucleotídeos do RNAm é "traduzida" em uma sequência de aminoácidos de um polipeptídeo (cadeia proteica).
Se este é um novo conceito para você, aprenda mais assistindo ao vídeo de Sal sobre transcrição e tradução.Códons
As células decodificam mRNAs lendo seus nucleotídeos em grupos de três, chamados de códons. Aqui estão algumas características dos códons:- A maioria dos códons especifica um aminoácido
- Três "códons de parada" marcam o fim de uma proteína
- Um "códon de início", AUG, marca o início de uma proteína e também codifica o aminoácido metionina.
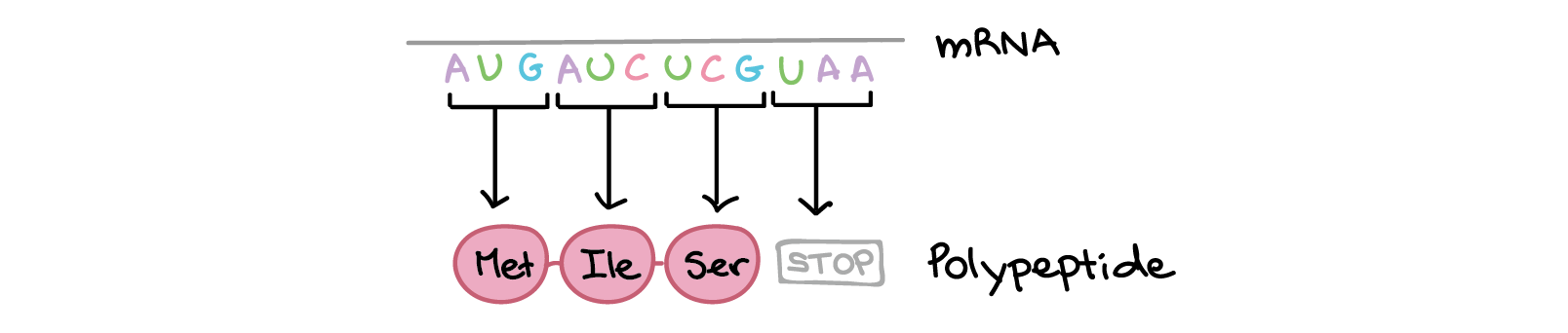
Os códons em um RNAm são lidos durante a tradução, começando com um códon de início e continuando até que um códon de parada é alcançado. Os códons de RNAm são lidos de 5' para 3', e especificam a ordem dos aminoácidos em uma proteína da região N-terminal (metionina) para a C-terminal.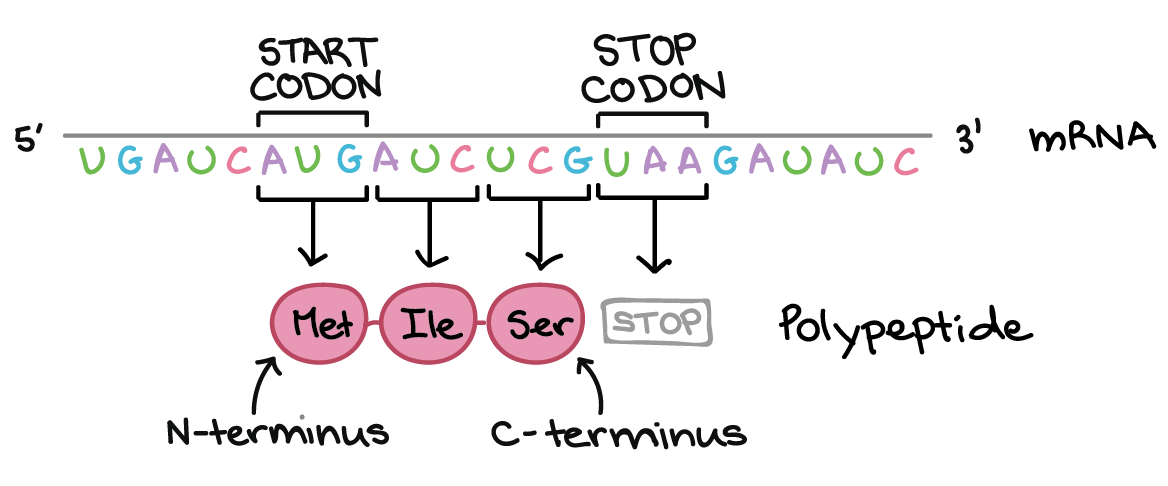 A sequência de RNAm é:5'-AUGAUCUCGUAA-5'A tradução envolve a leitura dos nucleotídeos do RNAm em grupos de três, cada qual especifica um aminoácido (ou fornece um sinal de parada indicando que a tradução terminou).3'-AUG AUC UCG UAA-5'AUG →right arrow Metionina (Início) AUC →right arrow Isoleucina UCG →right arrow Serina UAA →right arrow "Parada"Sequência de polipeptídeo: (N-terminal) Metionina-Isoleucina-Serina (C-terminal)Saiba mais:Os dois finais de uma fita de DNA ou RNA são diferentes um do outro. Isto é, uma molécula de DNA ou RNA tem direcionalidade.
A sequência de RNAm é:5'-AUGAUCUCGUAA-5'A tradução envolve a leitura dos nucleotídeos do RNAm em grupos de três, cada qual especifica um aminoácido (ou fornece um sinal de parada indicando que a tradução terminou).3'-AUG AUC UCG UAA-5'AUG →right arrow Metionina (Início) AUC →right arrow Isoleucina UCG →right arrow Serina UAA →right arrow "Parada"Sequência de polipeptídeo: (N-terminal) Metionina-Isoleucina-Serina (C-terminal)Saiba mais:Os dois finais de uma fita de DNA ou RNA são diferentes um do outro. Isto é, uma molécula de DNA ou RNA tem direcionalidade.- Na extremidade 5' da cadeia, o grupo fosfato do primeiro nucleotídeo está livre. O grupo fosfato é ligado ao carbono 5' do anel de açúcar, por isto chama-se de extremidade 5'.
- Na outra extremidade, chamada de extremidade 3', a hidroxila do último nucleotídeo adicionado à cadeia fica exposta. O grupo hidroxila é ligado ao carbono 3' do anel de açúcar, por isto chama-se de extremidade 3'.
Muitos processos, como a replicação e transcrição de DNA, podem apenas ocorrer em uma direção particular relativa à direcionalidade da fita de DNA ou RNA.Você pode ler mais sobre isso no artigo sobre ácidos nucleicos.
Os polipeptídeos (cadeias de aminoácidos ligados) têm dois finais distintos:- Um N-terminal com um grupo amino exposto
- Um C-terminal com um grupo carboxila exposto
Durante a tradução, o polipeptídeo é feito do N- para o C-terminal. Você pode aprender mais sobre os terminais N- e C- no artigo proteínas e aminoácidos. A tabela do código genético
O conjunto completo de relações entre códons e aminoácidos (ou sinais de parada) é chamado de código genético. O código genético é, muitas vezes, resumido em uma tabela.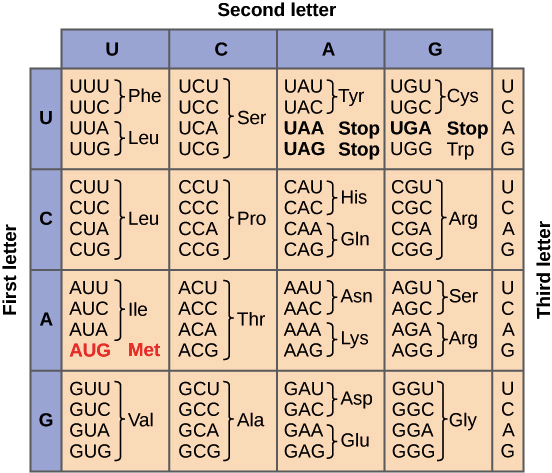 Tabela de código genético. Cada sequência de três letras de nucleotídeos do RNAm corresponde a um aminácido específico ou um códon de parada. UGA, UAA e UAG são códons de parada. AUG é o códon da metionina e também é o códon de início.Crédito da imagem: "The genetic code," por OpenStax College, Biology (CC BY 3.0).Note que muitos aminoácidos são representados na tabela por mais de um códon. Por exemplo, existem seis maneiras diferentes de "escrever" leucina na linguagem de RNAm (veja se você consegue achar todas as seis).Um ponto importante sobre o código genético é que ele é universal. Ou seja, com pequenas exceções, praticamente todas as espécies (de bactéria até você) utilizam o código genético mostrado acima para a síntese de proteínas.
Tabela de código genético. Cada sequência de três letras de nucleotídeos do RNAm corresponde a um aminácido específico ou um códon de parada. UGA, UAA e UAG são códons de parada. AUG é o códon da metionina e também é o códon de início.Crédito da imagem: "The genetic code," por OpenStax College, Biology (CC BY 3.0).Note que muitos aminoácidos são representados na tabela por mais de um códon. Por exemplo, existem seis maneiras diferentes de "escrever" leucina na linguagem de RNAm (veja se você consegue achar todas as seis).Um ponto importante sobre o código genético é que ele é universal. Ou seja, com pequenas exceções, praticamente todas as espécies (de bactéria até você) utilizam o código genético mostrado acima para a síntese de proteínas.Pauta de leitura
Para obter, de uma forma confiável, uma proteína a partir do RNAm, nós precisamos de mais um conceito: o de pauta de leitura. A pauta de leitura determina como a sequência de RNAm é dividida em códons durante a tradução.Esse é um conceito bastante abstrato, então vamos olhar para um exemplo para entender melhor. O RNAm abaixo consegue codificar três proteínas diferentes, dependendo da pauta em que é lida: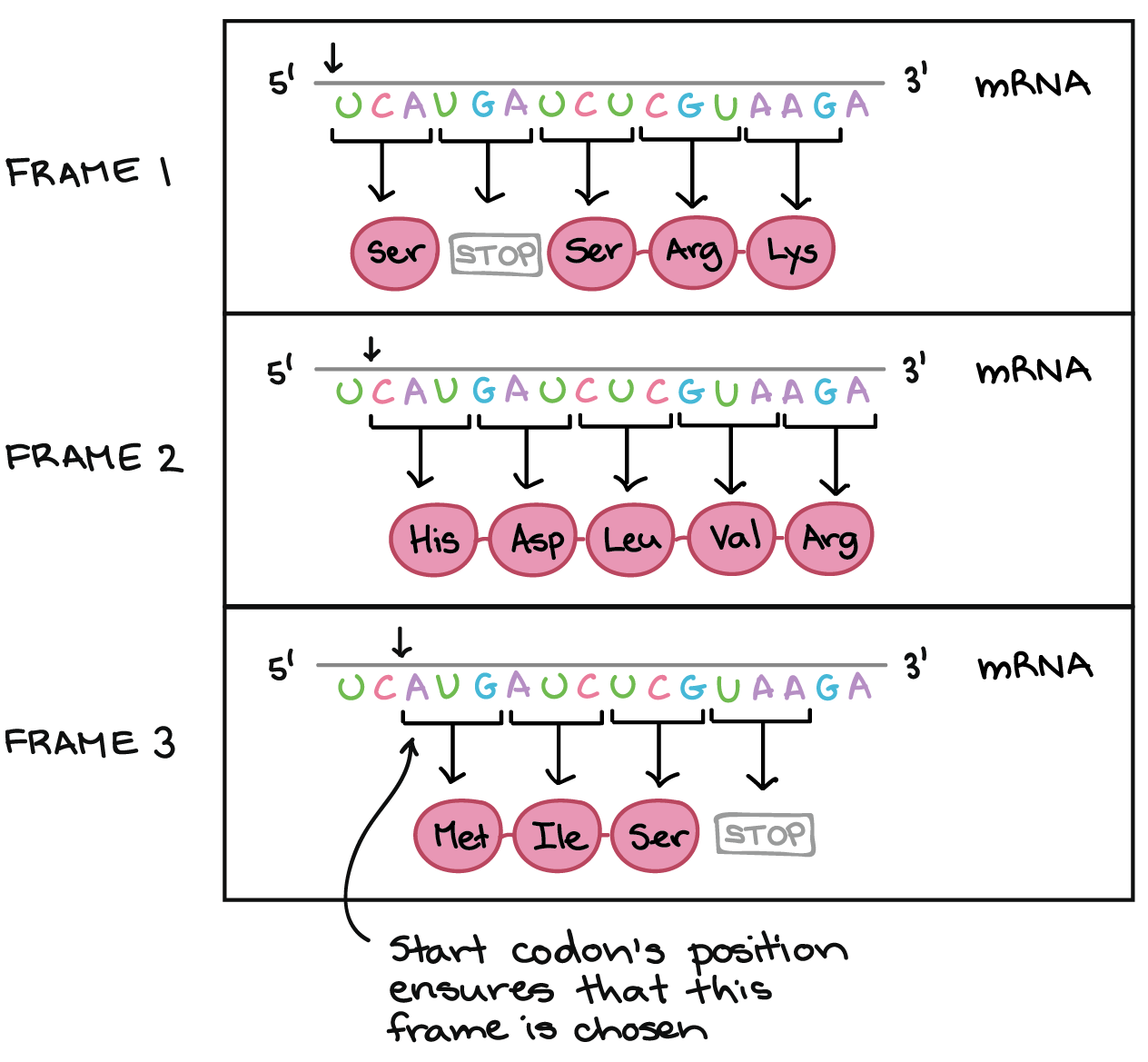 Sequência de RNAm: 5'-UCAUGAUCUCGUAAGA-3'Leia na Pauta 1:5'-UCA UGA UCU CGU AAG A-3'Ser-PARADA-Ser-Arg-LysLeia na Pauta 2:5'-U CAU GAU CUC GUA AGA-3'Sua-Asp-Leu-Val-ArgLeia na Pauta 3:5'-UC AGO AUC UEG UAA GA-3'Met(Início)-Ile-Ser-PARADAA posição do códon de início garante que a Pauta 3 seja escolhida para a tradução do RNAm.Então, como uma célula sabe qual destas proteínas deve fazer? O códon de início é o sinal chave. Como a tradução começa no códon de início e continua em grupos sucessivos de três, a posição do códon de início garante que o RNAm seja lido da forma correta (no exemplo acima, na pauta 3).Mutações (mudanças no DNA) que inserem ou deletam um ou dois nucleotídeos podem mudar a pauta de leitura, causando a produção de uma proteína incorreta "a jusante" do sítio de mutação:
Sequência de RNAm: 5'-UCAUGAUCUCGUAAGA-3'Leia na Pauta 1:5'-UCA UGA UCU CGU AAG A-3'Ser-PARADA-Ser-Arg-LysLeia na Pauta 2:5'-U CAU GAU CUC GUA AGA-3'Sua-Asp-Leu-Val-ArgLeia na Pauta 3:5'-UC AGO AUC UEG UAA GA-3'Met(Início)-Ile-Ser-PARADAA posição do códon de início garante que a Pauta 3 seja escolhida para a tradução do RNAm.Então, como uma célula sabe qual destas proteínas deve fazer? O códon de início é o sinal chave. Como a tradução começa no códon de início e continua em grupos sucessivos de três, a posição do códon de início garante que o RNAm seja lido da forma correta (no exemplo acima, na pauta 3).Mutações (mudanças no DNA) que inserem ou deletam um ou dois nucleotídeos podem mudar a pauta de leitura, causando a produção de uma proteína incorreta "a jusante" do sítio de mutação: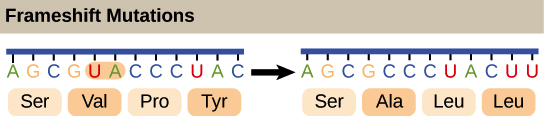 A ilustração mostra uma mutação por deslocamento de leitura em que a pauta de leitura é alterada pela deleção de dois aminoácidos._Crédito da imagem; "O código genético: Figura 3," por OpenStax College, Biology, CC BY 4.0._
A ilustração mostra uma mutação por deslocamento de leitura em que a pauta de leitura é alterada pela deleção de dois aminoácidos._Crédito da imagem; "O código genético: Figura 3," por OpenStax College, Biology, CC BY 4.0._Como o código genético foi descoberto?
A história de como o código genético foi descoberto é muito legal e épica. Nós escondemos a nossa versão no pop-up abaixo, para não distraí-lo caso você esteja com pressa. Mas, se você tiver algum tempo, é uma leitura definitivamente interessante.Eu sempre gosto de imaginar o quão legal teria sido ser uma das pessoas que decifraram o código molecular básico da vida. Apesar de agora conhecermos o código, há muitos outros mistérios biológicos ainda aguardando para serem decifrados (talvez por você!).Créditos:
Este artigo é proveniente da modificação de "Divisão celular," por OpenStax College, Biology, CC BY 4.0. Baixe o artigo original sem custos em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@10.53.O artigo adaptado está autorizado sob a licença CC BY-NC-SA 4.0Referências:
- Lorch, M. (2012, August 16). The most beautiful wrong ideas in science. Em Chemistry blog. Obtido em http://www.chemistry-blog.com/2012/08/16/the-most-beautiful-wrong-ideas-in-science/.
- Nirenberg, M. (2004). Historical review: Deciphering the genetic code – a personal account. TRENDS in Biochemical Sciences, 29(1), 46-54. http://dx.doi.org/10.1016/j.tibs.2003.11.009.
- Gellene, Denise. (2011, Novembro 14). H. Gobind Khorana, morre cientista de 89 anos vencedor do Nobel. The New York Times. Obtido de: http://www.nytimes.com/2011/11/14/us/h-gobind-khorana-1968-nobel-winner-for-rna-research-dies.html?_r=0.
- H. Gobind Khorana – Nobel Lecture. NobelPrize.org. Nobel Media AB 2019. Segunda-feira. 6 Maio 2019. https://www.nobelprize.org/prizes/medicine/1968/khorana/lecture/
Referências:
Arnaud, M.B., Inglis, D.O., Skrzypek, M.S., Binkley, J., Shah, P., Wymore, F., Binkley, G., Miyasato, S.R., Simison, M., and Sherlock, G. (2013). CGD help: Non-standard genetic codes. In Candida genome database. Obtido em http://www.candidagenome.org/help/code_tables.shtml.Codon. (2014). In Scitable. Obtido em http://www.nature.com/scitable/definition/codon-155.Gellene, Denise. (14 de novembro de 2011). H. Gobind Khorana, 89, Nobel-winning scientist, dies. The New York Times. Obtido em http://www.nytimes.com/2011/11/14/us/h-gobind-khorana-1968-nobel-winner-for-rna-research-dies.html?_r=0.Guevara Vasquez, F. (2013). Cracking the genetic code. In ACCESS - cryptography 2013. Obtido em http://www.math.utah.edu/~fguevara/ACCESS2013/Cracking_the_Code.pdf.Nirenberg/Khorana: Breaking the genetic code. (s.d.). Obtido em http://www.mhhe.com/biosci/genbio/raven6b/graphics/raven06b/howscientiststhink/14-lab.pdf.Nirenberg, M. (2004). Historical review: Deciphering the genetic code – a personal account. TRENDS in Biochemical Sciences, 29(1), 46-54. http://dx.doi.org/10.1016/j.tibs.2003.11.009 0.Nirenberg, M. and Leder, P. (1964). RNA codewords and protein synthesis. Science, 145(3639), 1399-1407. http://dx.doi.org/10.1126/science.145.3639.1399.Nirenberg, M. W. and Matthaei, J. H. (1961). The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides. PNAS, 47(10), 1588-1602. http://dx.doi.org/10.1073/pnas.47.10.1588.Office of NIH History. (s.d.). The poly-U experiment. In Deciphering the genetic code: Marshall Nirenberg. Obtido em https://history.nih.gov/exhibits/nirenberg/HS4_polyU.htm.Openstax College, Biology. (29 de setembro de 2015). The genetic code. In OpenStax CNX. Obtido em http://cnx.org/contents/GFy_h8cu@9.87:QEibhJMi@8/The-Genetic-Code.Purves, W. K., Sadava, D. E., Orians, G. H., and Heller, H.C. (2004). The genetic code. In Life: The science of biology (7th ed., pp. 239-241). Sunderland, MA: Sinauer Associates.Raven, P. H., Johnson, G. B., Mason, K. A., Losos, J. B., and Singer, S. R. (2014). The genetic code. In Biology (10th ed., AP ed., pp. 282-284). New York, NY: McGraw-HillReece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). The genetic code. In Campbell biology (10th ed., pp. 337-340). San Francisco, CA: Pearson.Söll, D., Ohtsuka, E., Jones, D. S., Lohrmann, R., Hayatsu, H., Nishimura, S., and Khorana, H. G. (1965). Studies on polynucleotides, XLIX. Stimulation of the binding of aminoacyl-sRNA's to ribosomes by ribotrinucleotides and a survey of codon assignments for 20 amino acids. PNAS, 54(5), 1378-1385. Obtido em http://www.ncbi.nlm.nih.gov/pmc/articles/PMC219908/. Um gene, uma enzimaPontos Principais:
- A hipótese um gene, uma enzima defende a ideia de que cada gene codifica uma única enzima. Atualmente sabe-se que esta ideia é no geral (mas não exatamente) correta.
- Sir Archibald Garrod, um médico inglês, foi o primeiro a sugerir que os genes estavam ligados à enzimas.
- Beadle e Tatum confirmaram a hipótese de Garrod, usando estudos genéticos e bioquímicos do mofo do pão Neurospora.
- Beadle e Tatum identificaram mutantes do mofo do pão que eram incapazes de produzir aminoácidos específicos. Em cada um, uma mutação "quebrou" uma enzima necessária à construção de um determinado aminoácido.
Introdução
Hoje sabemos que um gene típico fornece instruções para construção de uma proteína, que por sua vez ativa determinadas características observáveis de um organismo. Por exemplo, atualmente se sabe que o gene de coloração de flor, descoberta de Gregor Mendel, especifica uma proteína que ajuda na produção de moléculas de pigmento, dando uma coloração roxa às flores quando funciona corretamente.Mendel, no entanto, não sabia que os genes (que ele chamou de "fatores hereditários") especificam proteínas e outras moléculas funcionais. Na verdade, ele nem imaginava como os genes afetavam as características observáveis dos organismos vivos. Quem, então, fez a primeira conexão entre genes e proteínas?Os problemas "inatos do metabolismo" de Garrod"
Muitas vezes vemos casos onde os avanços da biologia básica acontecem no laboratório. Mas, eles também podem acontecer na cabeceira! Sir Archibald Garrod, um médico inglês trabalhando na virada do século 20, foi o primeiro a estabelecer a conexão entre os genes e a bioquímica no corpo humano. Retrato de Sir Archibald Garrod. A fotografia em preto e branco, descreve Garrod, olhando uma pilha de papéis, com óculos na mão._Imagem modificada de "Archibald Edward Garrod." Imagem original por Frederick Gowland Hopkins (CC BY 4.0)._Garrod trabalhou com pacientes que tinham doenças metabólicas e observou que essas doenças, com frequência, eram comuns na família. Ele focou em pacientes com o que chamamos de alcaptonúria. Esta é uma doeça não fatal onde a urina da pessoa tornava-se preta, pois eles não podem quebrar uma molécula chamada alcapton (a qual, em pessoas normais sem a doença, divide-se em outras moléculas incolores).1start superscript, 1, end superscriptObservando a árvore genealógica das pessoas com essa doença, Garrod concluiu que alcaptonuria seguia um padrão de herança recessiva, como alguns das características que Mendel estudou nas suas plantas de ervilhas. Garrod teve a ideia de que os pacientes com alcaptonuria deviam ter um defeito metabólico na quebra do alcapton e que esse defeito poderia ser causado pela forma recessiva de um fator hereditário de Mendel (isto é, um alelo recessivo de um gene).2squaredGarrod referiu-se a isto como um “erro inato do metabolismo” e observou que outras doenças seguiam padrões semelhantes. Ainda que a natureza do gene não estivesse bem compreendida naquela época, por Garrod ou por qualquer outra pessoa, Garrod é atualmente considerado "o pai da genética química" – ou seja, o primeiro a estabelecer a ligação entre os genes e as enzimas que realizam as reações metabólicas.3cubed
Retrato de Sir Archibald Garrod. A fotografia em preto e branco, descreve Garrod, olhando uma pilha de papéis, com óculos na mão._Imagem modificada de "Archibald Edward Garrod." Imagem original por Frederick Gowland Hopkins (CC BY 4.0)._Garrod trabalhou com pacientes que tinham doenças metabólicas e observou que essas doenças, com frequência, eram comuns na família. Ele focou em pacientes com o que chamamos de alcaptonúria. Esta é uma doeça não fatal onde a urina da pessoa tornava-se preta, pois eles não podem quebrar uma molécula chamada alcapton (a qual, em pessoas normais sem a doença, divide-se em outras moléculas incolores).1start superscript, 1, end superscriptObservando a árvore genealógica das pessoas com essa doença, Garrod concluiu que alcaptonuria seguia um padrão de herança recessiva, como alguns das características que Mendel estudou nas suas plantas de ervilhas. Garrod teve a ideia de que os pacientes com alcaptonuria deviam ter um defeito metabólico na quebra do alcapton e que esse defeito poderia ser causado pela forma recessiva de um fator hereditário de Mendel (isto é, um alelo recessivo de um gene).2squaredGarrod referiu-se a isto como um “erro inato do metabolismo” e observou que outras doenças seguiam padrões semelhantes. Ainda que a natureza do gene não estivesse bem compreendida naquela época, por Garrod ou por qualquer outra pessoa, Garrod é atualmente considerado "o pai da genética química" – ou seja, o primeiro a estabelecer a ligação entre os genes e as enzimas que realizam as reações metabólicas.3cubedBeadle e Tatum: conectando os genes com as enzimas
Lamentavelmente as ideias de Garrod passaram despercebidas naquele tempo. Na realidade, foi só depois que dois outros pesquisadores, George Beadle e Edward Tatum, realizaram uma série de experiências inovadoras nos anos 1940 que o trabalho de Garrod foi redescoberto e valorizado.4start superscript, 4, end superscriptBeadle e Tatum trabalharam com um organismo simples: o fungo/mofo comum de pão, ou Neurospora crassa. Utilizando Neurospora, eles foram capazes de mostrar uma clara ligação entre os genes e as enzimas metabólicas.Por que o fungo do pão é ótimo para experiências?
Você deve estar imaginando: por que Beadle e Tatum escolheram algo tão repulsivo (e aparentemente sem importância) para estudar, como o fungo do pão?Bem... em primeiro lugar, Beadle planejou trabalhar com a mosca de fruta Drosophila (também um pouco desagradável, mas um organismo muito mais comum para experimentos naquele tempo). No entanto, conforme ele foi ficando cada vez mais interessado na ligação entre genes e metabolismo, ele percebeu que Neurospora poderia dar a ele uma forma melhor de responder as questões acerca das quais ele tinha interesse. Por um lado, Neurospora tinha um ciclo de vida rápido e conveniente, com fases aploide e diploide que facilitam a realização de experimentos genéticos.5start superscript, 5, end superscript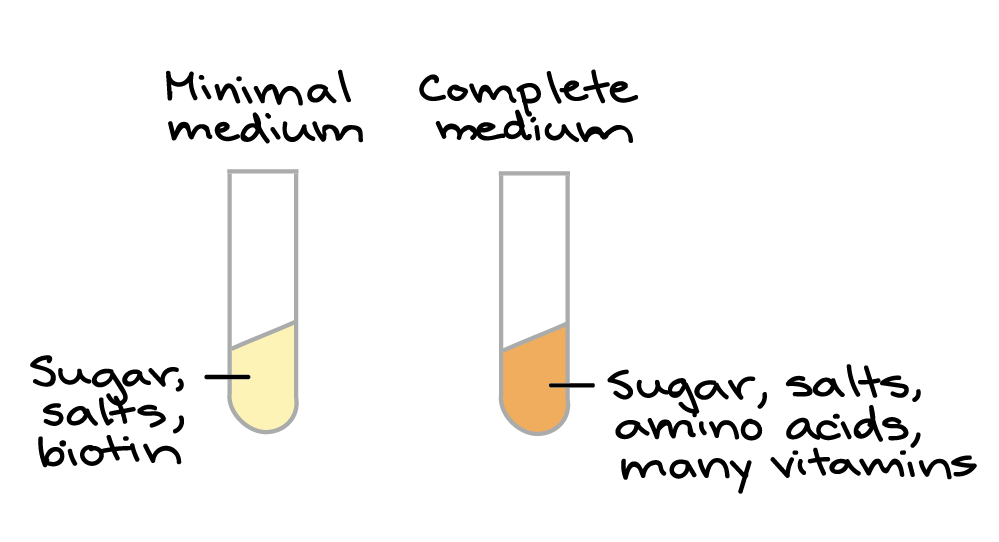 Meio mínimo: continha açúcar, sais e biotinaMeio completo: continha açúcar, sais, aminoácidos e muitas vitaminasTalvez o mais importante é que as células de Neurospora podiam ser cultivadas em laboratório, em meio de cultura simples (caldo ou gel de nutrientes) cuja composição química era 100% conhecida e controlada pelo pesquisador. De fato, as células podiam ser cultivadas em meio mínimo, uma fonte de nutrientes contendo apenas açúcar, sais e uma vitamina (biotina). As células de Neurospora podem sobreviver nesse meio, enquanto muitos outros organismos (como os humanos!) não podem. Isto se deve ao fato de que Neurospora tem vias bioquímicas que transformam açúcar, sais e biotina em todos os outros blocos de construção necessários para as células (tais como aminoácidos e vitaminas).6start superscript, 6, end superscriptAs células de Neurospora também crescem felizes em meio completo, o qual contém um conjunto completo de aminoácidos e vitaminas. Elas apenas não dependem de meio completo para viver.
Meio mínimo: continha açúcar, sais e biotinaMeio completo: continha açúcar, sais, aminoácidos e muitas vitaminasTalvez o mais importante é que as células de Neurospora podiam ser cultivadas em laboratório, em meio de cultura simples (caldo ou gel de nutrientes) cuja composição química era 100% conhecida e controlada pelo pesquisador. De fato, as células podiam ser cultivadas em meio mínimo, uma fonte de nutrientes contendo apenas açúcar, sais e uma vitamina (biotina). As células de Neurospora podem sobreviver nesse meio, enquanto muitos outros organismos (como os humanos!) não podem. Isto se deve ao fato de que Neurospora tem vias bioquímicas que transformam açúcar, sais e biotina em todos os outros blocos de construção necessários para as células (tais como aminoácidos e vitaminas).6start superscript, 6, end superscriptAs células de Neurospora também crescem felizes em meio completo, o qual contém um conjunto completo de aminoácidos e vitaminas. Elas apenas não dependem de meio completo para viver.Vamos fazer alguns mutantes!
Se os genes são ligados com as enzimas bioquímicas, Beadle e Tatum pensaram que seria possível induzir mutações, ou mudanças nos genes, que "quebrem" enzimas específicas (e assim, vias específicas) necessárias para o crescimento num meio de cultura mínimo. Uma linhagem de Neurospora com essa mutação iria crescer normalmente num meio completo, mas perderia sua capacidade de sobreviver num meio de cultura mínimo.7start superscript, 7, end superscript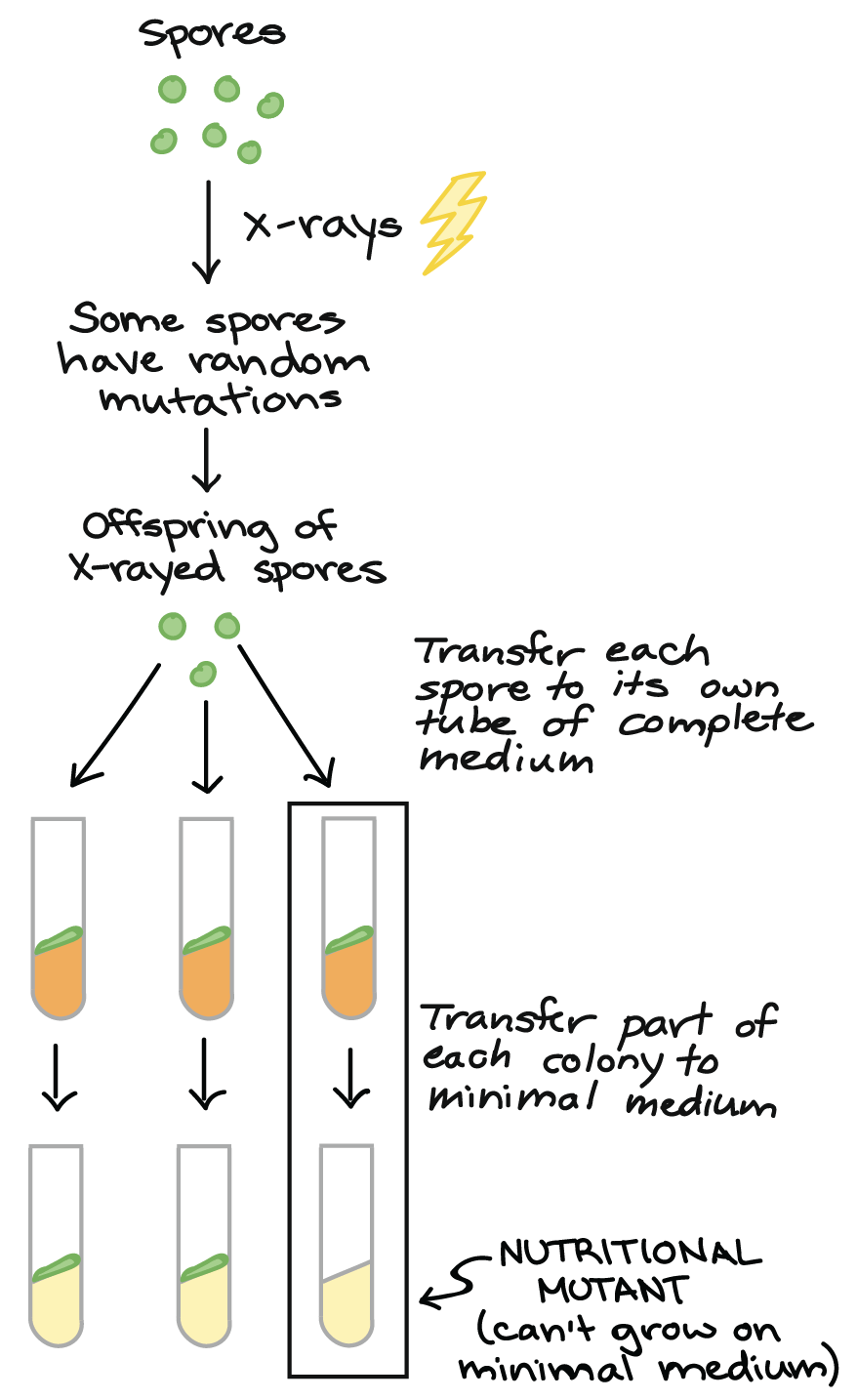
- Obter esporos de Neurospora.
- Expor os esporos aos raios-x. Alguns esporos, agora, têm mutações aleatórias.
- Cruzar os esporos normais (esporos não-irradiados) e coletar os esporos da progênie.
- Transferir cada esporo de progênie individualmente para seu próprio tubo de meio completo, para fazerem uma colônia.
- Transferir parte de cada colônia para seu próprio tubo de meio mínimo.
- Os mutantes nutricionais podem ser identificados como colônias que cresceram no meio completo, mas não cresceram quando transferido para o meio mínimo.
Diagrama baseado no diagrama similar em Griffiths et al. 8start superscript, 8, end superscript.Para procurar mutantes com essas características, Beadle e Tatum expuseram esporos de Neurospora à radiação (Raio X, UV ou nêutrons) para produzir novas mutações. Após alguns poucos passos de limpeza genética, eles pegaram os descendentes dos esporos irradiados e cultivaram-nos individualmente em tubos de ensaio contendo meio completo. Quando cada esporo estabeleceu uma colônia crescente, uma pequena parte da colônia foi transferida para outro tubo contendo meio de cultura mínimo.A maioria das colônias cresceu nos dois meios, completo ou mínimo. No entanto, algumas poucas colônias cresceram normalmente no meio completo. Esses eram os mutantes nutricionais que Beadle e Tatum esperavam encontrar. No meio mínimo, cada mutante iria morrer, pois ele não poderia produzir uma molécula essencial particular além dos nutrientes mínimos. O meio completo iria "resgatar" o mutante (permitir que ele vivesse) provendo a molécula que faltava, junto com uma variedade de outras.9start superscript, 9, end superscriptFocando a via da quebra
Para descobrir qual via metabólica foi "quebrada" em cada mutante, Beadle e Tatum realizaram um experimento inteligente, de duas etapas.Primeiro eles cultivaram cada mutante em meio de cultura mínimo suplementado ou com o conjunto completo de aminoácidos ou com o conjunto completo de vitaminas (ou açúcares, mas não vamos examinar esse caso aqui).8,10start superscript, 8, comma, 10, end superscript- Se um mutante cresce no meio mínimo com aminoácidos (mas sem vitaminas), ele deve ser incapaz de produzir um ou mais aminoácidos.
- Se um mutante cresce no meio de cultura com vitaminas mas não cresce no meio com aminoácidos, ele deve ser incapaz de produzir uma ou mais vitaminas.
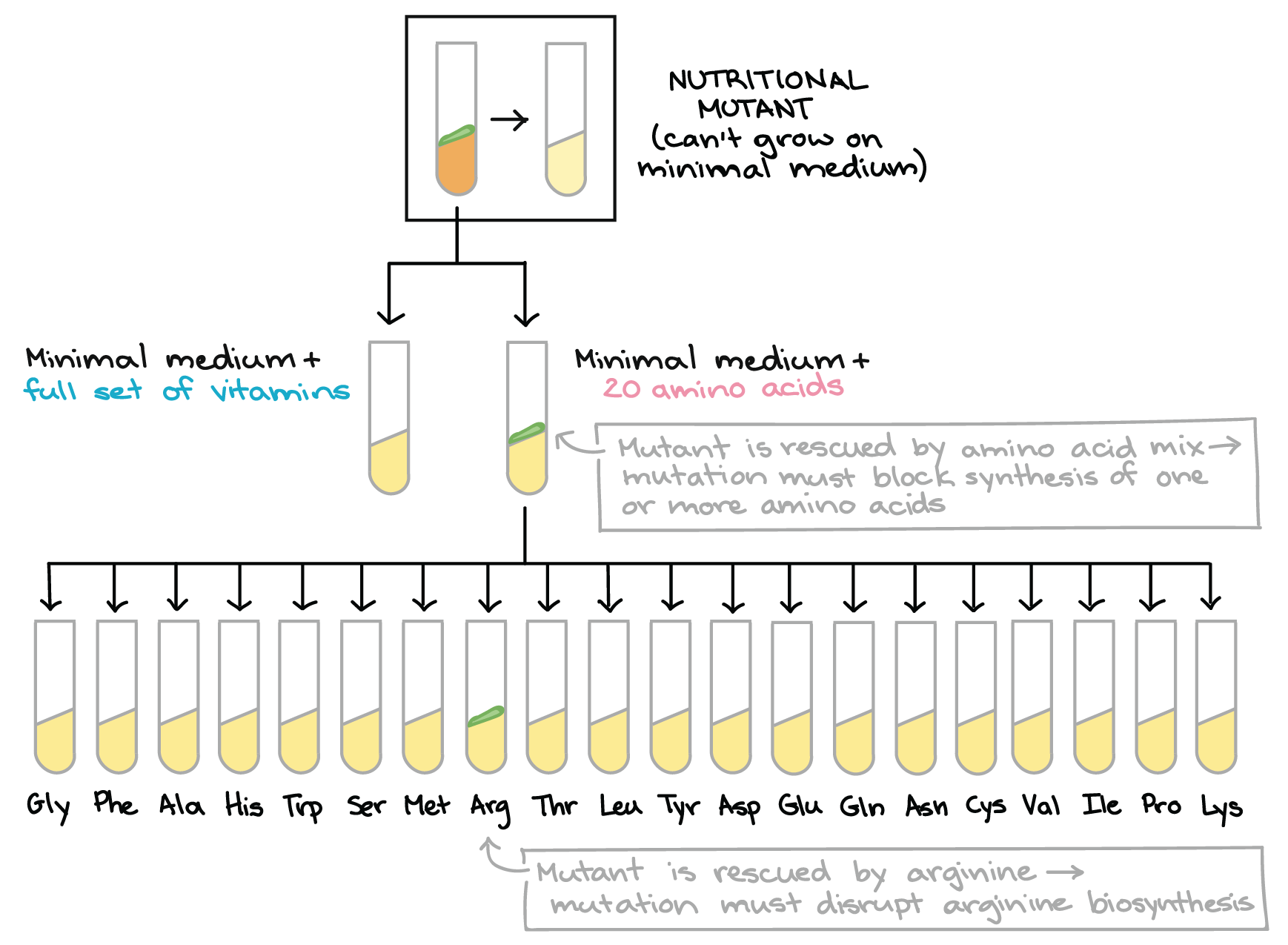
- Inicie com um mutante nutricional. Por definição, o mutante nutricional pode crescer no meio completo, mas não no meio mínimo.
- Agora, vamos descobrir o que no médio completo é o que o mutante nutricional precisava para crescer. Para fazer isso, transferimos um pouco da colônia em cada um dos dois tubos: um com um conjunto de meio mínimo + conjunto completo de vitaminas, outra para um meio mínimo + todos os 20 amino ácidos.
- Neste exemplo, o mutante é salvo pela mistura dos 20 aminoácidos, mas não pelo conjunto das vitaminas. Isto indica que a mutação tornou o mutante incapaz de sintetizar um ou mais aminoácidos
- Uma vez que o mutante é salvo pela mistura dos aminoácidos, a pergunta seguinte é: qual(is) aminoácido(s) ele é incapaz de produzir? Para responder a esta questão transferimos um pouco da colônia mutante para cada um de 20 tubos. Cada tubo contém meio mínimo e mais um dos 20 aminoácidos.
- Neste exemplo, o mutante pode crescer no tubo contendo meio mínimo + arginina, mas não em qualquer um dos outros 19 tubos (isto é, o mutante é salvo pela arginina). Isso indica que a mutação desse mutante interrompe a biossíntese de arginina.
Diagrama baseado no diagrama similar em Griffiths et al. 8start superscript, 8, end superscript.Beadle e Tatum além disso, determinaram o caminho "quebrado" em cada mutante através de uma segunda bateria de testes. Por exemplo, se um mutante cresce no meio mínimo contendo todos os 2020 aminoácidos, eles em seguida testavam em 2020 tubinhos diferentes, cada um contendo meio de cultura mínimo e apenas um dos 2020 aminoácidos. Se o mutante crescesse em algum desses tubinhos, Beadle e Tatum sabiam que o aminoácido desse frasco deveria ser o produto final da via interrompida no mutante.8start superscript, 8, end superscriptDessa forma, Beadle e Tatum ligaram muitos mutantes nutricionais com vias de aminoácidos específicos e a vitaminas biossintéticas. Seu trabalho produziu uma revolução no estudo da genética e mostrou que genes individuais estavam realmente ligados a enzimas específicas.11start superscript, 11, end superscript"Um gene - uma enzima" hoje
O elo descoberto entre genes e enzimas foi inicialmente chamado de hipótese de “um gene - uma enzima”. Esta hipótese sofreu algumas atualizações importantes desde Beadle e Tatum12,13start superscript, 12, comma, 13, end superscript:- Alguns genes codificam proteínas que não são enzimas. Enzimas são apenas uma categoria de proteína. Nas células há muitas proteínas que não são enzimas e essas proteínas também são codificadas pelos genes.
- Alguns genes codificam uma subunidade de proteína, não uma proteína inteira. Em geral, um gene codifica um polipeptídeo, isto é, uma cadeia de aminoácidos. Algumas proteínas consistem de vários polipeptídeos de diferentes genes.
- Alguns genes não codificam polipeptídeos. Alguns genes realmente codificam moléculas de RNA funcional ao invés de polipeptídeos!
Embora o conceito de "um gene-uma enzima" não seja exato, sua ideia central - de que um gene normalmente especifica uma proteína numa relação um para um - permanece útil aos geneticistas até hoje. Este artigo está autorizado sob licença CC BY-NC-SA 4.0.Referências:
- Piro, A. Tagarelli, G., Lagonia, P., Quattrone, A., and Tagarelli, A. (2010). Archibald Edward Garrod and alcaptonuria: “Inborn errors of metabolism” revisited. Genetics in Medicine, 12, 475. http://www.ncbi.nlm.nih.gov/pubmed/20703138.
- Garrod, A. E. (1902). The incidence of alkaptonuria: A study in chemical individuality. Lancet, 2, 1616-1620. Disponível em http://www.esp.org/foundations/genetics/classical/ag-02.pdf.
- Genome News Network. (2004). 1908: Archibald E. Garrod (1857-1936) postulates that genetic defects cause many inherited diseases. In Genetics and genomics timeline. Disponível em http://www.genomenewsnetwork.org/resources/timeline/1908_Garrod.php.
- Brief forward to ESP reprint of: Garrod, A. E. (1902). The incidence of alkaptonuria: A study in chemical individuality. Lancet, 2, 1616-1620. Disponível em http://www.esp.org/foundations/genetics/classical/ag-02.pdf.
- Nasrallah, J. B. (2012). Adrian M. Srb. In Biographical memoirs, 5. Disponível em http://www.nasonline.org/publications/biographical-memoirs/memoir-pdfs/srb-adrian.pdf
- Beadle, G. W. and Tatum, E. L. (1941). Genetic control of biochemical reactions in Neurospora. PNAS, 27, 502. Disponível em: http://www.pnas.org/content/27/11/499.full.pdf.
- Beadle, G. W. and Tatum, E. L. (1941). Genetic control of biochemical reactions in Neurospora. PNAS, 27, 500. Disponível em: http://www.pnas.org/content/27/11/499.full.pdf.
- Griffiths, A. J. F., Miller, J. H., Suzuki, D. T., Lewontin, R. C., and Gelbart, W. M. (2000). The procedure used by Beadle and Tatum. In An introduction to genetic analysis (7th ed.). New York, NY: W. H. Freeman. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK22044/figure/A1616/?report=objectonly.
- Beadle, G. W. and Tatum, E. L. (1945). Neurospora. II. Methods of producing and detecting mutations concerned with nutritional requirements. American Journal of Botany, 32, 679-680. Disponível em: http://www.jstor.org/stable/2437625.
- Beadle, G. W. and Tatum, E. L. (1945). Neurospora. II. Methods of producing and detecting mutations concerned with nutritional requirements. American Journal of Botany, 32, 681. Disponível em: http://www.jstor.org/stable/2437625.
- Horowitz, N. H., Berg, P., Singer, M., Lederberg, J., Susman, M., Doebley, J., and Crow, J.F. (2004). A Centennial: George W. Beadle, 1903–1989. In J. F. Crow and W. F. Dove (eds.), Perspectives: Anecdotal, historical and critical commentaries on genetics. Genetics, 166(1), 2. http://dx.doi.org/10.1534/genetics.166.1.1.
- Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). The products of gene expression: A developing story. In Campbell biology (10th ed., p. 336). San Francisco, CA: Pearson.
- Purves, W. K., Sadava, D. E., Orians, G. H., and Heller, H.C. (2004). One gene, one polypeptide. In Life: the science of biology (7th ed., p. 234). Sunderland, MA: Sinauer Associates.
Outras referências
Beadle, G. W. and Tatum, E. L. (1941). Genetic control of biochemical reactions in Neurospora. PNAS, 27, 499-506. Disponível em: http://www.pnas.org/content/27/11/499.full.pdf.Beadle, G. W. and Tatum, E. L. (1945). Neurospora. II. Methods of producing and detecting mutations concerned with nutritional requirements. American Journal of Botany, 32, 678-686. Disponível em: http://www.jstor.org/stable/2437625.Experimental basis for the one-gene, one-protein hypothesis. (2010). In Hospital centennial: The Rockefeller University hospital. Disponível em: http://centennial.rucares.org/index.php?page=One-gene_One-enzyme.Garrod, A. E. (1902). The incidence of alkaptonuria: A study in chemical individuality. Lancet, 2, 1616-1620. Disponível em: http://www.esp.org/foundations/genetics/classical/ag-02.pdf.Genome News Network. (2004). 1941: George W. Beadle (1903-1989) and Edward L. Tatum (1909-1975) show how genes direct the synthesis of enzymes that control metabolic processes. In Genetics and genomics timeline. Disponível em: http://www.genomenewsnetwork.org/resources/timeline/1941_Beadle_Tatum.php.George Wells Beadle (1903-1989). (2011). In DNA from the beginning. Disponível em: http://www.dnaftb.org/16/bio.html.Griffiths, A. J. F., Miller, J. H., Suzuki, D. T., Lewontin, R. C., and Gelbart, W. M. (2000). How genes work. In An introduction to genetic analysis (7th ed.). New York, NY: W. H. Freeman. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK22044/.Griffiths, A. J. F., Wessler, S. R., Lewontin, R. C., and Carroll, S. B. (2008). Neurospora. In Introduction to genetic analysis (10th ed., p. 107). New York, NY: W. H. Freeman and Company.Horowitz, N. H., Berg, P., Singer, M., Lederberg, J., Susman, M., Doebley, J., and Crow, J.F. (2004). A Centennial: George W. Beadle, 1903–1989. In J. F. Crow and W. F. Dove (eds.), Perspectives: Anecdotal, historical and critical commentaries on genetics. Genetics, 166(1), 1-10. http://dx.doi.org/10.1534/genetics.166.1.1.Nasrallah, J. B. (2012). Adrian M. Srb. In Biographical memoirs. Disponível em: http://www.nasonline.org/publications/biographical-memoirs/memoir-pdfs/srb-adrian.pdf.Neurospora life cycle. (2004, August 4). In Fungal genetics stock center. Disponível em: http://www.fgsc.net/neurospora/sectionb2.htm.One gene makes one protein. (2011). In DNA from the beginning. Disponível em: http://www.dnaftb.org/16/.Neurospora life cycle. (2004, August 4). In Fungal genetics stock center. Retrieved from http://www.fgsc.net/neurospora/sectionb2.htm.One gene makes one protein. (2011). In DNA from the beginning. Disponível em: http://www.dnaftb.org/16/.Piro, A. Tagarelli, G., Lagonia, P., Quattrone, A., and Tagarelli, A. (2010). Archibald Edward Garrod and alcaptonuria: “Inborn errors of metabolism” revisited. Genetics in Medicine, 12, 475-476. http://www.ncbi.nlm.nih.gov/pubmed/20703138.Raven, P. H., Johnson, G. B., Mason, K. A., Losos, J. B., and Singer, S. R. (2014). The nature of genes. In Biology (10th ed., AP ed., pp. 278-281). New York, NY: McGraw-Hill.Raven, P. H., and Johnson, G. B. (2002). The one gene/one polypeptide hypothesis. In Biology (6th ed., pp. 295-296). Boston, MA: McGraw-Hill.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Genes specify proteins via transcription and translation. In Campbell biology (10th ed., pp. 92-123). San Francisco, CA: Pearson.Srb, A. M. and Horowitz, N. H. (1944). The ornithine cycle in Neurospora and its genetic control. Journal of Biological Chemistry, 154, 129-139. Disponível em: http://www.jbc.org/content/154/1/129.full.pdf+html. Ácidos nucleicosIntrodução Ácidos nucleicos, e DNA em particular, são macromoléculas chave para a continuidade da vida. O DNA carrega a informação hereditária que é passada de pais para filhos, fornecendo instruções de como (e quando) fazer as muitas proteínas necessárias para construir e manter o funcionamento das células, tecidos, e organismos.Como o DNA carrega essa informação, e como ela é colocada em ação pelas células e organismos, é complexo, fascinante, e muito alucinante, e vamos explorar isso em mais detalhes na seção biologia molecular. Aqui, vamos apenas olhar rapidamente os ácidos nucleicos da perspectiva macromolecular.Papéis do DNA e RNA nas células
Ácidos nucleicos, macromoléculas feitas de unidade chamadas nucleotídeos, vêm em duas variedades naturais: ácido desoxirribonucleico (DNA) e ácido ribonucleico (RNA). O DNA é o material genético encontrado em seres vivos, desde bactérias unicelulares até mamíferos multicelulares como você e eu. Alguns virus usam RNA, não DNA, como seu material genético, mas não são tecnicamente considerados vivos (já que não podem reproduzir sem ajuda de um hospedeiro).DNA nas células
Em eucariontes, como plantas e animais, o DNA é encontrado no núcleo, um cofre especializado protegido por uma membrana, assim como em outros tipos de organelas (como as mitocôndrias e os cloroplastos das plantas). Nos procariontes, como as bactérias, o DNA não está em um envelope de membrana, apesar de estar localizado em uma região celular especializada chamada de nucleoide.Nos eucariontes, o DNA é tipicamente dividido em um número de longos pedaços lineares chamados cromossomos, enquanto que nos procariontes como bactérias, os cromossomos são muito menores e geralmente circulares (em forma de anel). Um cromossomo pode conter dezenas de milhares de genes, cada um provendo instruções de como fazer um produto específico que a célula precisa.Do DNA para RNA, do RNA para proteínas
Muitos genes codificam produtos proteicos, isto é, especificam a sequência de aminoácidos utilizada para construir uma proteína em particular. Antes que essa informação possa ser utilizada para a síntese de proteínas, no entanto, uma cópia de RNA (resultante da transcrição) do gene deve ser feita em primeiro lugar. Esse tipo de RNA é chamado de RNA mensageiro (RNAm), por servir como mensageiro entre o DNA e os ribossomos, máquinas moleculares que leem as sequências de RNAm e as utilizam para construir proteínas. Essa progressão de DNA para RNA para proteína é chamada de "dogma central" da biologia molecular.É importante observar que nem todos os genes codificam produtos proteicos. Por exemplo, alguns genes especificam RNAs ribossômicos (RNAr), que servem como componentes estruturais de ribossomos, ou RNAs transportadores (RNAt), moléculas de RNA em forma de trevo que trazem aminoácidos aos ribossomos para a síntese proteica. Ainda outras moléculas de RNA, como pequenos microRNAs (miRNA), agem como reguladores de outros genes, e novos tipos de RNAs não-codificadores de proteínas estão sendo descobertos o tempo todo.Nucleotídeos
DNA e RNA são polímeros (no caso do DNA, geralmente polímeros muito longos), e são feitos de monômeros conhecidos como nucleotídeos. Quando esses monômeros se combinam, a cadeia resultante é chamada de polinucleotídeo (poli- = "muitos").Cada nucleotídeo é feito de três partes: uma estrutura anelar contendo nitrogênio chamada de base nitrogenada, um açúcar de cinco carbonos, e pelo menos um grupo fosfato. A molécula de açúcar tem uma posição central no nucleotídeo, com a base ligada a um de seus carbonos e o grupo (ou grupos) fosfato ligado a outro. Vejamos cada parte de um nucleotídeo por vez.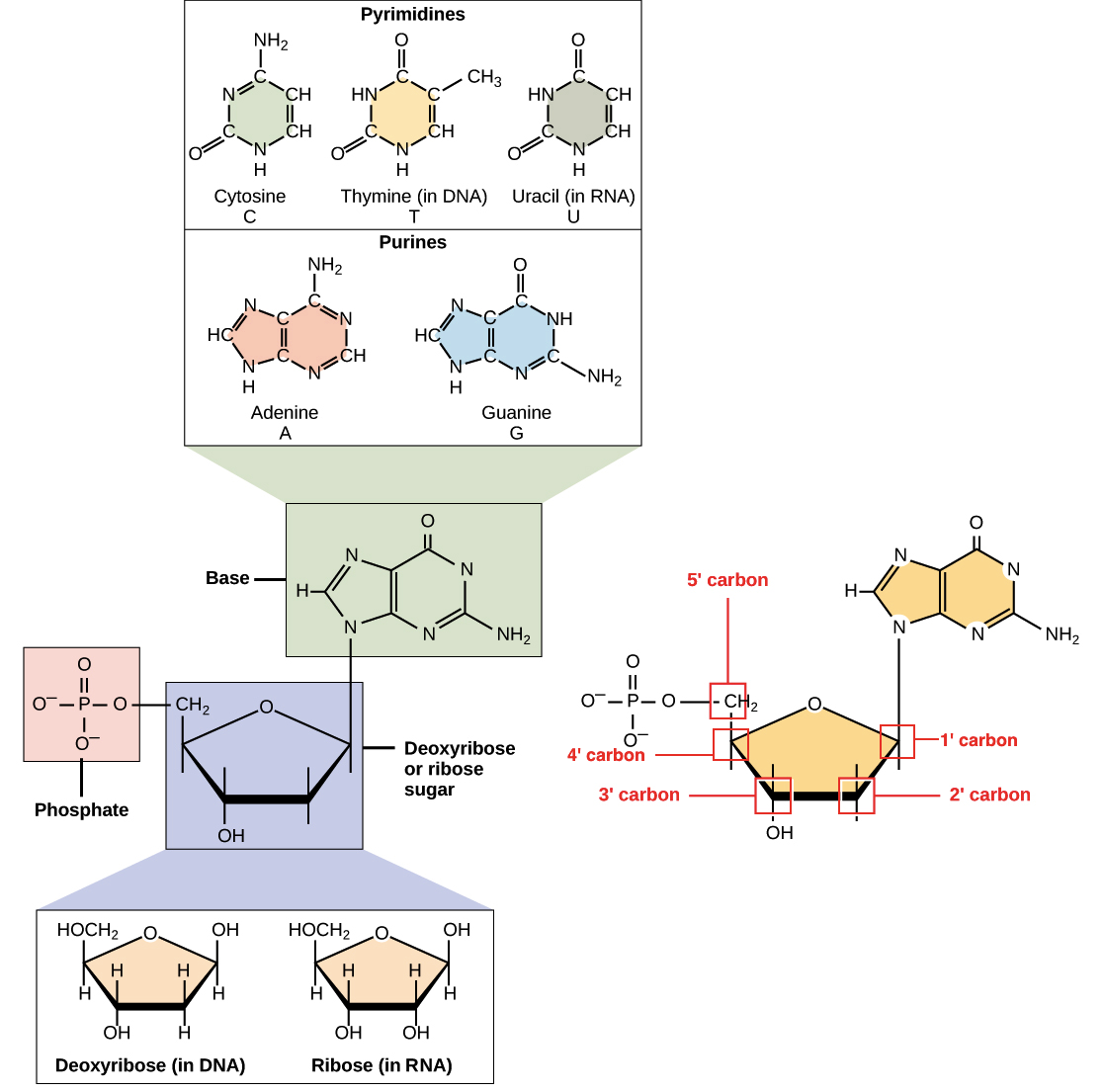 Imagem dos componentes de DNA e RNA, incluindo o açúcar (desoxirribose ou ribose), o grupo fosfato e a base nitrogenada. As bases são bases pirimidinas (citosina, timina no DNA e uracila no RNA, um anel) e as bases purinas (adenina e guanina, dois anéis). O grupo fosfato está ligado ao carbono 5'. O carbono 2' liga-se a um grupo hidroxila na ribose, mas nenhuma hidroxila (apenas hidrogênio) na desoxirribose._Imagem modificada de "Nucleic acids: Figure 1," por OpenStax College, Biology (CC BY 3.0)._
Imagem dos componentes de DNA e RNA, incluindo o açúcar (desoxirribose ou ribose), o grupo fosfato e a base nitrogenada. As bases são bases pirimidinas (citosina, timina no DNA e uracila no RNA, um anel) e as bases purinas (adenina e guanina, dois anéis). O grupo fosfato está ligado ao carbono 5'. O carbono 2' liga-se a um grupo hidroxila na ribose, mas nenhuma hidroxila (apenas hidrogênio) na desoxirribose._Imagem modificada de "Nucleic acids: Figure 1," por OpenStax College, Biology (CC BY 3.0)._Bases nitrogenadas
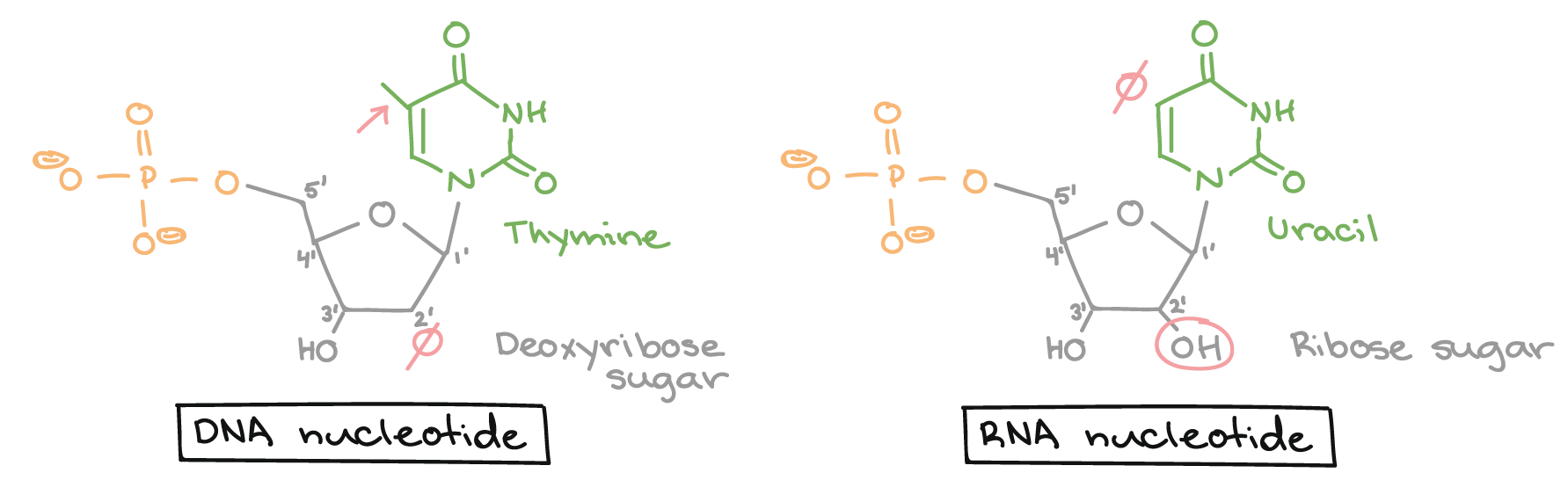
As bases nitrogenadas de nucleotídeos são moléculas orgânicas (com base de carbono) feitas de estruturas anelares contendo nitrogênio.Cada nucleotídeo no DNA contém uma de quatro possíveis bases nitrogenadas: adenina (A), guanina (G), citosina(C), e timina (T). Adenina e guanina são purinas, o que significa que suas estruturas contém dois anéis de carbono-nitrogênio unidos. Citosina e timina, em contraste, são pirimidinas e têm um único anel de carbono-nitrogênio. Os nucleotídeos de RNA também podem apresentar as bases adenina, guanina e citosina., mas em vez de timina eles têm outra base pirimidina chamada uracila (U). Como mostrado na figura acima, cada base tem uma estrutura única, com seu próprio conjunto de grupos funcionais ligados à estrutura anelar.Em biologia molecular abreviada, as bases nitrogenadas geralmente são mencionadas por suas letras, A, T, G, C e U. O DNA contém A, T, G e C, enquanto o RNA contém A, U, G e C (isto é, o U é colocado no lugar do T).Açúcares
Além de terem conjuntos de bases ligeiramente diferentes, os nucleotídeos de DNA e RNA tem açúcares ligeiramente diferentes. O açúcar de cinco carbonos no DNA é chamado de desoxirribose, enquanto que no RNA, o açúcar é ribose. Esses dois são muito similares na estrutura, com apenas uma diferença: o segundo carbono da ribose liga-se a um grupo hidroxila, enquanto o carbono equivalente da desoxirribose tem um hidrogênio. Os átomos de carbono de uma molécula de açúcar de nucleotídeo são numerados como 1', 2', 3', 4', e 5' (1' é lido como "um linha"), como mostrado na figura acima. Num nucleotídeo, o açúcar ocupa uma posição central, com a base ligada a seu carbono 1' e o grupo (ou grupos) fosfato ligado(s) ao carbono 5'.Fosfato Os nucleotídeos podem ter um único grupo fosfato, ou uma cadeia de até três grupos fosfato, ligados ao carbono 5' do açúcar. Algumas fontes, em química, utilizam o termo "nucleotídeo" apenas para o caso de fosfato único, mas na biologia molecular, a definição mais ampla é geralmente aceita1.
start superscript, 1, end superscriptEm uma célula, um nucleotídeo prestes a ser adicionado ao final de uma cadeia de polinucleotídeos estará ligado a uma série de três grupos fosfato. Quando o nucleotídeo se junta a cadeia crescente de DNA ou RNA, perde dois grupos fosfato. Portanto, em uma cadeia de DNA ou RNA, cada nucleotídeo tem apenas um grupo fosfato.Cadeias de polinucleotídeos
Uma consequência da estrutura de nucleotídeos é que uma cadeia de polinucleotídeos tem direcionalidade, ou seja, ela tem duas extremidades diferentes uma da outra. Na extremidade 5', ou início, da cadeia, o grupo fosfato 5' do primeiro nucleotídeo da cadeia se sobressai. Na outra extremidade, chamada de extremidade 3', a hidroxila 3' do último nucleotídeo adicionado à cadeia é exposta. Em geral, sequências de DNA são escritas na direção 5' para 3', o que significa que o nucleotídeo na extremidade 5' vem primeiro e que o nucleotídeo na extremidade 3' vem por último.Quando nucleotídeos são adicionados a uma fita de DNA ou RNA, a fita cresce em seu final 3', com o fosfato 5' de um nucleotídeo que entra se ligando ao grupo hidroxila no final 3' da cadeia. Isso faz uma cadeia com cada açúcar unido a seus vizinhos por uma série de ligações chamadas de ligação fosfodiéster.Propriedades do DNA
As cadeias de ácido desoxirribonucleico, ou DNA, são tipicamente encontradas em uma dupla hélice, uma estrutura na qual duas cadeias correspondentes (complementares) estão ligadas, como mostrado no diagrama à esquerda. Os açúcares e fosfatos localizam-se na parte externa da hélice, formando o arcabouço do DNA; esta porçao da molécula é algumas vezes chamada de esqueleto de açúcar-fosfato. As bases nitrogenadas se estendem para o interior, como os degraus de uma escada, em pares; as bases de um par se unem entre si por ligações de hidrogênio.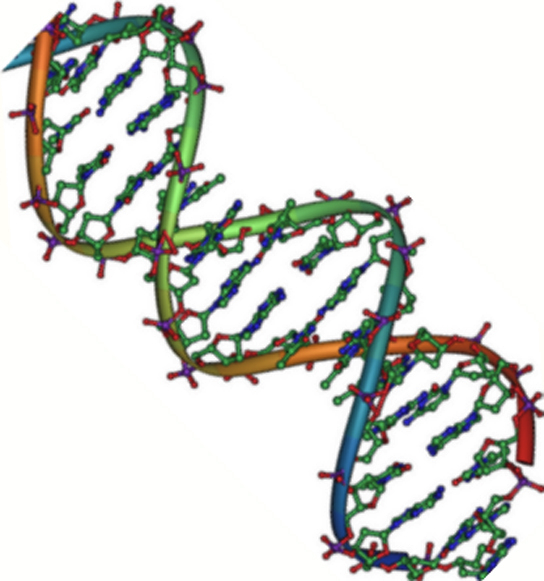 Modelo estrutural da dupla hélice de DNA.Crédito da imagem: Jerome Walker/Dennis Myts.As duas fitas da hélice vão em direções opostas, isto é, o final 5' de uma fita é pareado como final 3' de sua fita correspondente. (Nos referimos a isto como orientação antiparalela e é importante ao copiar o DNA.)Então, podem duas bases decidirem se juntar e formar um par na dupla hélice? A resposta é definitivamente não. Por conta dos tamanhos e grupos funcionais das bases, o pareamento de bases é muito específico: A pode apenas fazer par com T, e G pode apenas fazer par com C, como mostrado abaixo. Isso significa que as duas fitas da dupla hélice de DNA tem uma relação bem previsível uma com a outra.Por exemplo, se você sabe que a sequência de uma fita é 5' -AATTGGCC-3’, a fita complementar deve ter a sequência 3’-TTAACCGG-5’. Isso permite que cada base se combine com sua parceira:
Modelo estrutural da dupla hélice de DNA.Crédito da imagem: Jerome Walker/Dennis Myts.As duas fitas da hélice vão em direções opostas, isto é, o final 5' de uma fita é pareado como final 3' de sua fita correspondente. (Nos referimos a isto como orientação antiparalela e é importante ao copiar o DNA.)Então, podem duas bases decidirem se juntar e formar um par na dupla hélice? A resposta é definitivamente não. Por conta dos tamanhos e grupos funcionais das bases, o pareamento de bases é muito específico: A pode apenas fazer par com T, e G pode apenas fazer par com C, como mostrado abaixo. Isso significa que as duas fitas da dupla hélice de DNA tem uma relação bem previsível uma com a outra.Por exemplo, se você sabe que a sequência de uma fita é 5' -AATTGGCC-3’, a fita complementar deve ter a sequência 3’-TTAACCGG-5’. Isso permite que cada base se combine com sua parceira: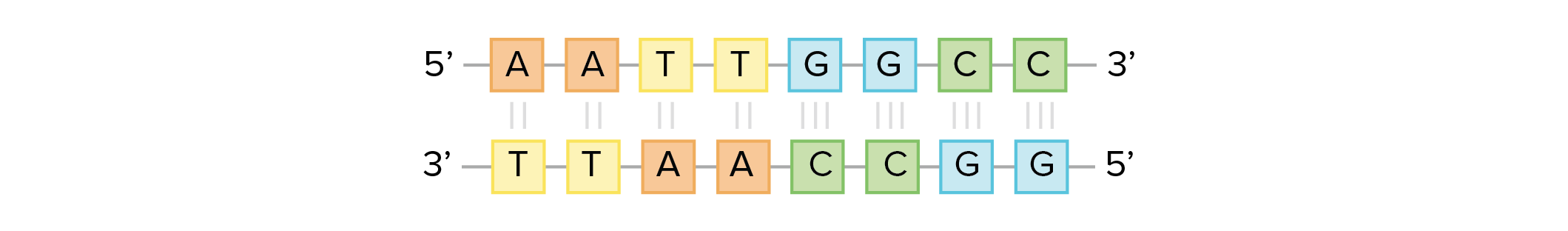 5'-AATTGGCC-3' 3'-TTAACCGG -5'Estas duas fitas são complementares, uma ligada na outra pelas bases pareadas. Os pares A-T estão ligados por duas ligações de hidrogênio, enquanto os pares G-C são conectados por três ligações de hidrogênio.Quando duas sequências de DNA se combinam desse jeito, possibilitando a ligação entre si de modo antiparalelo e formando uma hélice, elas são consideradas complementares.
5'-AATTGGCC-3' 3'-TTAACCGG -5'Estas duas fitas são complementares, uma ligada na outra pelas bases pareadas. Os pares A-T estão ligados por duas ligações de hidrogênio, enquanto os pares G-C são conectados por três ligações de hidrogênio.Quando duas sequências de DNA se combinam desse jeito, possibilitando a ligação entre si de modo antiparalelo e formando uma hélice, elas são consideradas complementares.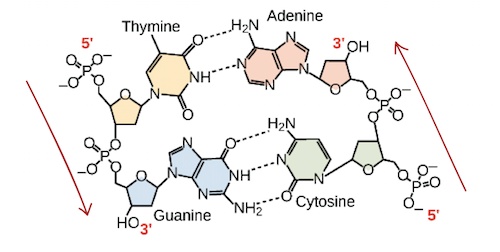 Ligação de hidrogênio entre bases complementares junta as fitas de DNA em uma hélice dupla de fitas antiparalelas. A timina forma duas ligações de hidrogênio com adenina, e guanina forma ligações de três hidrogênios com a citosina.Imagem adaptada de OpenStax Biology.
Ligação de hidrogênio entre bases complementares junta as fitas de DNA em uma hélice dupla de fitas antiparalelas. A timina forma duas ligações de hidrogênio com adenina, e guanina forma ligações de três hidrogênios com a citosina.Imagem adaptada de OpenStax Biology.Propriedades do RNA
O ácido ribonucleico (RNA), diferente do DNA, é geralmente de fita única. Um nucleotídeo em uma cadeia de RNA conterá ribose (o açúcar de cinco carbonos), uma das quatro bases nitrogenadas (A, U, G ou C), e um grupo fosfato. Aqui, olharemos os quatro tipos principais de RNA: RNA mensageiro (RNAm), RNA ribossômico (RNAr), RNA transportador (RNAt) e RNAs reguladores.RNA mensageiro (RNAm)
RNA mensageiro (RNAm) é um intermediário entre um gene codificador de proteína e seu produto proteico. Se uma célula precisa fazer uma proteína em especial, o gene codificador da proteína será "ligado", isto é, uma enzima RNA polimerase virá e fará uma cópia de RNA, ou transcrição da sequência de DNA do gene. A cópia carrega a mesma informação da sequência de DNA de seu gene. No entanto, na molécula de RNA, a base T é substituída por U. Por exemplo, se uma fita de DNA codificadora tem a sequência 5’-AATTGCGC-3’, a sequência do RNA correspondente será 3’-UUAACGCG-5’.Uma vez que um RNAm é produzido, será associado a um ribossomo, uma máquina molecular que é especializada em montar proteínas a partir de aminoácidos. O ribossomo usa a informação no RNAm para fazer uma proteína de uma sequência específica, "lendo" os nucleotídeos de RNAm em grupos de três (chamados códons) e adicionando um aminoácido particular a cada códon.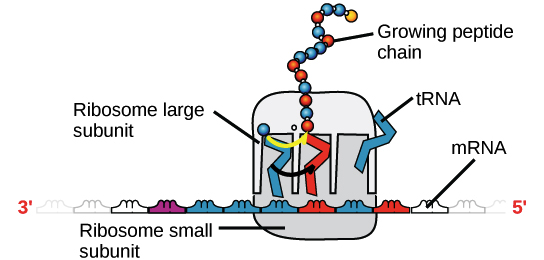 Imagem de um ribossomo (feito de proteínas e RNAr) ligado a um RNAm, com RNAt trazendo aminoácidos para seres adicionados à cadeia crescente. O RNAt que se liga, e consequentemente, o aminoácido que é acrescentado, em um determinado momento é determinado pela sequência do RNAm que está sendo "lido" naquele momento.Figura: OpenStax Biology.
Imagem de um ribossomo (feito de proteínas e RNAr) ligado a um RNAm, com RNAt trazendo aminoácidos para seres adicionados à cadeia crescente. O RNAt que se liga, e consequentemente, o aminoácido que é acrescentado, em um determinado momento é determinado pela sequência do RNAm que está sendo "lido" naquele momento.Figura: OpenStax Biology.RNA ribossômico (RNAr) e RNA transportador (RNAt)
O RNA ribossômico (RNAr) é um componente importante dos ribossomos, ajudando o mRNA a se ligar no local certo para que a sequência de informações possa ser lida. Alguns RNAr também atuam como enzimas, o que significa que eles ajudam a acelerar (catalisar) reações químicas – neste caso, a formação de ligações que unem os aminoácidos para formar uma proteína. Os RNAs que atuam como enzimas são conhecidos como ribozimas.RNAs transportadores (RNAt) também estão envolvidos na síntese proteica, mas seu trabalho é agir como carregadores - trazer aminoácidos ao ribossomo, assegurando que o aminoácido adicionado a cadeia é o especificado pelo RNAm. RNAs transportadores consistem de uma fita única de RNA, mas essa fita tem segmentos complementares que ficam juntos para fazer regiões de fita dupla. Esse pareamento de bases cria uma estrutura 3D complexa importante à função da molécula.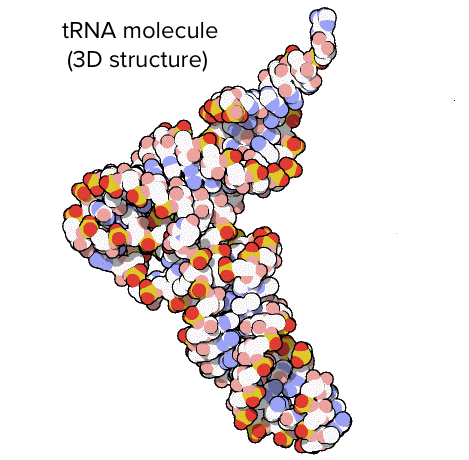 Estrutura de um RNAt. A molécula geral tem uma forma mais ou menos parecida com um L.Imagem adaptada de Protein Data Bank (trabalho do governo dos EUA).
Estrutura de um RNAt. A molécula geral tem uma forma mais ou menos parecida com um L.Imagem adaptada de Protein Data Bank (trabalho do governo dos EUA).RNA regulatório (miRNAs e siRNAs)
Alguns tipos de RNAs não codificadores (RNAs que não codificam proteínas) ajudam a regular a expressão de outros genes. Esses RNAs podem ser chamados de RNAs regulatórios. Por exemplo, microRNAs (miRNAs) e RNAs de pequena interferência siRNA são pequenas moléculas de RNA regulatório de 22 nucleotídeos de extensão. Elas se ligam a moléculas específicas de RNAm (com sequências parcial ou completamente complementares) e reduzem sua estabilidade ou interferem em sua tradução, fornecendo uma maneira de a célula diminuir ou ajustar níveis desses RNAm.Estes são apenas alguns exemplos de vários tipos de RNAs regulatórios e não codificadores. Cientistas ainda estão descobrindo novas variedades de RNA não codificador.Resumo: características do DNA e RNA
DNA RNA Função Repositório de informação genética Envolvido na síntese protéica e regulação gênica; portador da informação genética em alguns vírus Açúcar Desoxirribose Ribose Estrutura Dupla hélice Geralmente fita simples Bases C, T, A, G C, U, A, G Tabela adaptada de OpenStax Biology.Créditos:
Este artigo foi adaptado de “Eukaryotic cells,” de OpenStax College, Biology (CC BY 3.0). Baixe o artigo original de graça em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@9.85:18/Biology.O artigo adaptado está autorizado sob a licença CC BY-NC-SA 4.0Referências:
- Nucleotídeo. In: Wikipédia: a enciclopédia livre. Disponível em: https://en.wikipedia.org/wiki/Nucleotide. Acesso em: 23 jul 2015.
- Soifer, H. S., Rossi, J. J., and Sætrom, P. (2007). MicroRNAs in disease and potential therapeutic applications. Molecular Therapy, 15(12), 2070-2079. http://dx.doi.org/10.1038/sj.mt.6300311.
Outras referências
Biomolecules: DNA 1. (n.d.). Em Biomolecules Tutorial. Disponível em: https://www.chem.wisc.edu/deptfiles/genchem/netorial/modules/biomolecules/modules/dna1/dna13.htm.Cao, J. (2014). The functional role of long non-coding RNAs and epigenetics. Biol. Proced. Online, 16, 11. http://dx.doi.org/10.1186/1480-9222-16-11.Giraldez, A. (n.d.) MicroRNAs. Em Giraldez lab. Retrieved de http://www.giraldezlab.org/miRNA.htmlMore functional RNA molecules. (n.d.). In Genetics. Disponível em: http://bio.sunyorange.edu/updated2/GENETICS/10%20snRNA.htm.Mourão, A., Varrot, A., Mackereth, C. D., Cusack, S., and Sattler, M. (2010). Structure and RNA recognition by the snRNA and snoRNA transport factor PHAX. RNA, 16(6), 1205-1216. http://dx.doi.org/10.1261/rna.2009910.Novina, C. D. and Sharp, P. A. (2004). The RNAi revolution. Nature, 430, 161-164. http://dx.doi.org/10.1038/430161a. Disponível em: http://faculty.buffalostate.edu/wadswogj/courses/450/RNAi%20Sharp%20Review.pdf.Nucleic acid. (22 de junho de 2015). Acesso em: 24 de julho de 2015. Disponível em Wikipedia: https://en.wikipedia.org/wiki/Nucleic_acid.Nucleotide. (23 de julho de 2015). Acesso em: 24 de julho de 2015. Disponível em Wikipedia: https://en.wikipedia.org/wiki/Nucleotide.Phosphodiester bond. (26 de junho de 2015). Acesso em: 24 de julho de 2015. Disponível em: https://en.wikipedia.org/wiki/Phosphodiester_bond.Raven, P. H., Johnson, G. B., Mason, K. A., Losos, J. B., and Singer, S. R. (2014). Nucleic acids: Information molecules. In Biology (10th ed., AP ed., pp. 41-44). New York, NY: McGraw-Hill.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Nucleic acids store, transmit, and help express hereditary information. In Campbell biology (10th ed., pp. 84-87). San Francisco, CA: Pearson.Weick, E.-M. and Miska, E. A. (2014). piRNAs: From biogenesis to function. Development, 141, 3458-3471. http://dx.doi.org/10.1242/dev.094037.What's the difference between siRNA and microRNA (miRNA)? (2013, January 9). In ResearchGate. Disponível em: https://www.researchgate.net/post/whats_the_difference_between_siRNA_and_microRNA_miRNA Resumo da transcriçãoPontos Principais:
- Transcrição é a primeira etapa da expressão do gene. Envolve a cópia da sequência de DNA de um gene para produzir uma molécula de RNA
- A transcrição é realizada por enzimas chamadas RNA polimerases, que ligam nucleotídeos para produzir uma cadeia de RNA (usando uma cadeia de DNA como modelo).
- A transcrição tem três estágios: iniciação, alongamento e término.
- Nos eucariontes, as moléculas de RNA devem ser processadas após a transcrição: elas são emendadas e têm um cap 5' e uma cauda poli A colocadas em suas extremidades.
- A transcrição é controlada separadamente para cada gene em seu genoma.
Introdução
- Você alguma vez já teve que transcrever algo? Talvez alguém tenha deixado uma mensagem em seu e-mail de voz e você teve que anotá-la num papel. Ou pode ser que você tenha feito anotações na aula e então as tenha reescrito ordenadamente para ajudá-lo na revisão.Como esses exemplos mostram, transcrição é um processo em que a informação é reescrita. Transcrição é algo que fazemos em nosso cotidiano, e também é uma coisa que nossas células precisam fazer, mas de uma forma mais especializada e bem definida. Em biologia, transcrição é o processo de cópia da seqüência do DNA de um gene para um alfabeto semelhante do RNA.
Resumo da transcrição
- Transcrição é o primeiro passo na expressão gênica, no qual as informações de um gene são usadas para construir um produto funcional, como uma proteína. O objetivo da transcrição é fazer uma cópia de RNA a partir da sequência de DNA de um gene. Para um gene codificador de proteína, a cópia de RNA, ou transcrito, carrega as informações necessárias para construir um polipeptídeo (proteína ou subunidade de proteína). As transcrições eucarióticas precisam passar por algumas etapas de processamento antes da sua tradução em proteínas.
 Na transcrição, uma região de DNA se abre. Uma fita, a fita molde (ou template, do inglês), serve como gabarito para a síntese de um transcrito de RNA complementar. A outra fita, a fita codificadora, é idêntica ao RNA transcrito quanto à sequência, exceto que ela tem bases uracila (U) no lugar de bases timina (T).Exemplo:Fita codificante: 5'-ATGATCTCGTAA-3' Fita molde: 3'-TACTAGAGCATT-5' RNA transcrito: 5'-AUGAUCUCGUAA-3'Para um gene codificador de proteína, o RNA transcrito contém a informação necessária para sintetizar um polipeptídeo (proteína ou sub unidade de proteína) com uma sequência de aminoácidos determinada. Neste caso:RNA transcrito (agindo como RNA mensageiro): 5'-AUGAUCUCGUAA-3' Polipeptídeo: Met-Ile-Ser-STOP
Na transcrição, uma região de DNA se abre. Uma fita, a fita molde (ou template, do inglês), serve como gabarito para a síntese de um transcrito de RNA complementar. A outra fita, a fita codificadora, é idêntica ao RNA transcrito quanto à sequência, exceto que ela tem bases uracila (U) no lugar de bases timina (T).Exemplo:Fita codificante: 5'-ATGATCTCGTAA-3' Fita molde: 3'-TACTAGAGCATT-5' RNA transcrito: 5'-AUGAUCUCGUAA-3'Para um gene codificador de proteína, o RNA transcrito contém a informação necessária para sintetizar um polipeptídeo (proteína ou sub unidade de proteína) com uma sequência de aminoácidos determinada. Neste caso:RNA transcrito (agindo como RNA mensageiro): 5'-AUGAUCUCGUAA-3' Polipeptídeo: Met-Ile-Ser-STOP
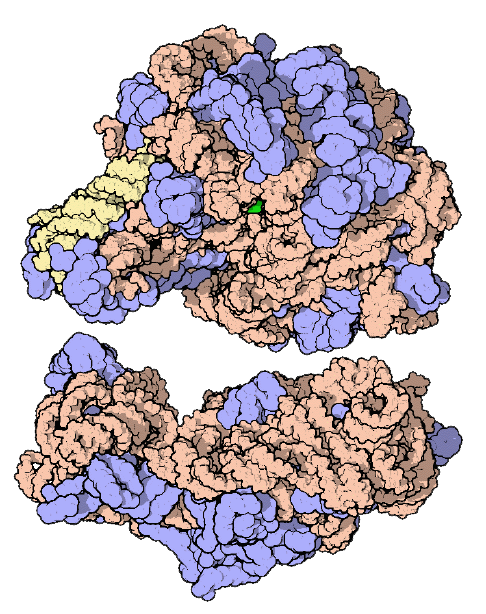
RNA polimerase
- A principal enzima envolvida na transcrição é a RNA polimerase, que usa um modelo de DNA de fita simples, para sintetizar uma fita complementar de RNA. Especificamente, a RNA polimerase constrói uma fita de RNA na direção de 5' para 3' , adicionando cada novo nucleotídeos à extremidade 3' do filamento.
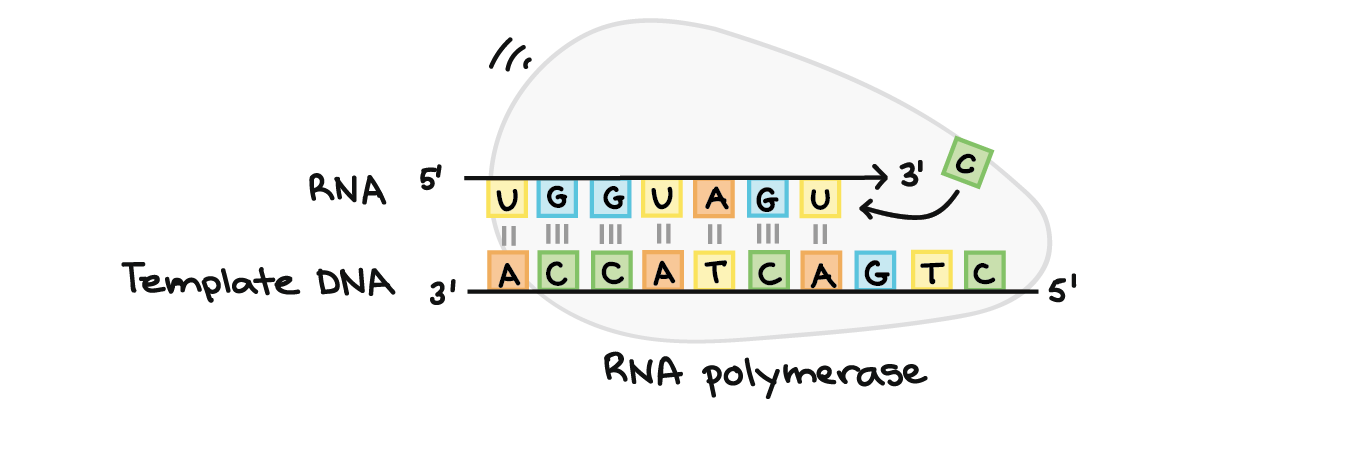 A RNA polimerase sintetiza uma sequência de RNA complementar à fita de DNA molde. Ela sintetiza a fita de RNA na direção de 5´para 3´, enquanto a leitura da fita molde de DNA ocorre na direção 3´para 5´. A fita de DNA molde e a fita de RNA são antiparalelas entre si.RNA transcrito: 5'-UGGUAGU...-3' (pontos indicando onde os nucleotídeos ainda estão sendo adicionados no final 3´) DNA molde: 3'-ACCATCAGTC-5'
A RNA polimerase sintetiza uma sequência de RNA complementar à fita de DNA molde. Ela sintetiza a fita de RNA na direção de 5´para 3´, enquanto a leitura da fita molde de DNA ocorre na direção 3´para 5´. A fita de DNA molde e a fita de RNA são antiparalelas entre si.RNA transcrito: 5'-UGGUAGU...-3' (pontos indicando onde os nucleotídeos ainda estão sendo adicionados no final 3´) DNA molde: 3'-ACCATCAGTC-5'
Etapas da transcrição
- A transcrição de um gene ocorre em três estágios: iniciação, alongamento e término. Aqui, veremos um resumo de como essas etapas ocorrem nas bactérias. Você pode aprender mais sobre os detalhes de cada etapa (e sobre as diferenças da transcrição nos eucariontes) no artigo estágios da transcrição.
- Iniciação. A RNA polimerase liga-se a uma sequência de DNA chamada promotor, encontrada próximo ao início de um gene. Cada gene (ou grupo de genes co-transcritos, nas bactérias) tem seu próprio promotor. Uma vez ligada, a RNA polimerase separa as fitas de DNA, provendo o molde de cadeia simples, de um só filamento, necessário à transcrição.
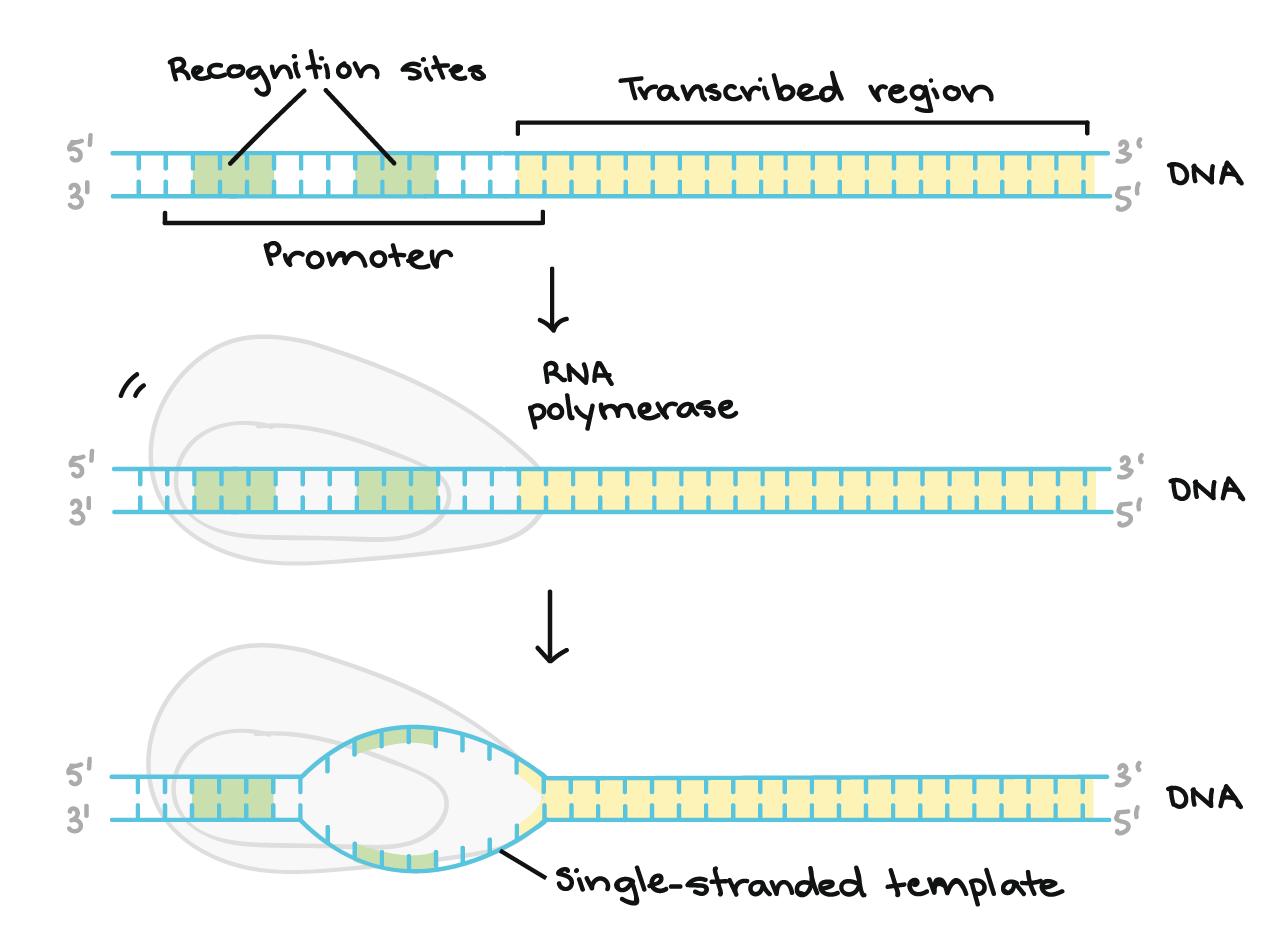 A região promotora precede (e ligeiramente sobrepõe-se) à região transcrita, cuja transcrição especifica. Ela contém locais de reconhecimento para a RNA polimerase ou suas proteínas auxiliares se ligarem. O DNA se abre na região promotora para que a RNA polimerase possa começar a transcrição.
A região promotora precede (e ligeiramente sobrepõe-se) à região transcrita, cuja transcrição especifica. Ela contém locais de reconhecimento para a RNA polimerase ou suas proteínas auxiliares se ligarem. O DNA se abre na região promotora para que a RNA polimerase possa começar a transcrição. - Alongamento. Um filamento de DNA, a fita molde, age como molde para a RNA polimerase. Conforme ela "lê" esse molde uma base por vez, a polimerase constrói uma molécula de RNA feita de nucleotídeos complementares, formando uma cadeia que cresce de 5´para 3´. O transcrito de RNA carrega a mesma informação que o filamento não-molde (codificador) de DNA , mas ele contém a base Uracila (U) em vez de tiamina (T).
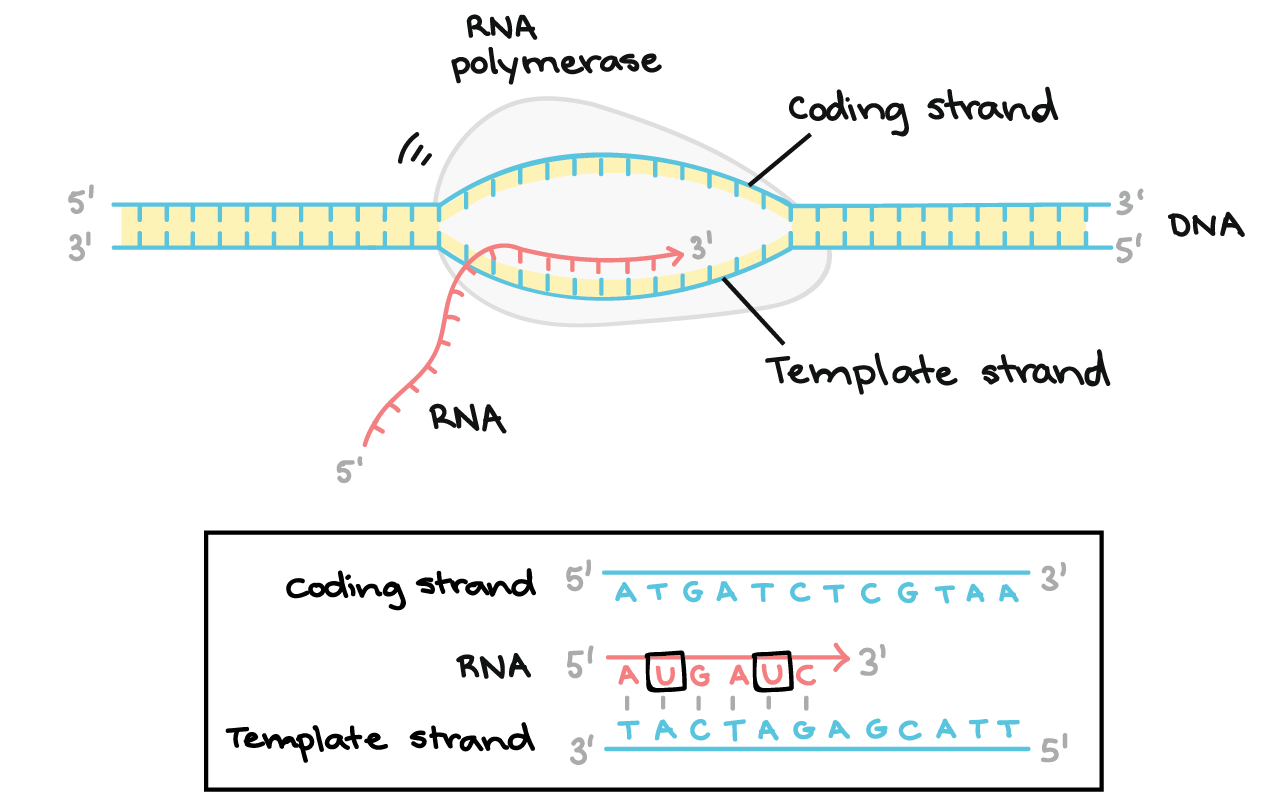 A RNA polimerase sintetiza um transcrito de RNA complementar à fita de DNA molde na direção 5' para 3'. Ela avança ao longo da fita molde na direção de 3' a 5', abrindo a dupla hélice do DNA, conforme avança. O RNA sintetizado só permanece ligado à fita molde por um curto período, então, deixa a polimerase como uma cadeia pendente, que permite que o DNA volte a fechar e formar uma dupla-hélice.Neste exemplo, as sequências da fita codificadora, da fita molde e do transcrito de RNA são:Fita codificadora: 5'- ATGATCTCGTAA-3'Fita molde: 3'-TACTAGAGCATT-5'RNA: 5'-AUGAUC...-3' (os pontos mostram onde nucleotídeos ainda estão sendo adicionados à fita em sua extremidade 3')
A RNA polimerase sintetiza um transcrito de RNA complementar à fita de DNA molde na direção 5' para 3'. Ela avança ao longo da fita molde na direção de 3' a 5', abrindo a dupla hélice do DNA, conforme avança. O RNA sintetizado só permanece ligado à fita molde por um curto período, então, deixa a polimerase como uma cadeia pendente, que permite que o DNA volte a fechar e formar uma dupla-hélice.Neste exemplo, as sequências da fita codificadora, da fita molde e do transcrito de RNA são:Fita codificadora: 5'- ATGATCTCGTAA-3'Fita molde: 3'-TACTAGAGCATT-5'RNA: 5'-AUGAUC...-3' (os pontos mostram onde nucleotídeos ainda estão sendo adicionados à fita em sua extremidade 3')- Término. Sequências chamadas finalizadores sinalizam que o transcrito de RNA está completo. Uma vez transcritos os finalizadores, o transcrito se libera da RNA polimerase. Um exemplo de um mecanismo de término envolvendo a formação de um grampo no RNA é mostrado abaixo.
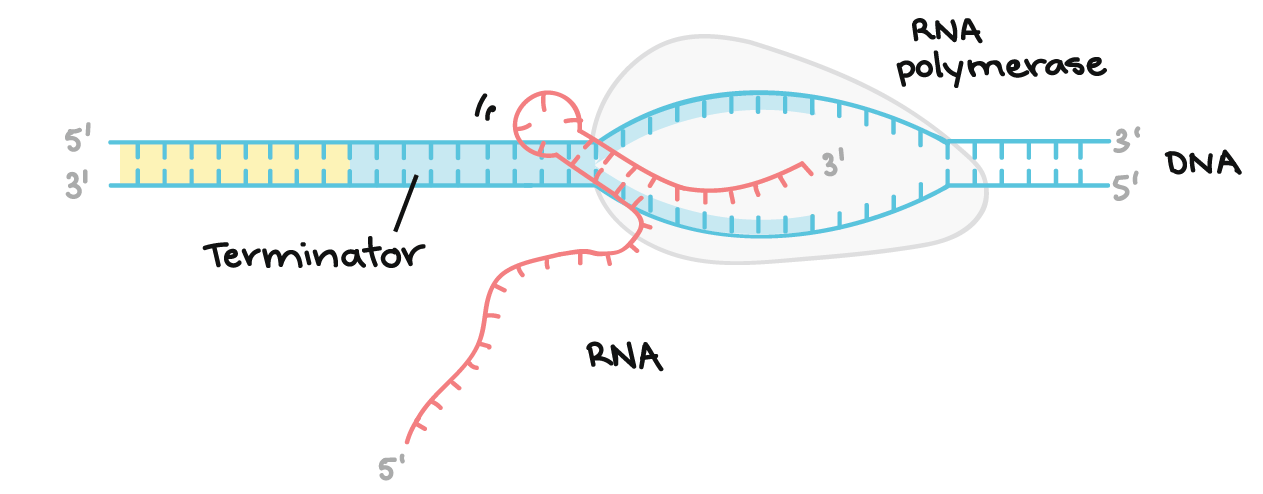 O DNA finalizador codifica uma região de RNA que forma uma estrutura em grampo, seguida por uma seqüência de nucleotídeos U. Essa estrutura em grampo no transcrito, faz com que a RNA polimerase pare. Os nucleotídeos U que vêm depois do grampo formam ligações fracas com os nucleotídeos A da fita molde de DNA, permitindo a separação do transcrito do molde e a finalização da transcrição.
O DNA finalizador codifica uma região de RNA que forma uma estrutura em grampo, seguida por uma seqüência de nucleotídeos U. Essa estrutura em grampo no transcrito, faz com que a RNA polimerase pare. Os nucleotídeos U que vêm depois do grampo formam ligações fracas com os nucleotídeos A da fita molde de DNA, permitindo a separação do transcrito do molde e a finalização da transcrição.
Modificações no RNA eucariota
- Nas bactérias, os transcritos de RNA podem atuar imediatamente como RNAs mensageiro (RNAms). Nos eucariontes, o transcrito de um gene codificador de proteínas é chamado um pré-RNAm e deve passar por um processamento extra antes de poder direcionar a tradução.
- Nos eucariontes os pré-RNAms devem ter suas pontas modificadas, pela adição de um cap 5' (no começo) e uma cauda poli A 3' (no final).
- Em muitos eucariontes os pré-RNAms sofrem splicing. Neste processo, partes do pré-RNAm (chamadas íntrons) são cortadas fora e as peças remanescentes (chamadas éxons) são unidas novamente.
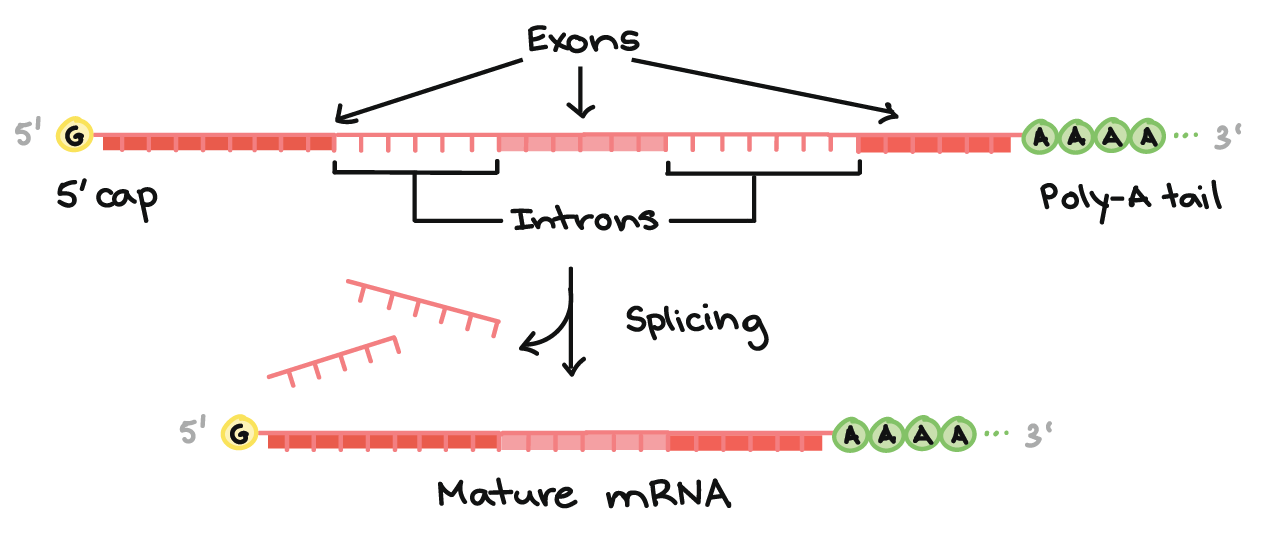 Parte superior da imagem: Diagrama de um pré-RNAm com um cap 5' e 3' cauda poli-A 3'. O cap 5' está na extremidade 5' do pré-RNAm e é um nucleotídeo G modificado. A cauda poli-A está na extremidade 3' do pré-RNAm e consiste de uma longa seqüência de nucleotídeos A (apenas alguns dos quais são mostrados).O pré-RNAm ainda contém éxons e íntrons. Ao longo do comprimento do RNAm, há um padrão alternado de éxons e íntrons: Éxon 1 - Íntron 1 - Éxon 2 - Íntron 2 - Éxon 3. Cada um consiste de um trecho de nucleotídeos de RNA.Durante o splicing, os íntrons são removidos do pré-RNAm, e os éxons são unidos para formar um RNAm maduro.Parte inferior da imagem: RNAm maduro que não contém as sequências de íntrons (Éxon 1 - Éxon 2 - Éxon 3 apenas).
Parte superior da imagem: Diagrama de um pré-RNAm com um cap 5' e 3' cauda poli-A 3'. O cap 5' está na extremidade 5' do pré-RNAm e é um nucleotídeo G modificado. A cauda poli-A está na extremidade 3' do pré-RNAm e consiste de uma longa seqüência de nucleotídeos A (apenas alguns dos quais são mostrados).O pré-RNAm ainda contém éxons e íntrons. Ao longo do comprimento do RNAm, há um padrão alternado de éxons e íntrons: Éxon 1 - Íntron 1 - Éxon 2 - Íntron 2 - Éxon 3. Cada um consiste de um trecho de nucleotídeos de RNA.Durante o splicing, os íntrons são removidos do pré-RNAm, e os éxons são unidos para formar um RNAm maduro.Parte inferior da imagem: RNAm maduro que não contém as sequências de íntrons (Éxon 1 - Éxon 2 - Éxon 3 apenas).
Modificações nas pontas aumentam a estabilidade do RNAm, enquanto o splicing dá ao RNAm sua seqüência correta. (Se nos íntrons não forem removidos, eles serão traduzidos juntamente com os éxons, produzindo um polipeptídeo "sem nexo".)Para saber mais sobre modificações de pré-RNAm em eucariontes, confira o artigo sobre processamento de pré-RNAm.
A transcrição acontece para genes individuais
- Nem todos os genes são transcritos todo tempo. Em vez disso, a transcrição é controlada individualmente para cada gene (ou, nas bactérias, para pequenos grupos de genes que são transcritos juntos). As células cuidadosamente regulam a transcrição, transcrevendo apenas os genes cujos produtos são necessários em um determinado momento.Por exemplo, o diagrama abaixo mostra um "instantâneo" de RNAs de uma célula imaginária num dado momento. Nesta célula, genes 1, 2 e 3, são transcritos, enquanto o gene 4 não é. Além disso, os genes, 1, 2 e 3 são transcritos em diferentes níveis, significando que números diferentes de moléculas de RNA são feitas para cada um.
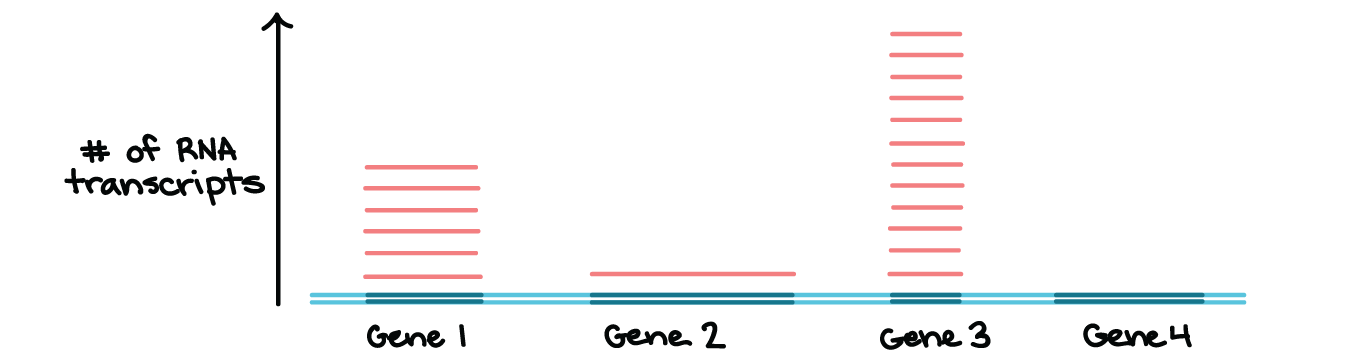 Diagrama mostrando que os genes individuais são transcritos em diferentes quantidades.Uma região de DNA contendo quatro genes é mostrada, com a região transcrita de cada gene destacada em azul escuro. O número de transcritos de cada gene é indicado acima do DNA (no eixo-Y). Existem seis transcritos do gene 1, um transcrito do gene 2, doze transcritos do gene 3 e nenhum transcrito do gene 4.Isto não é uma ilustração de conjunto real qualquer de genes e seus níveis de transcrição, mas ilustra que a transcrição é controlada individualmente para genes e outras unidades de transcrição.Nos artigos seguintes, daremos atenção mais detalhada à RNA polimerase, às etapas da transcrição e ao processo de modificação do RNA nos eucariontes. Vamos também considerar algumas diferenças importantes entre a transcrição bacteriana e a transcrição em eucariontes. Este artigo está autorizado sob licença CC BY-NC-SA 4.0.
Diagrama mostrando que os genes individuais são transcritos em diferentes quantidades.Uma região de DNA contendo quatro genes é mostrada, com a região transcrita de cada gene destacada em azul escuro. O número de transcritos de cada gene é indicado acima do DNA (no eixo-Y). Existem seis transcritos do gene 1, um transcrito do gene 2, doze transcritos do gene 3 e nenhum transcrito do gene 4.Isto não é uma ilustração de conjunto real qualquer de genes e seus níveis de transcrição, mas ilustra que a transcrição é controlada individualmente para genes e outras unidades de transcrição.Nos artigos seguintes, daremos atenção mais detalhada à RNA polimerase, às etapas da transcrição e ao processo de modificação do RNA nos eucariontes. Vamos também considerar algumas diferenças importantes entre a transcrição bacteriana e a transcrição em eucariontes. Este artigo está autorizado sob licença CC BY-NC-SA 4.0.
Referências:
- 3'-end cleavage and polyadenylation. (2016). In Nobelprize.org. Disponível em http://www.nobelprize.org/educational/medicine/dna/a/splicing/splicing_endformation.html.Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., and Walter, P. (2002). Electron-transport chains and their proton pumps. Em Molecular biology of the cell (4ª ed.). Nova York, NY: Garland Science. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK26904/.Berger, Shanna. (2006). transcrição eukaryotic. Em Transcription e RNA polimerase II. Disponível em http://www.chem.uwec.edu/Webpapers2006/sites/bergersl/pages/eukaryotic.html.Boundless (2016, January 8). Initiation of transcription in eukaryotes. Em Boundless biology. Disponível em https://www.boundless.com/biology/textbooks/boundless-biology-textbook/genes-and-proteins-15/eukaryotic-transcription-108/initiation-of-transcription-in-eukaryotes-445-11670/.Brown, T. A. (2002). Assembly of the transcription initiation complex. Em Genomes (2nd ed., Ch. 9). Oxford, UK: Wiley-Liss. Disponível em www.ncbi.nlm.nih.gov/books/NBK21115/.Griffiths, A. J. F., Miller, J. H., Suzuki, D. T., Lewontin, R. C., and Gelbart, W. M. (2000). Using genetic ratios. Em An introduction to genetic analysis (7th ed.). New York, NY: W. H. Freeman. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK21812/.Inverted repeat. (2016, February 13). Acesso em 13 de fevereiro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Inverted_repeat.Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). Bacterial transcription initiation. Em Molecular cell biology (4th ed., section 10.2). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21612/.Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). RNA polymerase II transcription-initiation complex. Em Molecular cell biology (4th ed., section 10.6). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21610/.Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). Transcription termination. Em Molecular cell biology (4th ed., section 11.1). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21601/.Moran, L. A. (2008, September 16). How RNA polymerase binds to DNA [Web log post]. Em Sandwalk: Strolling with a skeptical biochemist. Disponível em http://sandwalk.blogspot.com/2008/09/how-rna-polymerase-binds-to-dna.htmlPolyadenylation. (24 de janeiro de 2016). Acesso em 11 de fevereiro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Polyadenylation.Purves, W. K., Sadava, D. E., Orians, G. H., and Heller, H.C. (2004). Transcription: DNA-directed RNA synthesis. Em Life: the science of biology (7th ed., pp. 237-239). Sunderland, MA: Sinauer Associates.Raven, P. H., Johnson, G. B., Mason, K. A., Losos, J. B., and Singer, S. R. (2014). Genes and how they work. Em Biology (10th ed., AP ed., pp. 278-303). New York, NY: McGraw-Hill.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Transcription is the DNA-directed synthesis of RNA: A closer look.. Em Campbell biology (10th ed., pp. 340-342). San Francisco, CA: Pearson.Saunders, A., Core, L. J., and Lis, J. T. (2006). Nature Reviews Molecular Cell Biology, 7, 557-567. http://dx.doi.org/10.1038/nrm1981.Webb, S. Witte, L., Wong, K., Woreta, T., and Yoo, E. (2002, May 8). TFIIH. Em RNA polymerase II in eukaryotes and prokaryotes. Disponível em http://www.biochem.umd.edu/biochem/kahn/molmachines/newpolII/TFIIH.html. Etapas da transcrição
Pontos Principais:
- A transcrição é o processo no qual um gene da sequência de DNA é copiado (transcrito) para fazer uma molécula de RNA.
- RNA polimerase é a principal enzima de transcrição.
- A transcrição começa quando a RNA polimerase se liga a uma sequência promotora próxima ao início de um gene (diretamente ou através das proteínas auxiliares).
- A RNA polimerase usa uma das fitas de DNA (a fita molde) como uma referência para fazer uma molécula de RNA nova, complementar.
- A transcrição acaba num processo chamado terminação. A terminação depende das sequências no RNA, que sinalizam que a transcrição acabou.
Introdução
- O que torna a morte por cogumelo cicuta verde mortal? Estes cogumelos obtêm seus efeitos letais ao produzir uma toxina específica, a qual se acopla a uma enzima crucial no corpo humano: a RNA polimerase.1start superscript, 1, end superscript
 Fotografia de cogumelos Amanita phalloides (cicuta verde)._Imagem adaptada de "Amanita phalloides," por Archenzo (CC BY-SA 3.0), sob licença CC BY-SA 3.0 license._A RNA polimerase é crucial porque ela executa a transcrição, o processo de copiar o DNA (ácido desoxirribonucleico, o material genético) em RNA (ácido ribonucleico, uma molécula similar porém de vida mais curta).A transcrição é um passo essencial no uso da informação de genes em nosso DNA para fazer proteínas. As proteínas são as principais moléculas que dão estrutura às células e que as mantêm funcionando. Bloquear a transcrição com a toxina do cogumelo provoca falha hepática e morte, porque nenhum RNA novo - e portanto, nenhuma nova proteína - pode ser feito.2squaredA transcrição é essencial para a vida e compreender como ela funciona é importante para a saúde humana. Vamos dar uma olhada no que acontece durante a transcrição.
Fotografia de cogumelos Amanita phalloides (cicuta verde)._Imagem adaptada de "Amanita phalloides," por Archenzo (CC BY-SA 3.0), sob licença CC BY-SA 3.0 license._A RNA polimerase é crucial porque ela executa a transcrição, o processo de copiar o DNA (ácido desoxirribonucleico, o material genético) em RNA (ácido ribonucleico, uma molécula similar porém de vida mais curta).A transcrição é um passo essencial no uso da informação de genes em nosso DNA para fazer proteínas. As proteínas são as principais moléculas que dão estrutura às células e que as mantêm funcionando. Bloquear a transcrição com a toxina do cogumelo provoca falha hepática e morte, porque nenhum RNA novo - e portanto, nenhuma nova proteína - pode ser feito.2squaredA transcrição é essencial para a vida e compreender como ela funciona é importante para a saúde humana. Vamos dar uma olhada no que acontece durante a transcrição.
Visão Geral da Transcrição
- A Transcrição é o primeiro passo da expressão gênica. Durante esse processo, a sequência de DNA de um gene é copiada em RNA.Antes que a transcrição possa ocorrer, a dupla hélice de DNA deve se desenrolar próximo ao gene que está sendo transcrito. A região do DNA aberto é chamada de bolha de transcrição.
![Na transcrição, uma região de DNA se abre. Uma fita, a fita molde (ou template, do inglês), serve como gabarito para a síntese de um transcrito de RNA complementar. A outra fita, a fita codificadora, é idêntica ao RNA transcrito quanto à sequência, exceto que ela tem bases uracila (U) no lugar de bases timina (T).
Exemplo:
Fita codificante: 5'-ATGATCTCGTAA-3'
Fita molde: 3'-TACTAGAGCATT-5'
RNA transcrito: 5'-AUGAUCUCGUAA-3'
Na tradução, o RNA transcrito é lido para produzir um polipeptídeo.
Exemplo:
Transcrito de RNA: 5'-AUG AUC UCG UAA-3'
Polipeptídeo: (N-terminal) Met - Ile - Ser - [STOP] (C-terminal)](https://cdn.kastatic.org/ka-perseus-images/534d6b2edba81dc1803eb97ba4de457c48de28af.png) Na transcrição, uma região de DNA se abre. Uma fita, a fita molde (ou template, do inglês), serve como gabarito para a síntese de um transcrito de RNA complementar. A outra fita, a fita codificadora, é idêntica ao RNA transcrito quanto à sequência, exceto que ela tem bases uracila (U) no lugar de bases timina (T).Exemplo:Fita codificante: 5'-ATGATCTCGTAA-3' Fita molde: 3'-TACTAGAGCATT-5' RNA transcrito: 5'-AUGAUCUCGUAA-3'Na tradução, o RNA transcrito é lido para produzir um polipeptídeo.Exemplo:Transcrito de RNA: 5'-AUG AUC UCG UAA-3' Polipeptídeo: (N-terminal) Met - Ile - Ser - [STOP] (C-terminal)A transcrição usa uma das duas fitas de DNA expostas como molde; essa fita é chamada de fita molde. O produto de RNA é complementar à fita molde e é quase idêntico à outra fita de DNA, chamada de fita não-molde (ou codificante). No entanto, existe uma diferença importante: no RNA recém-formado todos os nucleótidos T são substituídos por nucleótidos U.O local no DNA de onde o primeiro nucleotídeo de RNA é transcrito é chamado de local +1plus, 1, ou sítio de iniciação. Nucleotídeos que vem antes do sítio de iniciação recebem números negativos e são ditos estar a montante. Nucleotídeos que vem depois do sítio de iniciação são marcados com números positivos e ditos estar a jusante.Se o gene transcrito codifica uma proteína (o que muitos genes fazem), a molécula de RNA será lida para fazer uma proteína em um processo chamado de tradução.
Na transcrição, uma região de DNA se abre. Uma fita, a fita molde (ou template, do inglês), serve como gabarito para a síntese de um transcrito de RNA complementar. A outra fita, a fita codificadora, é idêntica ao RNA transcrito quanto à sequência, exceto que ela tem bases uracila (U) no lugar de bases timina (T).Exemplo:Fita codificante: 5'-ATGATCTCGTAA-3' Fita molde: 3'-TACTAGAGCATT-5' RNA transcrito: 5'-AUGAUCUCGUAA-3'Na tradução, o RNA transcrito é lido para produzir um polipeptídeo.Exemplo:Transcrito de RNA: 5'-AUG AUC UCG UAA-3' Polipeptídeo: (N-terminal) Met - Ile - Ser - [STOP] (C-terminal)A transcrição usa uma das duas fitas de DNA expostas como molde; essa fita é chamada de fita molde. O produto de RNA é complementar à fita molde e é quase idêntico à outra fita de DNA, chamada de fita não-molde (ou codificante). No entanto, existe uma diferença importante: no RNA recém-formado todos os nucleótidos T são substituídos por nucleótidos U.O local no DNA de onde o primeiro nucleotídeo de RNA é transcrito é chamado de local +1plus, 1, ou sítio de iniciação. Nucleotídeos que vem antes do sítio de iniciação recebem números negativos e são ditos estar a montante. Nucleotídeos que vem depois do sítio de iniciação são marcados com números positivos e ditos estar a jusante.Se o gene transcrito codifica uma proteína (o que muitos genes fazem), a molécula de RNA será lida para fazer uma proteína em um processo chamado de tradução.
RNA polimerase
- RNA polimerases são enzimas que transcrevem DNA em RNA. Usando um molde de DNA, a RNA polimerase constrói uma nova molécula de RNA através do pareamento de bases. Por exemplo, se houver um G no molde de DNA, a RNA polimerase adicionará um C à nova cadeia crescente de RNA.
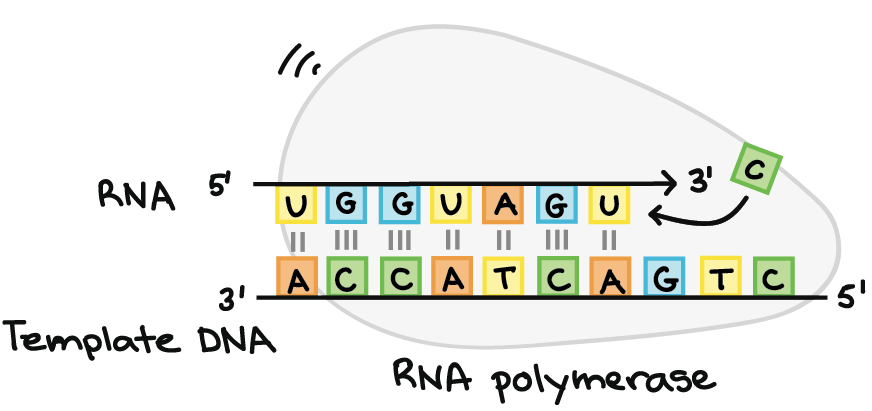 A RNA polimerase sintetiza uma sequência de RNA complementar à fita de DNA molde. Ela sintetiza a fita de RNA na direção de 5´para 3´, enquanto a leitura da fita molde de DNA ocorre na direção 3´para 5´. A fita de DNA molde e a fita de RNA são antiparalelas entre si.RNA transcrito: 5'-UGGUAGU...-3' (pontos indicando onde os nucleotídeos ainda estão sendo adicionados no final 3´) DNA molde: 3'-ACCATCAGTC-5'A RNA polimerase sempre constrói uma nova fita de RNA na direção 5' para 3'. Isto é, ela só pode adicionar nucleótidos de RNA (A, U, C ou G) à extremidade 3' da cadeia.RNA polimerases são grandes enzimas com múltiplas subunidades, mesmo em organismos simples como bactérias. Além disso, os seres humanos e outros eucariontes têm três tipos diferentes de RNA polimerases: I, II e III. Cada uma especializa-se em transcrever determinadas classes de genes.
A RNA polimerase sintetiza uma sequência de RNA complementar à fita de DNA molde. Ela sintetiza a fita de RNA na direção de 5´para 3´, enquanto a leitura da fita molde de DNA ocorre na direção 3´para 5´. A fita de DNA molde e a fita de RNA são antiparalelas entre si.RNA transcrito: 5'-UGGUAGU...-3' (pontos indicando onde os nucleotídeos ainda estão sendo adicionados no final 3´) DNA molde: 3'-ACCATCAGTC-5'A RNA polimerase sempre constrói uma nova fita de RNA na direção 5' para 3'. Isto é, ela só pode adicionar nucleótidos de RNA (A, U, C ou G) à extremidade 3' da cadeia.RNA polimerases são grandes enzimas com múltiplas subunidades, mesmo em organismos simples como bactérias. Além disso, os seres humanos e outros eucariontes têm três tipos diferentes de RNA polimerases: I, II e III. Cada uma especializa-se em transcrever determinadas classes de genes.
Transcrição: iniciação
- Para iniciar a transcrição de um gene, a RNA polimerase liga-se ao DNA do gene numa região denominada promotor. Basicamente, o promotor informa à polimerase onde "se sentar" no DNA e começar a transcrever.
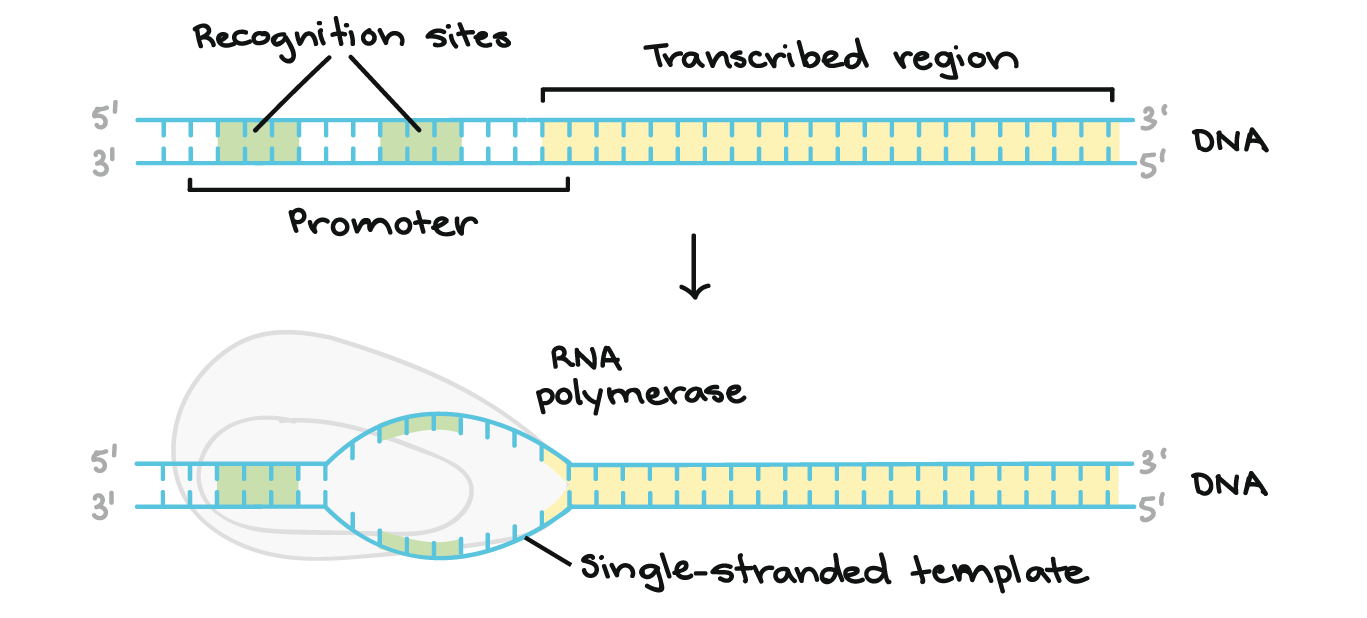 A região promotora precede (e ligeiramente sobrepõe-se) à região transcrita, cuja transcrição especifica. Ela contém locais de reconhecimento para a RNA polimerase ou suas proteínas auxiliares se ligarem. O DNA se abre na região promotora para que a RNA polimerase possa começar a transcrição.Cada gene (ou, em bactérias, cada grupo de genes transcritos juntos) tem seu próprio promotor. Um promotor contém sequências de DNA que deixam a RNA polimerase ou as suas proteínas auxiliares se ligarem ao DNA. Uma vez formada a bolha de transcrição, a polimerase pode iniciar a transcrição.
A região promotora precede (e ligeiramente sobrepõe-se) à região transcrita, cuja transcrição especifica. Ela contém locais de reconhecimento para a RNA polimerase ou suas proteínas auxiliares se ligarem. O DNA se abre na região promotora para que a RNA polimerase possa começar a transcrição.Cada gene (ou, em bactérias, cada grupo de genes transcritos juntos) tem seu próprio promotor. Um promotor contém sequências de DNA que deixam a RNA polimerase ou as suas proteínas auxiliares se ligarem ao DNA. Uma vez formada a bolha de transcrição, a polimerase pode iniciar a transcrição.
Promotores nas bactérias
- Para obter uma melhor ideia de como funciona um promotor, vamos olhar um exemplo de bactérias. Um promotor bacteriano típico contém duas sequências de DNA importantes, os elementos
- 1010 e- 3535.A RNA polimerase reconhece e se liga diretamente à essas sequências. As sequências posicionam a polimerase no ponto certo para iniciar a transcrição de um gene alvo e elas também asseguram que a mesma está apontando para direção correta.Assim que a RNA polimerase se estabelece, ela abre o DNA e começa a trabalhar. A abertura de DNA ocorre no elemento- 1010 , onde as fitas são fáceis de separar devido aos diversos As e Ts (pois se ligam entre si apenas com duas ligações de hidrogênio, em vez das três ligações de hidrogênio entre os Gs e Cs).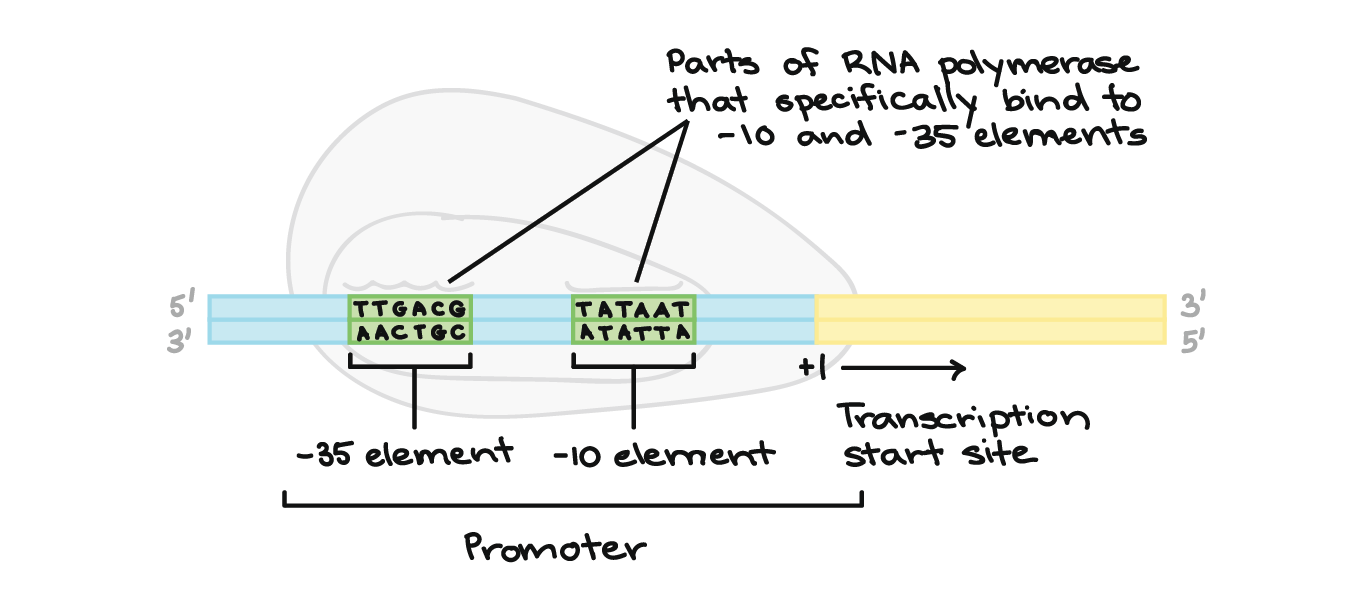 Promotor bacteriano. O promotor encontra-se no início da região transcrita, engloba o DNA anterior e se sobrepõe ligeiramente ao inicio do sitio transcricional. O promotor contém dois elementos, o elemento -35 e o elemento -10. O elemento -35 localiza-se em torno de 35 nucleotídeos antes do início do sitio transcricional (+1), enquanto o elemento -10 em torno de 10 nucleotídeos antes do inicio do sitio transcricional. Neste exemplo específico, a sequência do elemento -35 (na fita codificante) é 5'-TTGACG-3 ', enquanto a sequência do elemento -10 (na fita codificante) é 5'-TATAAT-3'. A RNA polimerase possui regiões que se ligam especificamente aos elementos -10 e -35.Os elementos
Promotor bacteriano. O promotor encontra-se no início da região transcrita, engloba o DNA anterior e se sobrepõe ligeiramente ao inicio do sitio transcricional. O promotor contém dois elementos, o elemento -35 e o elemento -10. O elemento -35 localiza-se em torno de 35 nucleotídeos antes do início do sitio transcricional (+1), enquanto o elemento -10 em torno de 10 nucleotídeos antes do inicio do sitio transcricional. Neste exemplo específico, a sequência do elemento -35 (na fita codificante) é 5'-TTGACG-3 ', enquanto a sequência do elemento -10 (na fita codificante) é 5'-TATAAT-3'. A RNA polimerase possui regiões que se ligam especificamente aos elementos -10 e -35.Os elementos- 1010 e- 3535 são nomeados assim porque eles vêm 3535 e 1010 nucleotídeos antes do sítio de iniciação (+1plus, 1 no DNA). Os sinais de menos apenas significam que eles estão antes, e não depois, do sítio de iniciação.
Promotores em humanos
- Em eucariontes como humanos, a principal RNA polimerase em suas células não se liga diretamente aos promotores como a RNA polimerase bacteriana. Ao invés disso, proteínas acessórias chamadas fatores de transcrição basais (gerais) se ligam primeiramente ao promotor, auxiliando a RNA polimerase em suas células a obter um ponto de apoio no DNA.Muitos promotores eucarióticos possuem uma sequência chamada de TATA box. O "TATA box" tem um papel muito similar ao elemento
- 1010 em bactérias. Ele é reconhecido por um dos fatores gerais de transcrição, permitindo que outros fatores de transcrição e eventualmente a RNA polimerase se liguem. Ele também contém muitos As e Ts, o que torna mais fácil de separar as fitas de DNA.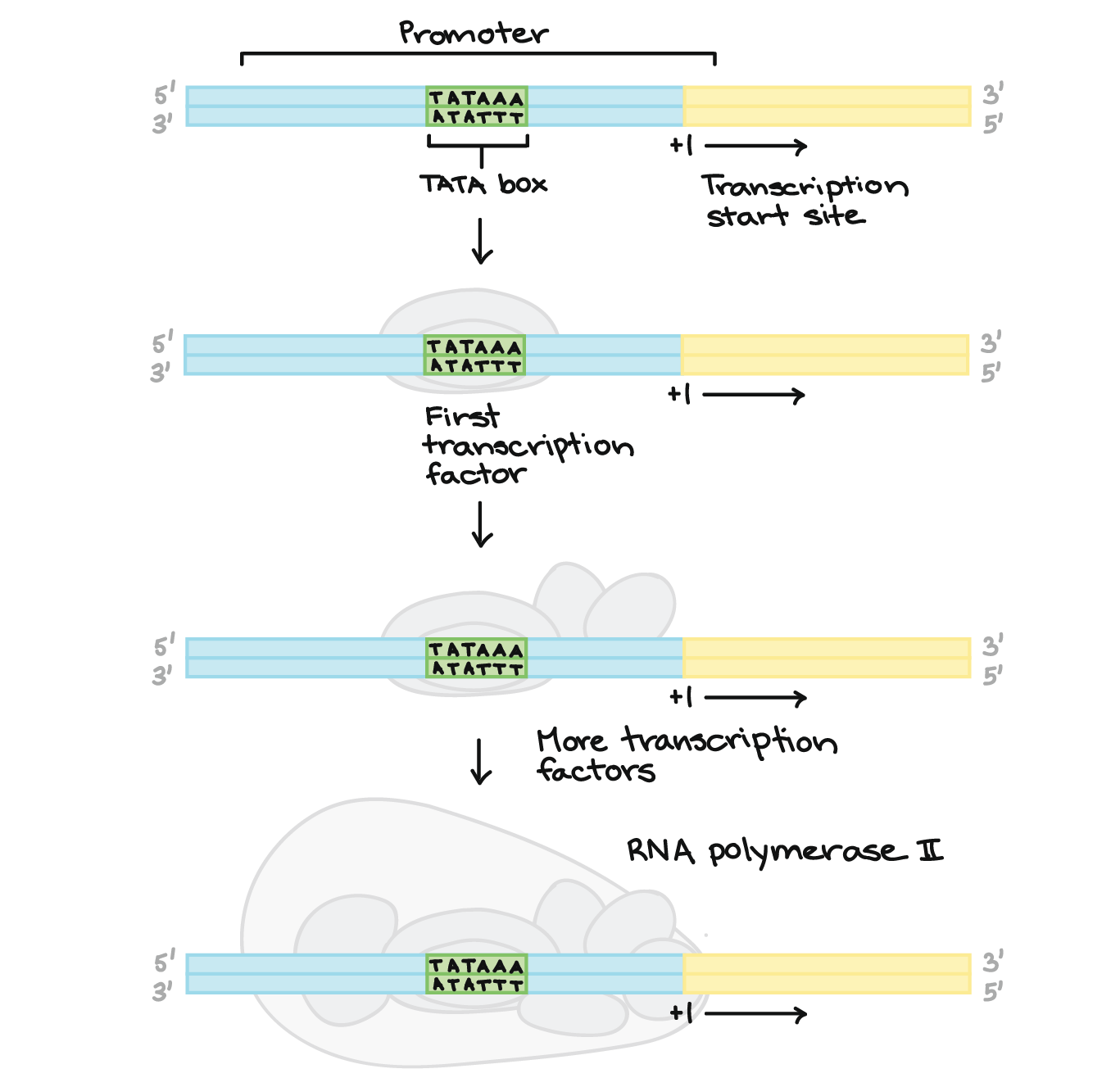 O promotor de um gene eucariótico é representado. O promotor se encontra anteriormente e ligeiramente sobreposto ao sítio de iniciação transcricional (+1). Ele contém o TATA box, que possui uma sequência (na fita codificante) de 5'-TATAAA-3'. O primeiro fator de transcrição geral eucariótico se liga ao TATA box. Então, outros fatores de transcrição geral se ligam. Finalmente, a RNA polimerase II e alguns fatores de transcrição adicionais se ligam ao promotor.
O promotor de um gene eucariótico é representado. O promotor se encontra anteriormente e ligeiramente sobreposto ao sítio de iniciação transcricional (+1). Ele contém o TATA box, que possui uma sequência (na fita codificante) de 5'-TATAAA-3'. O primeiro fator de transcrição geral eucariótico se liga ao TATA box. Então, outros fatores de transcrição geral se ligam. Finalmente, a RNA polimerase II e alguns fatores de transcrição adicionais se ligam ao promotor.
Alongamento
- Uma vez que a RNA polimerase está na posição do promotor, o próximo passo da transcrição — o alongamento — pode começar. Basicamente, o a longamento é a fase que a sequência de RNA fica mais longa, graças à adição de novos nucleotídeos.Durante o alongamento, a RNA polimerase "caminha" ao longo de uma fita de DNA, conhecida como fita molde, da 3' para 5'. Para cada nucleotídeo no molde, a RNA polimerase adiciona um nucleotídeo de RNA correspondente (complementar) à extremidade 3' da fita do RNA.
 A RNA polimerase sintetiza um transcrito de RNA complementar à fita de DNA molde na direção 5' para 3'. Ela avança ao longo da fita molde na direção de 3' a 5', abrindo a dupla hélice do DNA, conforme avança. O RNA sintetizado só permanece ligado à fita molde por um curto período, então, deixa a polimerase como uma cadeia pendente, que permite que o DNA volte a fechar e formar uma dupla-hélice.Neste exemplo, as sequências da fita codificadora, da fita molde e do transcrito de RNA são:Fita codificadora: 5'- ATGATCTCGTAA-3'Fita molde: 3'-TACTAGAGCATT-5'RNA: 5'-AUGAUC...-3' (os pontos mostram onde nucleotídeos ainda estão sendo adicionados à fita em sua extremidade 3')O transcrito de RNA é quase idêntico à fita de DNA não-molde ou codificante. Contudo, as fitas de RNA têm a base uracila (U) em vez de timina (T), assim como um açúcar ligeiramente diferente no nucleótido. Assim, como podemos ver no diagrama acima, cada T da fita codificante é substituído por um U no transcrito de RNA.A figura abaixo mostra o DNA sendo transcrito por muitas RNA polimerases ao mesmo tempo, cada uma com uma "cauda" de RNA atrás dela. As polimerases próximas ao início do gene têm caudas de RNA curtas que ficam cada vez maiores à medida que a polimerase transcreve mais do gene.
A RNA polimerase sintetiza um transcrito de RNA complementar à fita de DNA molde na direção 5' para 3'. Ela avança ao longo da fita molde na direção de 3' a 5', abrindo a dupla hélice do DNA, conforme avança. O RNA sintetizado só permanece ligado à fita molde por um curto período, então, deixa a polimerase como uma cadeia pendente, que permite que o DNA volte a fechar e formar uma dupla-hélice.Neste exemplo, as sequências da fita codificadora, da fita molde e do transcrito de RNA são:Fita codificadora: 5'- ATGATCTCGTAA-3'Fita molde: 3'-TACTAGAGCATT-5'RNA: 5'-AUGAUC...-3' (os pontos mostram onde nucleotídeos ainda estão sendo adicionados à fita em sua extremidade 3')O transcrito de RNA é quase idêntico à fita de DNA não-molde ou codificante. Contudo, as fitas de RNA têm a base uracila (U) em vez de timina (T), assim como um açúcar ligeiramente diferente no nucleótido. Assim, como podemos ver no diagrama acima, cada T da fita codificante é substituído por um U no transcrito de RNA.A figura abaixo mostra o DNA sendo transcrito por muitas RNA polimerases ao mesmo tempo, cada uma com uma "cauda" de RNA atrás dela. As polimerases próximas ao início do gene têm caudas de RNA curtas que ficam cada vez maiores à medida que a polimerase transcreve mais do gene. Na imagem de microscopia representada aqui, um gene está sendo transcrito por várias RNA polimerases de uma vez. As cadeias de RNA são menores próximas ao começo do gene e se tornam maiores à medida que a enzima se move para o final do gene. Esse padrão cria uma espécie de estrutura em formato de cunha feita pelos transcritos de RNA se abrindo a partir do DNA do gene._Imagem modificada de "Transcription label en," by Dr. Hans-Heinrich Trepte (CC BY-SA 3.0). A imagem modificada está licenciada sob a licença CC BY-SA 3.0._
Na imagem de microscopia representada aqui, um gene está sendo transcrito por várias RNA polimerases de uma vez. As cadeias de RNA são menores próximas ao começo do gene e se tornam maiores à medida que a enzima se move para o final do gene. Esse padrão cria uma espécie de estrutura em formato de cunha feita pelos transcritos de RNA se abrindo a partir do DNA do gene._Imagem modificada de "Transcription label en," by Dr. Hans-Heinrich Trepte (CC BY-SA 3.0). A imagem modificada está licenciada sob a licença CC BY-SA 3.0._
Transcrição: terminação
- A RNA polimerase vai continuar transcrevendo até encontrar sinais para parar. O processo de término da transcrição é chamado terminação e isso acontece uma vez que a polimerase transcreve uma sequência de DNA conhecida como terminador.
Terminação em bactérias
- Existem duas estratégias de terminação principais encontradas em bactérias: Rho-dependente e Rho-independente.Em terminações Rho-dependente, o RNA contém um sítio de ligação para uma proteína chamada fator Rho. O fator Rho se liga à essa sequência e começa a "subir" o transcrito em direção à RNA polimerase.
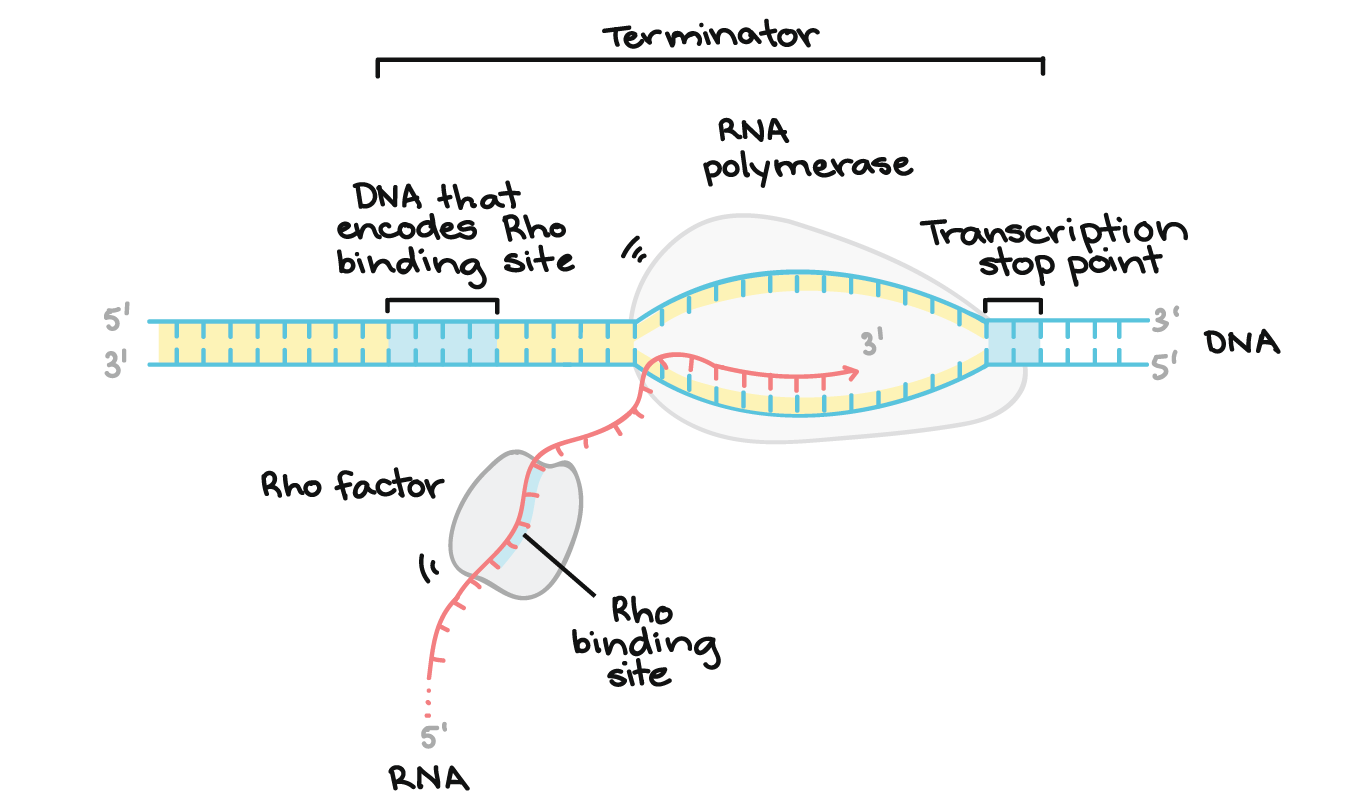 Terminação Rho-dependente. O terminador é uma região de DNA que inclui a sequência que codifica o sítio de ligação de Rho no RNAm, bem como o ponto real de parada da transcrição (que é uma sequência que faz com que a RNA polimerase pause para que a Rho possa alcançá-la). A Rho liga-se ao sítio de ligação de Rho no mRNA e sobe o transcrito de RNA, no sentido 5' para 3', em direção à bolha de transcrição onde está a polimerase. Quando ela alcança a polimerase, fará com que o transcrito seja liberado, terminando a transcrição.Quando ele alcança a polimerase na bolha de transcrição, o Rho puxa a transcrição do RNA e o molde de DNA se separa, liberando a molécula de RNA e terminando a transcrição. Outra sequência, encontrada mais tarde no DNA, chamada de ponto de parada da transcrição, faz com que a RNA polimerase pause e portanto ajuda o Rho alcançar.4start superscript, 4, end superscriptTerminações Rho-independentes dependem das sequências específicas do modelo do DNA. Enquanto a RNA polimerase se aproxima do final do gene que está sendo transcrito, ele atinge uma região rica em nucleotídeos C e G. O RNA transcrito desta região se dobra de volta para si mesmo e os nucleotídeos complementares C e G se ligam. O resultado é um grampo de cabelo estável que faz com que a RNA polimerase fique presa.
Terminação Rho-dependente. O terminador é uma região de DNA que inclui a sequência que codifica o sítio de ligação de Rho no RNAm, bem como o ponto real de parada da transcrição (que é uma sequência que faz com que a RNA polimerase pause para que a Rho possa alcançá-la). A Rho liga-se ao sítio de ligação de Rho no mRNA e sobe o transcrito de RNA, no sentido 5' para 3', em direção à bolha de transcrição onde está a polimerase. Quando ela alcança a polimerase, fará com que o transcrito seja liberado, terminando a transcrição.Quando ele alcança a polimerase na bolha de transcrição, o Rho puxa a transcrição do RNA e o molde de DNA se separa, liberando a molécula de RNA e terminando a transcrição. Outra sequência, encontrada mais tarde no DNA, chamada de ponto de parada da transcrição, faz com que a RNA polimerase pause e portanto ajuda o Rho alcançar.4start superscript, 4, end superscriptTerminações Rho-independentes dependem das sequências específicas do modelo do DNA. Enquanto a RNA polimerase se aproxima do final do gene que está sendo transcrito, ele atinge uma região rica em nucleotídeos C e G. O RNA transcrito desta região se dobra de volta para si mesmo e os nucleotídeos complementares C e G se ligam. O resultado é um grampo de cabelo estável que faz com que a RNA polimerase fique presa. Terminação independente de Rho. A sequencia de DNA terminadora codifica uma região de RNA que dobra sobre si mesma para formar um grampo. O grampo é seguido por séries de nucleotídeos U no RNA (não mostrado). O grampo causa a interrupção da polimerase, e o pareamento fraco entre os nucleotídeos A do DNA molde e U do transcrito de RNA permitem a separação entre o transcrito e o molde, finalizando a transcrição.Em uma terminação, o grampo de cabelo é seguido de um trechos de nucleotídeos U no RNA, que se pareiam com nucleotídeos A no modelo de DNA. A região complementar U-A da transcrição do RNA forma somente uma fraca interação com o modelo de DNA. isto, juntamente com a polimerase paralisada, produz instabilidade suficiente para a enzima cair e liberar o novo RNA transcrito.
Terminação independente de Rho. A sequencia de DNA terminadora codifica uma região de RNA que dobra sobre si mesma para formar um grampo. O grampo é seguido por séries de nucleotídeos U no RNA (não mostrado). O grampo causa a interrupção da polimerase, e o pareamento fraco entre os nucleotídeos A do DNA molde e U do transcrito de RNA permitem a separação entre o transcrito e o molde, finalizando a transcrição.Em uma terminação, o grampo de cabelo é seguido de um trechos de nucleotídeos U no RNA, que se pareiam com nucleotídeos A no modelo de DNA. A região complementar U-A da transcrição do RNA forma somente uma fraca interação com o modelo de DNA. isto, juntamente com a polimerase paralisada, produz instabilidade suficiente para a enzima cair e liberar o novo RNA transcrito.
O que acontece com o transcrito de RNA?
- Após a terminação, a transcrição está concluída. Um transcrito de RNA que está pronto para ser utilizado na tradução é chamado de RNA mensageiro (RNAm). Em bactérias, os transcritos de RNA estão prontos para serem traduzidos logo após a transcrição. Na verdade, eles estão realmente prontos um pouco mais cedo do que isso: a tradução pode começar enquanto a transcrição ainda está acontecendo!No diagrama abaixo, os RNAm estão sendo transcritos a partir de vários genes diferentes. Embora a transcrição ainda esteja em andamento, os ribossomos se ligaram a cada RNAm e começaram a traduzí-lo em proteína. Quando um RNAm está sendo traduzido por múltiplos ribossomos, diz-se que o RNAm e os ribossomos formam um polirribossomo.
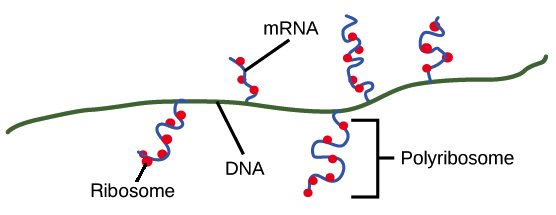 A ilustração mostra mRNAs que estão sendo transcritos fora dos genes. Ribossomos se ligam aos mRNAs antes da transcrição ser concluída e começam a fazer proteínas.Imagem modificada de "Transcrição procariótica: Figura 3, por OpenStax College, Biology, CC BY 4.0.Por que a transcrição e a tradução podem ocorrer simultaneamente para um RNAm em bactérias? Uma razão é que estes processos ocorrem na mesma direção de 5' para 3'. Isso significa que um pode seguir ou "perseguir" o outro que ainda está ocorrendo. Além disso, nas bactérias, não existem compartimentos internos de membranas para separar a transcrição da tradução.O quadro é diferente nas células de seres humanos e outros eucariontes. Isso acontece porque a transcrição ocorre no núcleo de células humanas, enquanto a tradução ocorre no citosol. Além disso, em eucariotos, as moléculas de RNA precisam passar por etapas especiais de processamento antes da tradução. Isso significa que a tradução não pode começar até que a transcrição e o processamento de RNA estejam totalmente concluídos. Você pode aprender mais sobre esses passos no vídeo transcrição e processamento de RNA.
A ilustração mostra mRNAs que estão sendo transcritos fora dos genes. Ribossomos se ligam aos mRNAs antes da transcrição ser concluída e começam a fazer proteínas.Imagem modificada de "Transcrição procariótica: Figura 3, por OpenStax College, Biology, CC BY 4.0.Por que a transcrição e a tradução podem ocorrer simultaneamente para um RNAm em bactérias? Uma razão é que estes processos ocorrem na mesma direção de 5' para 3'. Isso significa que um pode seguir ou "perseguir" o outro que ainda está ocorrendo. Além disso, nas bactérias, não existem compartimentos internos de membranas para separar a transcrição da tradução.O quadro é diferente nas células de seres humanos e outros eucariontes. Isso acontece porque a transcrição ocorre no núcleo de células humanas, enquanto a tradução ocorre no citosol. Além disso, em eucariotos, as moléculas de RNA precisam passar por etapas especiais de processamento antes da tradução. Isso significa que a tradução não pode começar até que a transcrição e o processamento de RNA estejam totalmente concluídos. Você pode aprender mais sobre esses passos no vídeo transcrição e processamento de RNA.
Créditos:
- Este artigo foi produzido com base nos seguintes artigos:
- "Prokaryotic transcription," por OpenStax College, Biology, CC BY 4.0. Faça o download gratuito do artigo original em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@10.61.
- "Eukaryotic transcription," por OpenStax College, Biology, CC BY 4.0. Faça o download gratuito do artigo original em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@10.61.
O artigo adaptado está autorizado sob a licença CC BY-NC-SA 4.0
Referências:
- Berger, S. (2006). The mushroom Amanita phalloides. Em Transcription and RNA polymerase II. Disponível em http://www.chem.uwec.edu/Webpapers2006/sites/bergersl/pages/amanitin.html.
- Amanita phalloides. (2016, February 6). Acessado em February 13, 2016 Disponível em Wikipedia: https://en.wikipedia.org/wiki/Amanita_phalloides.
- CyberBridge. (2007). RNA structure. Em Structure of DNA. Disponível em http://cyberbridge.mcb.harvard.edu/dna_3.html.
- Rho factor. (2016, October 19). Acessado em November 20, 2016 Disponível em Wikipedia: https://en.wikipedia.org/wiki/Rho_factor.
- Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). Three eukaryotic RNA polymerases employ different termination mechanisms. Em Molecular cell biology (4th ed., section 11.1). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21601/#_A2857_.
- Richard, P. and Manley, J. L. (2009). Transcription termination by nuclear RNA polymerases. Genes & Dev., 23, 1247-1269. http://genesdev.cshlp.org/content/23/11/1247.full.
Referências:
- 3'-end cleavage and polyadenylation. (2016). In Nobelprize.org. Disponível em http://www.nobelprize.org/educational/medicine/dna/a/splicing/splicing_endformation.html.Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., and Walter, P. (2002). Electron-transport chains and their proton pumps. Em Molecular biology of the cell (4ª ed.). Nova York, NY: Garland Science. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK26904/.Alpha-amanitin. (11 de fevereiro de 2016). Acessado em 13 de fevereiro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Alpha-Amanitin.Amanita phalloides. (2016, February 6). Acessado em 13 de fevereiro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Amanita_phalloides.Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). Transcription is catalyzed by RNA polymerase. In Biochemistry (5th ed., section 28.1). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK22546/.Berger, Shanna. (2006). transcrição eukaryotic. Em Transcription e RNA polimerase II. Disponível em http://www.chem.uwec.edu/Webpapers2006/sites/bergersl/pages/eukaryotic.html.Berger, S. (2006). The mushroom Amanita phalloides. In Transcription and RNA polymerase II. Disponível em http://www.chem.uwec.edu/Webpapers2006/sites/bergersl/pages/amanitin.html.Boundless (2016, January 8). Initiation of transcription in eukaryotes. Em Boundless biology. Disponível em https://www.boundless.com/biology/textbooks/boundless-biology-textbook/genes-and-proteins-15/eukaryotic-transcription-108/initiation-of-transcription-in-eukaryotes-445-11670/.Brown, T. A. (2002). Assembly of the transcription initiation complex. Em Genomes (2nd ed., Ch. 9). Oxford, UK: Wiley-Liss. Disponível em www.ncbi.nlm.nih.gov/books/NBK21115/.Gong, X. Q., Nedialkov, Y. A., and Burton, Z. F. (2004). α-amanitin blocks translocation by human RNA polymerase II. The Journal of Biological Chemistry, 279, 27422-27427. http://dx.doi.org/10.1074/jbc.M402163200.Griffiths, A. J. F., Miller, J. H., Suzuki, D. T., Lewontin, R. C., and Gelbart, W. M. (2000). Using genetic ratios. Em An introduction to genetic analysis (7th ed.). New York, NY: W. H. Freeman. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK21812/.Inverted repeat. (2016, February 13). Acesso em 13 de fevereiro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Inverted_repeat.Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). Bacterial transcription initiation. Em Molecular cell biology (4th ed., section 10.2). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21612/.Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). RNA polymerase II transcription-initiation complex. Em Molecular cell biology (4th ed., section 10.6). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21610/.Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). Transcription termination. Em Molecular cell biology (4th ed., section 11.1). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21601/.Moran, L. A. (2008, September 16). How RNA polymerase binds to DNA [Web log post]. Em Sandwalk: Strolling with a skeptical biochemist. Disponível em http://sandwalk.blogspot.com/2008/09/how-rna-polymerase-binds-to-dna.htmlOpenStax College, Biology. (2016, March 23). Eukaryotic transcription. In OpenStax CNX. Disponível em http://cnx.org/contents/GFy_h8cu@10.8:6l70P9u6@5/Eukaryotic-Transcription.OpenStax College, Concepts of Biology. (2016, October 31). Transcription. In OpenStax CNX. Retirado de http://cnx.org/contents/s8Hh0oOc@9.11:TkuNUJis@3/Transcription.Polyadenylation. (24 de janeiro de 2016). Acesso em 11 de fevereiro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Polyadenylation.Purves, W. K., Sadava, D. E., Orians, G. H., and Heller, H.C. (2004). Transcription: DNA-directed RNA synthesis. Em Life: the science of biology (7th ed., pp. 237-239). Sunderland, MA: Sinauer Associates.Raven, P. H., Johnson, G. B., Mason, K. A., Losos, J. B., and Singer, S. R. (2014). Genes and how they work. Em Biology (10th ed., AP ed., pp. 278-303). New York, NY: McGraw-Hill.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Transcription is the DNA-directed synthesis of RNA: A closer look.. Em Campbell biology (10th ed., pp. 340-342). San Francisco, CA: Pearson.Rho factor. (2016, October 19). Acessado em November 20, 2016 Disponível em Wikipedia: https://en.wikipedia.org/wiki/Rho_factor.Richard, P. and Manley, J. L. (2009). Transcription termination by nuclear RNA polymerases. Genes & Dev., 23, 1247-1269. http://genesdev.cshlp.org/content/23/11/1247.full.Saunders, A., Core, L. J., and Lis, J. T. (2006). Breaking barriers to transcription elongation. Nature Reviews Molecular Cell Biology, 7, 557-567. http://dx.doi.org/10.1038/nrm1981.Terminator (genetics). (2015, December 14). Disponível em 13 de fevereiro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Terminator_%28genetics%29.Webb, S. Witte, L., Wong, K., Woreta, T., and Yoo, E. (2002, May 8). TFIIH. Em RNA polymerase II in eukaryotes and prokaryotes. Disponível em http://www.biochem.umd.edu/biochem/kahn/molmachines/newpolII/TFIIH.html. Processamento de pré-mRNA eucarióta
Pontos Principais:
- Logo que um transcrito de RNA é produzido em uma célula eucarionte, ele é considerado um pré-RNAm e deve ser processado para formar RNA mensageiro (RNAm).
- Um cap 5' é adicionado ao começo do transcrito de RNA, e uma cauda poli-A é adicionada ao final.
- No splicing, algumas seções do transcrito de RNA (íntrons) são removidas, e as seções restantes (éxons) são acopladas novamente.
- Alguns genes podem sofrer splicing alternativo, levando à produção de diferentes moléculas maduras de RNAm a partir do mesmo transcrito inicial.
Introdução
- Imagine que você gerencie uma fábrica de livros e acabou de imprimir todas as páginas de seu livro predileto. Agora que você tem as páginas, o livro já está pronto para a venda? Bem... os livros costumam ter capa na frente e atrás. Então talvez você queira colocá-las. Além disso, havia alguma página em branco ou danificada feita durante a impressão? Você provavelmente deveria checar e, se houver, removê-las antes de vender seu livro, ou você pode acabar se deparando com uns clientes insatisfeitos.Os passos sobre os quais acabamos de falar são bastante parecidos com o que acontece a transcritos de RNA nas células do seu corpo. Nos humanos e outros eucariontes, uma molécula de RNA recém-fabricada (saída quentinha das "prensas" de RNA polimerase) não está exatamente pronta. Ao invés disso, é chamada de pré-RNAm e tem que passar por algumas etapas de processamento para se tornar um RNA mensageiro (RNAm) maduro que pode ser traduzido em uma proteína. Essas incluem:
- Adição de moléculas cap e cauda nas duas extremidades do transcrito. Elas exercem um papel de proteção, como as capas dianteira e traseira de um livro.
- Remoção de sequências "lixo" chamadas íntrons. Íntrons são como páginas em branco ou danificadas feitas durante a impressão do livro, que têm de ser removidas para que o livro se torne legível.
Neste artigo, olharemos mais de perto as modificações cap, cauda e splicing que os transcritos eucariontes de RNA recebem, vendo como são conduzidos e por que são tão importantes para que consigamos a proteína correta do nosso RNA.
Visão geral do processamento do pré-RNAm em eucariontes
- Como uma revisão rápida, a expressão genética (a "leitura" de um gene para se fazer uma proteína ou o pedaço de uma proteína) acontece um pouco diferente nas bactérias e nos eucariontes, como os humanos.
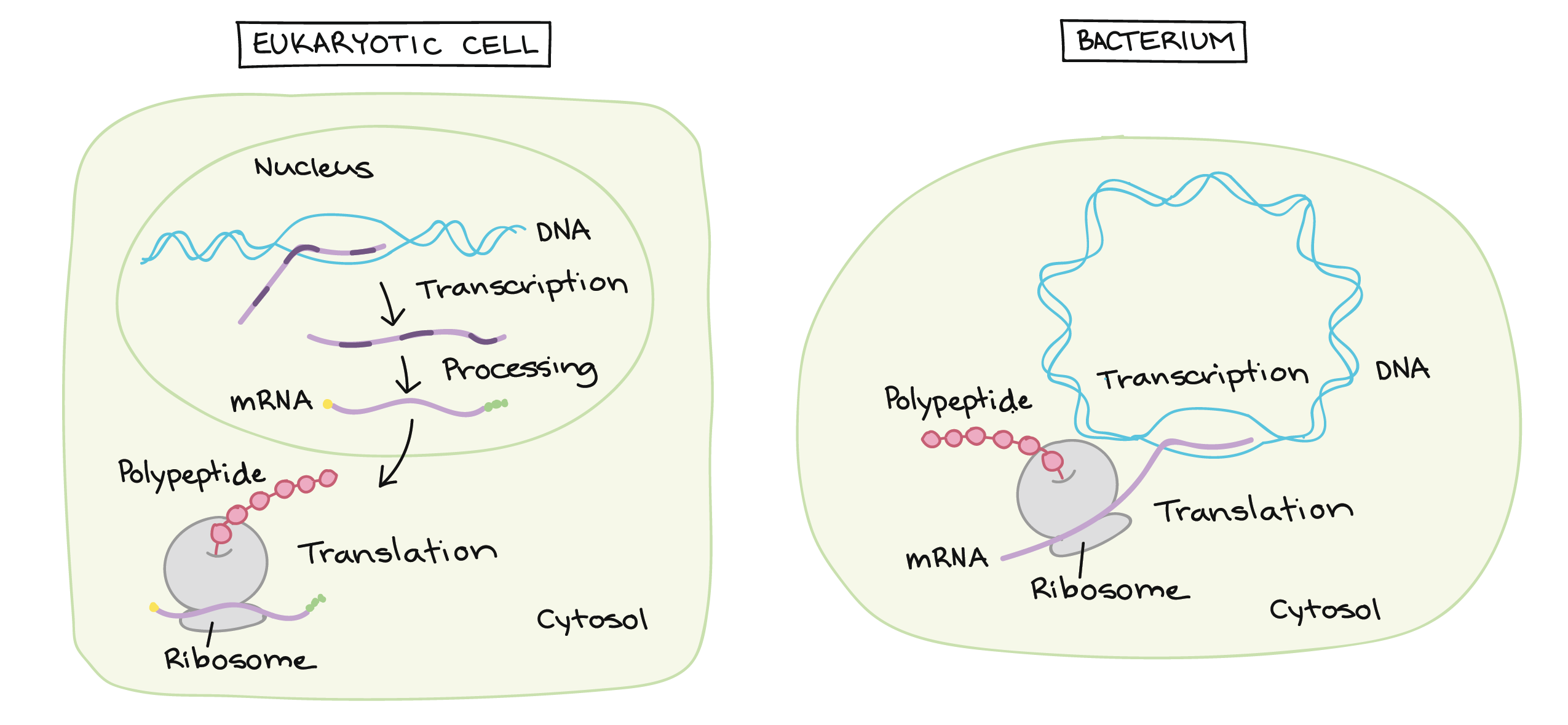 Painel esquerdo: célula eucariótica. No núcleo, um pré-RNAm é produzido por meio da transcrição de uma região do DNA de um cromossomo linear. Esse transcrito precisa passar por processamento (splicing e adição do cap 5' e da cauda poli-A) enquanto ainda está no núcleo para que possa se tornar um RNAm maduro. O RNAm maduro é exportado do núcleo ao citosol, onde é traduzido num ribossomo para produzir um polipeptídeo.Painel esquerdo: bactéria. O DNA assume a forma de um cromossomo circular e se localiza no citosol. Enquanto o DNA é transcrito para fazer um RNA, o RNA (que neste ponto já pode ser considerado um RNAm) pode se associar a um ribossomo e começar a ser traduzido para produzir um polipeptídeo.Na bactéria, os transcritos de RNA estão prontos para atuar como RNAs mensageiros e serem logo traduzidos em proteínas. Em eucariontes, as coisas são um pouco mais complexas, embora aconteça de uma forma bem interessante. A molécula diretamente feita por transcrição em uma de suas células (eucariontes) é chamada de pré-RNAm, refletindo que ela precisa passar por mais alguns passos para se tornar de fato um RNA mensageiro (RNAm). Esses são:
Painel esquerdo: célula eucariótica. No núcleo, um pré-RNAm é produzido por meio da transcrição de uma região do DNA de um cromossomo linear. Esse transcrito precisa passar por processamento (splicing e adição do cap 5' e da cauda poli-A) enquanto ainda está no núcleo para que possa se tornar um RNAm maduro. O RNAm maduro é exportado do núcleo ao citosol, onde é traduzido num ribossomo para produzir um polipeptídeo.Painel esquerdo: bactéria. O DNA assume a forma de um cromossomo circular e se localiza no citosol. Enquanto o DNA é transcrito para fazer um RNA, o RNA (que neste ponto já pode ser considerado um RNAm) pode se associar a um ribossomo e começar a ser traduzido para produzir um polipeptídeo.Na bactéria, os transcritos de RNA estão prontos para atuar como RNAs mensageiros e serem logo traduzidos em proteínas. Em eucariontes, as coisas são um pouco mais complexas, embora aconteça de uma forma bem interessante. A molécula diretamente feita por transcrição em uma de suas células (eucariontes) é chamada de pré-RNAm, refletindo que ela precisa passar por mais alguns passos para se tornar de fato um RNA mensageiro (RNAm). Esses são:- Adição de um cap 5' ao início do RNA
- Adição de uma cauda poli-A (cauda de nucleotídeos A) ao final do RNA.
- Separação dos íntrons ou "sequências lixo" e conexão do que sobrou , das sequências boas (os éxons).
Uma vez que esses passos estão completos, o RNA se torna um RNAm maduro. Ele pode sair do núcleo e ser usado para fazer uma proteína.
Cap 5' e a cauda poli-A
- Ambas extremidades finais do pre-RNAm são modificadas pela adição de grupos químicos. O grupo do início (final 5') é chamado de cap, enquanto o grupo de terminação (final 3') é chamado de cauda. Tanto o cap quanto a cauda protegem o transcrito e ajudam-no a ser exportado do núcleo e a ser traduzido nos ribossomos ("máquinas" de fazer proteínas) encontrados no citosol1start superscript, 1, end superscript.A cap 5' é adicionada ao primeiro nucleotídeo do transcrito durante a transcrição. A cap é uma guanina (G) modificada e protege o transcrito de ser quebrado. Ela também auxilia o ribossomo a se ligar ao RNAm e começar a leitura para fazer uma proteína.
 Imagem de um pré-RNAm com um cap 5' e uma cauda poli-A 3'. O cap 5' está na extremidade 5' do pré-RNAm e é um nucleotídeo G modificado. A cauda poli-A está na extremidade 3' do pré-RNAm e consiste em uma longa linha de nucleotídeos A (dos quais apenas alguns são mostrados).Como a cauda poli-A é adicionada? O final 3' do RNA se forma de um jeito um pouco bizarro. Quando uma sequência chamada sinal de poliadenilação aparece em uma molécula de RNA durante a transcrição, uma enzima corta o RNA em dois naquele ponto. Outra enzima adiciona aproximadamente 100100
Imagem de um pré-RNAm com um cap 5' e uma cauda poli-A 3'. O cap 5' está na extremidade 5' do pré-RNAm e é um nucleotídeo G modificado. A cauda poli-A está na extremidade 3' do pré-RNAm e consiste em uma longa linha de nucleotídeos A (dos quais apenas alguns são mostrados).Como a cauda poli-A é adicionada? O final 3' do RNA se forma de um jeito um pouco bizarro. Quando uma sequência chamada sinal de poliadenilação aparece em uma molécula de RNA durante a transcrição, uma enzima corta o RNA em dois naquele ponto. Outra enzima adiciona aproximadamente 100100- 200200 nucleotídeos de adenina (A) para cotar o final, formando uma cauda poli-A. A cauda torna o transcrito mais estável e ajuda a exportá-lo do núcleo para o citosol.
Splicing do RNA
- O terceiro grande evento do processamento de RNA que ocorre nas suas células é o splicing do RNA. No splicing do RNA, partes específicas do pré-RNAm, chamadas íntrons são reconhecidas e removidas por complexos proteína-e-RNA chamados de spliceossomos. Os íntrons podem ser vistos como sequências "lixo" que devem ser retiradas para que a "versão de partes boas" da molécula de RNA possa ser montada.O que são as "partes boas"? As partes do RNA que não são cortadas são chamadas exons. Os exons são colocados juntos pelo spliceossomo para formar o RNAm maduro final, que é enviado para fora do núcleo.
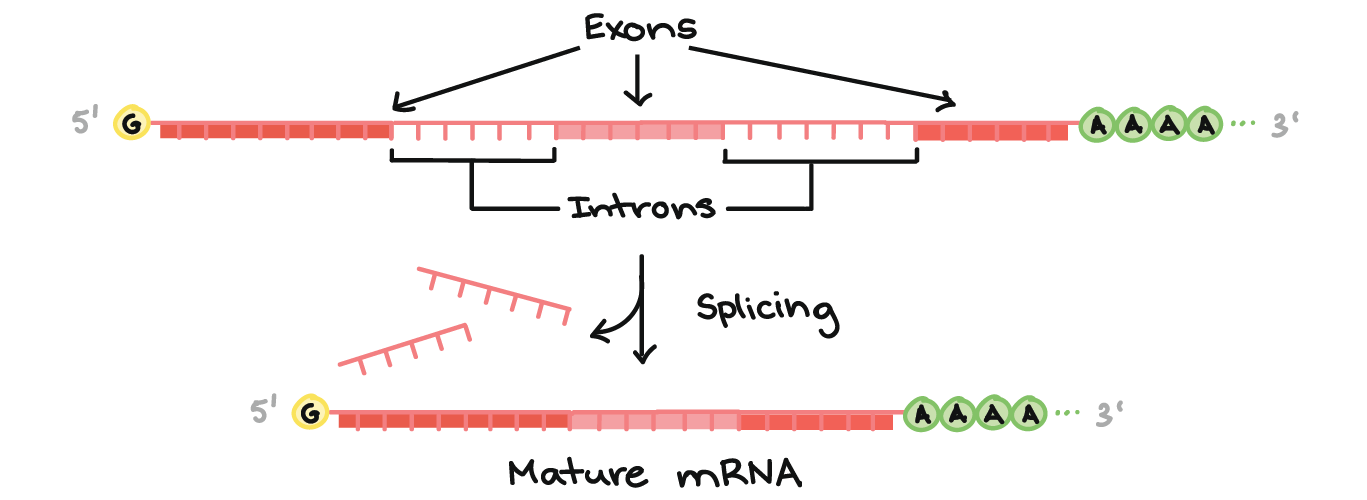 Diagrama de um pré-RNAm mostrando éxons e íntrons. Ao longo do comprimento do RNAm, há um padrão alternante de éxons e íntrons: Éxon 1 - Íntron 1 - Éxon 2 - Íntron 2 - Éxon 3. Cada um consiste em um trecho de nucleotídeos de RNA. Durante o splicing, os íntrons são removidos do pré-RNAm, e os éxons são acoplados para formar o RNAm maduro que não contém as sequências de íntron.Um ponto-chave aqui é que são somente os éxons de um gene que codificam uma proteína. Não só os íntrons não carregam informações para construir uma proteína, como eles realmente têm que ser removidos para que o mRNA codifique uma proteína com a sequência certa. Se o spliceossomo falha ao remover um íntron, um mRNA com "lixo" extra será feito, e uma proteína errada vai ser produzida durante a tradução.
Diagrama de um pré-RNAm mostrando éxons e íntrons. Ao longo do comprimento do RNAm, há um padrão alternante de éxons e íntrons: Éxon 1 - Íntron 1 - Éxon 2 - Íntron 2 - Éxon 3. Cada um consiste em um trecho de nucleotídeos de RNA. Durante o splicing, os íntrons são removidos do pré-RNAm, e os éxons são acoplados para formar o RNAm maduro que não contém as sequências de íntron.Um ponto-chave aqui é que são somente os éxons de um gene que codificam uma proteína. Não só os íntrons não carregam informações para construir uma proteína, como eles realmente têm que ser removidos para que o mRNA codifique uma proteína com a sequência certa. Se o spliceossomo falha ao remover um íntron, um mRNA com "lixo" extra será feito, e uma proteína errada vai ser produzida durante a tradução.
Splicing alternativo
- Por que splicing? Não sabemos com certeza por que o splicing existe e, de alguma maneira, ele parece um sistema dispendioso. Contudo, o splicing permite um processo chamado splicing alternativo, em que mais de um RNAm pode ser formado a partir do mesmo gene. Através do splicing alternativo, nós (e outros eucariontes) podemos sorrateiramente codificar mais proteínas diferentes do que temos de genes em nosso DNA.No splicing alternativo, um pré-RNAm pode sofrer splicing em qualquer uma de duas maneiras (ou algumas vezes muito mais que duas!). Por exemplo, no diagrama abaixo, o mesmo pré-RNAm pode sofrer splicing de três maneiras diferentes, dependendo de quais éxons sejam mantidos. Isso resulta em três RNAm maduros, cada qual é traduzido em uma proteína de estrutura diferente.
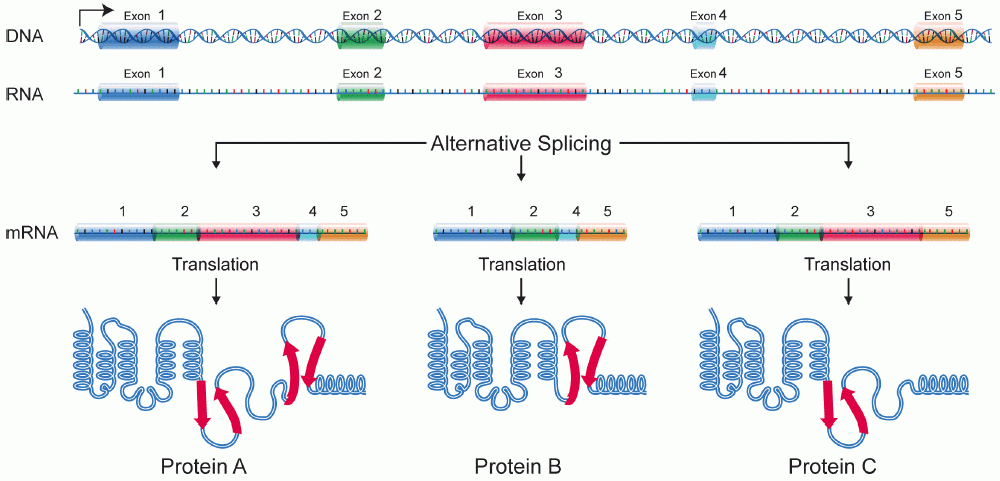 Diagrama do splicing alternativo.Uma sequência de DNA codifica um transcrito de pré-RNAm que contém cinco áreas que potencialmente podem ser usadas como éxons: Éxon 1, Éxon 2, Éxon 3, Éxon 4 e Éxon 5. Os éxons são arranjados em ordem linear ao longo do pré-RNAm e têm íntrons entre eles.No evento de splicing #1, todos os cinco éxons são mantidos no RNAm maduro. Ele consiste em Éxon 1 - Éxon 2 - Éxon 3 - Éxon 4 - Éxon 5. Quando ele é traduzido, ele especifica a Proteína A, uma proteína com cinco domínios: Mola1 (especificada pelo Éxon 1), Mola 2 (especificada pelo Éxon 2), Loop 3 (especificado pelo Éxon 3), Loop 4 (especificado pelo Éxon 4), e Mola 5 (especificado pelo Éxon 5).No evento de splicing #2, Éxon 3 não é incluído no RNAm maduro. Ele consiste em Éxon 1 - Éxon 2 - Éxon 4 - Éxon 5. Quando traduzido, ele especifica a Proteína, B uma proteína com cinco domínios: Mola 1 (especificada pelo Éxon 1), Mola 2 (especificada pelo Éxon 2), Loop 4 (especificado pelo Éxon 4), e Mola 5 (especificada pelo Éxon 5). Ela não contém o Loop 3 porque o Éxon 3 não está presente no RNAm.No evento de splicing #3, o Éxon 4 não está incluído no RNAm maduro. Ele consiste do Éxon 1 - Éxon 2 - Éxon 3 - Éxon 5. Quando traduzido ele especifica a Proteína C, uma proteína com quatro domínios: Mola 1 (especificada pelo Éxon 1), Mola 2 (especificada pelo Éxon 2), Loop 3 (especificado pelo Éxon 3), e Mola 5 (especificada pelo Éxon 5). Ela não contém o Loop 4 porque o Éxon 4 não está presente no RNAm._Crédito da imagem: "DNA, alternative splicing," pelo National Human Genome Research Institute (public domain)._
Diagrama do splicing alternativo.Uma sequência de DNA codifica um transcrito de pré-RNAm que contém cinco áreas que potencialmente podem ser usadas como éxons: Éxon 1, Éxon 2, Éxon 3, Éxon 4 e Éxon 5. Os éxons são arranjados em ordem linear ao longo do pré-RNAm e têm íntrons entre eles.No evento de splicing #1, todos os cinco éxons são mantidos no RNAm maduro. Ele consiste em Éxon 1 - Éxon 2 - Éxon 3 - Éxon 4 - Éxon 5. Quando ele é traduzido, ele especifica a Proteína A, uma proteína com cinco domínios: Mola1 (especificada pelo Éxon 1), Mola 2 (especificada pelo Éxon 2), Loop 3 (especificado pelo Éxon 3), Loop 4 (especificado pelo Éxon 4), e Mola 5 (especificado pelo Éxon 5).No evento de splicing #2, Éxon 3 não é incluído no RNAm maduro. Ele consiste em Éxon 1 - Éxon 2 - Éxon 4 - Éxon 5. Quando traduzido, ele especifica a Proteína, B uma proteína com cinco domínios: Mola 1 (especificada pelo Éxon 1), Mola 2 (especificada pelo Éxon 2), Loop 4 (especificado pelo Éxon 4), e Mola 5 (especificada pelo Éxon 5). Ela não contém o Loop 3 porque o Éxon 3 não está presente no RNAm.No evento de splicing #3, o Éxon 4 não está incluído no RNAm maduro. Ele consiste do Éxon 1 - Éxon 2 - Éxon 3 - Éxon 5. Quando traduzido ele especifica a Proteína C, uma proteína com quatro domínios: Mola 1 (especificada pelo Éxon 1), Mola 2 (especificada pelo Éxon 2), Loop 3 (especificado pelo Éxon 3), e Mola 5 (especificada pelo Éxon 5). Ela não contém o Loop 4 porque o Éxon 4 não está presente no RNAm._Crédito da imagem: "DNA, alternative splicing," pelo National Human Genome Research Institute (public domain)._
Tente você mesmo: Faça o splicing da mensagem
- Sua missão, caso você a aceite: decodificar a seguinte mensagem ultra-secreta. Primeiro, retire as letras "inúteis", coloridas de roxo e sublinhadas. Segundo, arranje as letras restantes em grupos de três, iniciando no começo.THEDOGRstart color #ca337c, start bold text, T, H, E, D, O, G, R, end bold text, end color #ca337cAMAPQstart color #aa87ff, start underline, start text, A, M, A, P, Q, end text, end underline, end color #aa87ffANANDAstart color #ca337c, start bold text, A, N, A, N, D, A, end bold text, end color #ca337cZAPTQMstart color #aa87ff, start underline, start text, Z, A, P, T, Q, M, end text, end underline, end color #aa87ffTETHEHATstart color #ca337c, start bold text, T, E, T, H, E, H, A, T, end bold text, end color #ca337cVocê tentou?
- Se você remover as sequências em roxo, você deve obter esta série de letras:
- Se você agrupar as letras restantes em sequências de três, você deverá obter esta mensagem:
O processo pelo qual você acabou de passar é basicamente o que as suas células têm que fazer quando elas expressam um gene. Como discutimos anteriormente neste artigo, a maioria dos pré-RNAm eucariontes contêm sequências "lixo" chamadas íntrons, que são como as letras roxas na mensagem. Essas sequências devem ser removidas, e as sequências significativas (éxons), equivalentes às letras em vinho na mensagem acima, devem ser acopladas novamente para formar um RNAm maduro.Durante a tradução, a sequência de RNAm é lida em grupos de três nucleotídeos. Cada "palavra" de três letras corresponde a um aminoácido que é adicionado a um polipeptídeo (proteína ou subunidade de proteína). Se um RNAm não tiver sofrido splicing, ele irá conter nucleotídeos a mais que não deveria, levando a uma "mensagem" de proteína incorreta. Algo similar acontece se tentamos decodificar a mensagem acima sem remover as letras roxas:THE DOG Rstart color #ca337c, start bold text, T, H, E, space, D, O, G, space, R, end bold text, end color #ca337cAM APQstart color #aa87ff, start underline, start text, A, M, space, A, P, Q, end text, end underline, end color #aa87ff ANA NDAstart color #ca337c, start bold text, space, A, N, A, space, N, D, A, end bold text, end color #ca337c ZAP TQM start color #aa87ff, start underline, start text, space, Z, A, P, space, T, Q, M, space, end text, end underline, end color #aa87ffTET HEH ATstart color #ca337c, start bold text, T, E, T, space, H, E, H, space, A, T, end bold text, end color #ca337cAssim como a remoção das letras roxas é essencial para se chegar à mensagem correta, o splicing também é essencial para garantir que o RNAm carregue a informação correta (e direcione a produção do polipeptídeo correto).Aqui está a sequência de pré-RNAm, com éxons em vinho vivo e um íntron em letras roxas sublinhadas:5’-...AUGCACUAUstart text, 5, apostrophe, negative, point, point, point, end text, start color #ca337c, start bold text, A, U, G, C, A, C, U, A, U, end bold text, end color #ca337cGUAUGUGCCUUUUUGUGCUGUGstart color #aa87ff, start underline, start text, G, U, A, U, G, U, G, C, C, U, U, U, U, U, G, U, G, C, U, G, U, G, end text, end underline, end color #aa87ffGAAGCGUAA...-3’start color #ca337c, start bold text, G, A, A, G, C, G, U, A, A, end bold text, end color #ca337c, start text, point, point, point, negative, 3, apostrophe, end textSe o pré-RNAm sofrer splicing corretamente, os nucleotídeos do RNAm maduro são lidos em grupos de três como se segue, produzindo o polipeptídeo mostrado abaixo:5’-...AUG CAC UAU start text, 5, apostrophe, negative, point, point, point, end text, start color #ca337c, start bold text, A, U, G, space, C, A, C, space, U, A, U, space, end bold text, end color #ca337cGAA GCG UAA...-3’start color #ca337c, start bold text, G, A, A, space, G, C, G, space, U, A, A, end bold text, end color #ca337c, start text, point, point, point, negative, 3, apostrophe, end textMet - His - Tyr - Glu - Ala - STOPstart text, M, e, t, space, negative, space, H, i, s, space, negative, space, T, y, r, space, negative, space, G, l, u, space, negative, space, A, l, a, space, negative, space, S, T, O, P, end textSe o íntron não for removido do pré-RNAm, o RNAm anormal resultante conterá nucleotídeos a mais. Quando seus nucleotídeos forem lidos em grupos de três, como mostrado abaixo, um polipeptídeo com uma sequência incorreta de aminoácidos será produzido. Há aminoácidos em excesso, e os aminoácidos especificados pelo segundo éxon não serão produzidos (porque a janela de leitura está deslocada):5’-...AUG CAC UAU start text, 5, apostrophe, negative, point, point, point, end text, start color #ca337c, start bold text, A, U, G, space, C, A, C, space, U, A, U, space, end bold text, end color #ca337cGUA UGU GCC UUU UUG UGC UGU Gstart color #aa87ff, start underline, start text, G, U, A, space, U, G, U, space, G, C, C, space, U, U, U, space, U, U, G, space, U, G, C, space, U, G, U, space, G, end text, end underline, end color #aa87ffGA AGC GUA A...-3’start color #ca337c, start bold text, G, A, space, A, G, C, space, G, U, A, space, A, end bold text, end color #ca337c, start text, point, point, point, negative, 3, apostrophe, end textMet - His - Tyr - Val - Cys - Ala - Phe - Leu - Cys - Cys - Gly - Ser - Val ...start text, M, e, t, space, negative, space, H, i, s, space, negative, space, T, y, r, space, negative, space, V, a, l, space, negative, space, C, y, s, space, negative, space, A, l, a, space, negative, space, P, h, e, space, negative, space, L, e, u, space, negative, space, C, y, s, space, negative, space, C, y, s, space, negative, space, G, l, y, space, negative, space, S, e, r, space, negative, space, V, a, l, space, point, point, point, end textTodas as relações acima entre grupos de nucleotídeos e os aminoácidos que eles codificam podem ser determinadas usando a tabela do código genético: _Crédito da imagem: "O código genético: Figura 3," por OpenStax College, Biology (CC BY 3.0)._Sequência de íntron modificada de Bourdin et al.4start superscript, 4, end superscript. Este artigo está autorizado sob licença CC BY-NC-SA 4.0.
_Crédito da imagem: "O código genético: Figura 3," por OpenStax College, Biology (CC BY 3.0)._Sequência de íntron modificada de Bourdin et al.4start superscript, 4, end superscript. Este artigo está autorizado sob licença CC BY-NC-SA 4.0.
Referências:
- Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Alteration of mRNA ends. Em Campbell biology (10th ed., p. 342-343). San Francisco, CA: Pearson.
- Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). The functional and evolutionary importance of introns. Em Campbell biology (10th ed., p. 344). San Francisco, CA: Pearson.
- Keren, H., Lev-Maor, G., and Ast, G. (2010). Alternative splicing and evolution: diversification, exon definition and function. Nature Reviews Genetics, 11, 345-355. http://dx.doi.org/10.1038/nrg2776. Disponível em https://www.tau.ac.il/~gilast/PAPERS/nrg2776.pdf.
- Standage, D. (2012, April 8). Why do eukaryotic organisms have introns in their DNA? [Answer] Em Biology stack exchange. Disponível em http://biology.stackexchange.com/questions/1724/why-do-eukaryotic-organisms-have-introns-in-their-dna.
- Bourdin, C. M., Moignot, B., Wang, L., Murillo, L., Juchaux, M., Quinchard, S., Lapied, B., Guérineau, N. C., Dong, K., and Legros, C. (2013). Intron retention in mRNA encoding ancillary subunit of insect voltage-gated sodium channel modulates channel expression, gating regulation and drug sensitivity. PLoS ONE, 8(8), e67290. http://dx.doi.org/10.1371/journal.pone.0067290.
Outras referências
- 3'-end cleavage and polyadenylation. (2016). In Nobelprize.org. Disponível em http://www.nobelprize.org/educational/medicine/dna/a/splicing/splicing_endformation.html.Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., and Walter, P. (2002). Electron-transport chains and their proton pumps. Em Molecular biology of the cell (4ª ed.). Nova York, NY: Garland Science. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK26904/.Intron/introns. (2014). In Scitable. Disponível em http://www.nature.com/scitable/definition/intron-introns-67.Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). Processing of eukaryotic mRNA. Em Molecular cell biology (4th ed., section 11.2). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21563/.Lodish, H., Berk, A., Zipursky, S. L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000). Transcription termination. Em Molecular cell biology (4th ed., section 11.1). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK21601/.Polyadenylation. (24 de janeiro de 2016). Acesso em 11 de fevereiro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Polyadenylation.Raven, P. H., Johnson, G. B., Mason, K. A., Losos, J. B., and Singer, S. R. (2014). Genes and how they work. Em Biology (10th ed., AP ed., pp. 278-303). New York, NY: McGraw-Hill.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Eukaryotic cells modify RNA after transcription . Em Campbell biology (10th ed., p. 342-345). San Francisco, CA: Pearson. tRNAs e ribossomos
Introdução
- A Tradução requer alguns equipamentos especializados. Assim como não jogaríamos tênis sem as raquetes e as bolas, a célula não poderia traduzir um RNAm em proteína sem duas peças de engrenagem molecular: RNAt e ribossomos.
- Ribossomos fornecem a estrutura na qual a tradução pode acontecer. Eles também catalisam a reação que liga aminoácidos para formar uma nova proteína.
- RNAts (RNAs transportadores) transportam aminoácidos para o ribossomo. Eles atuam como "pontes", combinando um códon em um RNAm com o aminoácido que o códon especifica.
Aqui daremos uma olhada mais de perto nos ribossomos e RNAts. Se você ainda não está familiarizado com o RNA (sigla para ácido ribonucleico, do inglês), eu recomendo fortemente que cheque a seção sobre ácidos nucleicos primeiro, para que possa tirar o máximo deste artigo.
Ribossomos: onde a tradução acontece
- A tradução ocorre dentro de estruturas chamadas ribossomos, os quais são feitos de RNA e proteínas. Ribossomos organizam a tradução e catalisam a reação que une os aminoácidos para formar uma cadeia proteica.
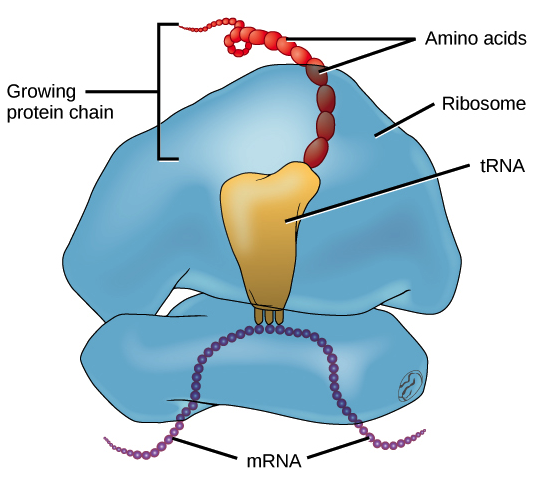 Ilustração das moléculas envolvidas na tradução de proteínas. Um ribossomo é mostrado com RNAm e RNAt. Aminoácidos emergem para formar uma proteína.
Ilustração das moléculas envolvidas na tradução de proteínas. Um ribossomo é mostrado com RNAm e RNAt. Aminoácidos emergem para formar uma proteína.
Estrutura do ribossomo
- Um ribossomo é feito de dois componentes básicos: uma subunidade grande e uma pequena. Durante a tradução as duas subunidades juntam-se ao redor de uma molécula de RNAm, formando um ribossomo completo. O ribossomo se move à frente pelo RNAm, códon a códon, conforme o lê e traduz em um polipeptídeo (cadeia de proteína). Então, uma vez a tradução finalizada, os dois componentes separam-se novamente e podem ser reusados.Em geral o ribossomo é um terço proteico e dois terços RNA ribossômico (RNAr). Os RNArs são aparentemente responsáveis pela maior parte da estrutura e função do ribossomo, enquanto as proteínas ajudam-no a mudar sua conformação enquanto ele cataliza reações químicas1start superscript, 1, end superscript.Abaixo, pode-se observar um modelo tridimensional de ribossomo. Proteínas estão representadas em azul, enquanto cadeias de RNAr estão representadas em laranja e pêssego. O ponto verde marca o sítio ativo, que cataliza a reação que liga aminoácidos para fazer uma proteína. Me surpreende o ribossomo ser tão rugoso, meio que como a superfície do cérebro!
O ribossomo possui sítios para os RNAts
- Como vimos brevemente na introdução, moléculas chamadas RNAs transportadores (RNAts) trazem aminoácidos aos ribossomos. Aprenderemos muito mais a respeito dos RNAs transportadores e sobre como eles funcionam na próxima sessão.Por ora, apenas tenha em mente que o ribossomo possui três espaços para RNAts: o sítio A, o sítio P e o sítio E. RNAts se movem através destes sítios (do A ao P ao E) enquanto entregam aminoácidos durante a tradução.
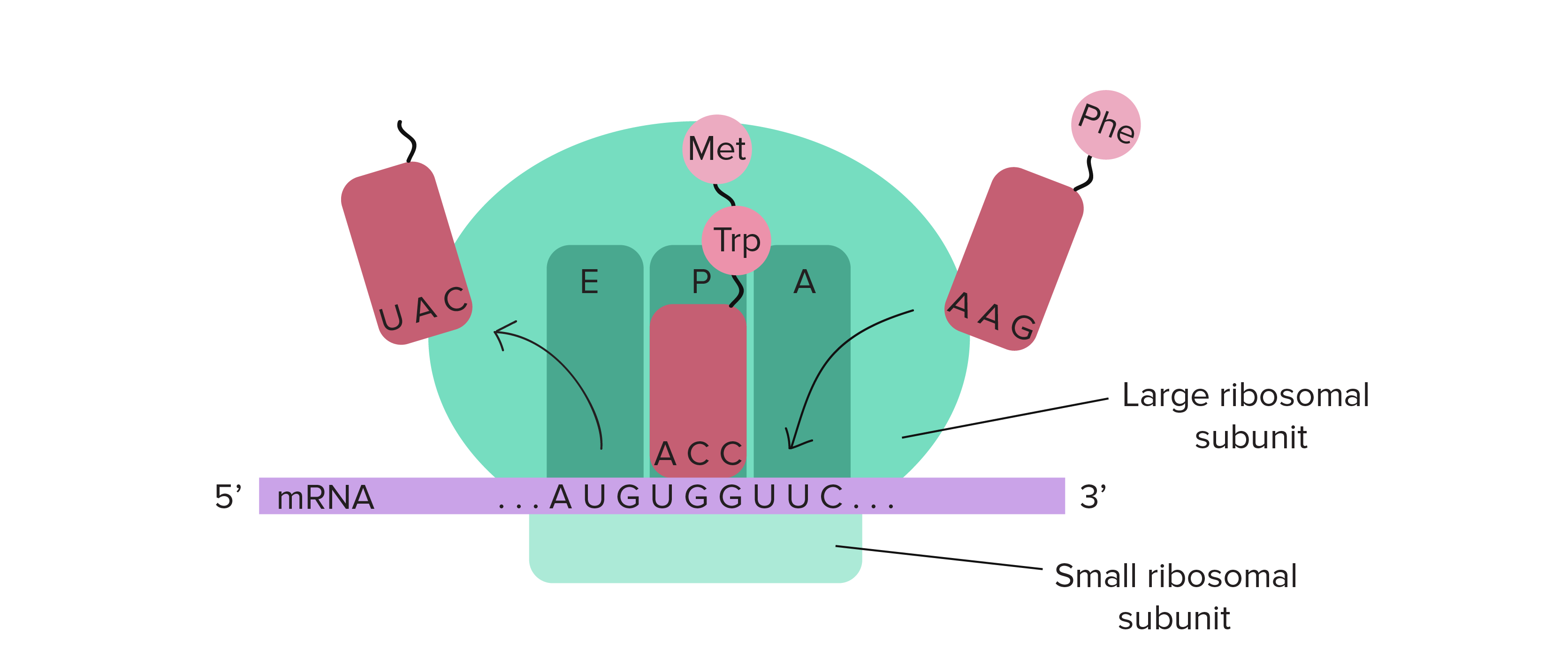 O ribossomo é constituído de uma subunidade grande e outra pequena. A subunidade pequena se liga a um transcrito de RNAm e ambas as subunidades se reúnem para fornecer três sítios para RNAts (sítio A, sítio P e sítio E). No diagrama, os sítios A, P e E aparecem na ordem A-P-E da direita para a esquerda.Após a ligação inicial do primeiro RNAt no sítio P, um RNAt carregado, de chegada, vai se ligar ao sítio A. A formação de uma ligação peptídica vai transferir o aminoácido do primeiro RNAt (Met) para o aminoácido do segundo RNAt (nesse caso, Trp). Essa cadeia de dois aminoácidos será ligada ao RNAt no sítio A. O ribossomo vai avançar um códon no molde de RNAm. O RNAt no sítio A (com a cadeia polipeptídica) vai se deslocar para o sítio P, e o RNAt vazio, anteriormente no sítio P, vai se deslocar para o sítio E (por onde vai sair do ribossomo). Um novo RNAt (nesse caso, um carregando Phe) vai se ligar ao códon recém exposto no sítio A, e assim o processo pode se repetir.Imagem modificada de "Tradução: Figura 3," by OpenStax College, Biology (CC BY 4.0).Para aprender mais sobre o papel específico de cada um dos sítios, visite o artigo sobre estágios da tradução.
O ribossomo é constituído de uma subunidade grande e outra pequena. A subunidade pequena se liga a um transcrito de RNAm e ambas as subunidades se reúnem para fornecer três sítios para RNAts (sítio A, sítio P e sítio E). No diagrama, os sítios A, P e E aparecem na ordem A-P-E da direita para a esquerda.Após a ligação inicial do primeiro RNAt no sítio P, um RNAt carregado, de chegada, vai se ligar ao sítio A. A formação de uma ligação peptídica vai transferir o aminoácido do primeiro RNAt (Met) para o aminoácido do segundo RNAt (nesse caso, Trp). Essa cadeia de dois aminoácidos será ligada ao RNAt no sítio A. O ribossomo vai avançar um códon no molde de RNAm. O RNAt no sítio A (com a cadeia polipeptídica) vai se deslocar para o sítio P, e o RNAt vazio, anteriormente no sítio P, vai se deslocar para o sítio E (por onde vai sair do ribossomo). Um novo RNAt (nesse caso, um carregando Phe) vai se ligar ao códon recém exposto no sítio A, e assim o processo pode se repetir.Imagem modificada de "Tradução: Figura 3," by OpenStax College, Biology (CC BY 4.0).Para aprender mais sobre o papel específico de cada um dos sítios, visite o artigo sobre estágios da tradução.
O que exatamente é um RNAt?
- Um RNA transportador (RNAt) é um tipo especial de molécula de RNA. Seu trabalho é parear um códon de RNAm com o aminoácido que ele codifica. Você pode imaginá-lo como uma "ponte molecular" entre os dois.Cada RNAt contém um conjunto de três nucleotídeos chamado de anticódon. O anticódon de um dado RNAt pode se ligar a um ou alguns códons específicos de RNAm. A molécula de RNAt também carrega um aminoácido: especificamente, aquele codificado pelos códons com os quais o RNAt se liga.
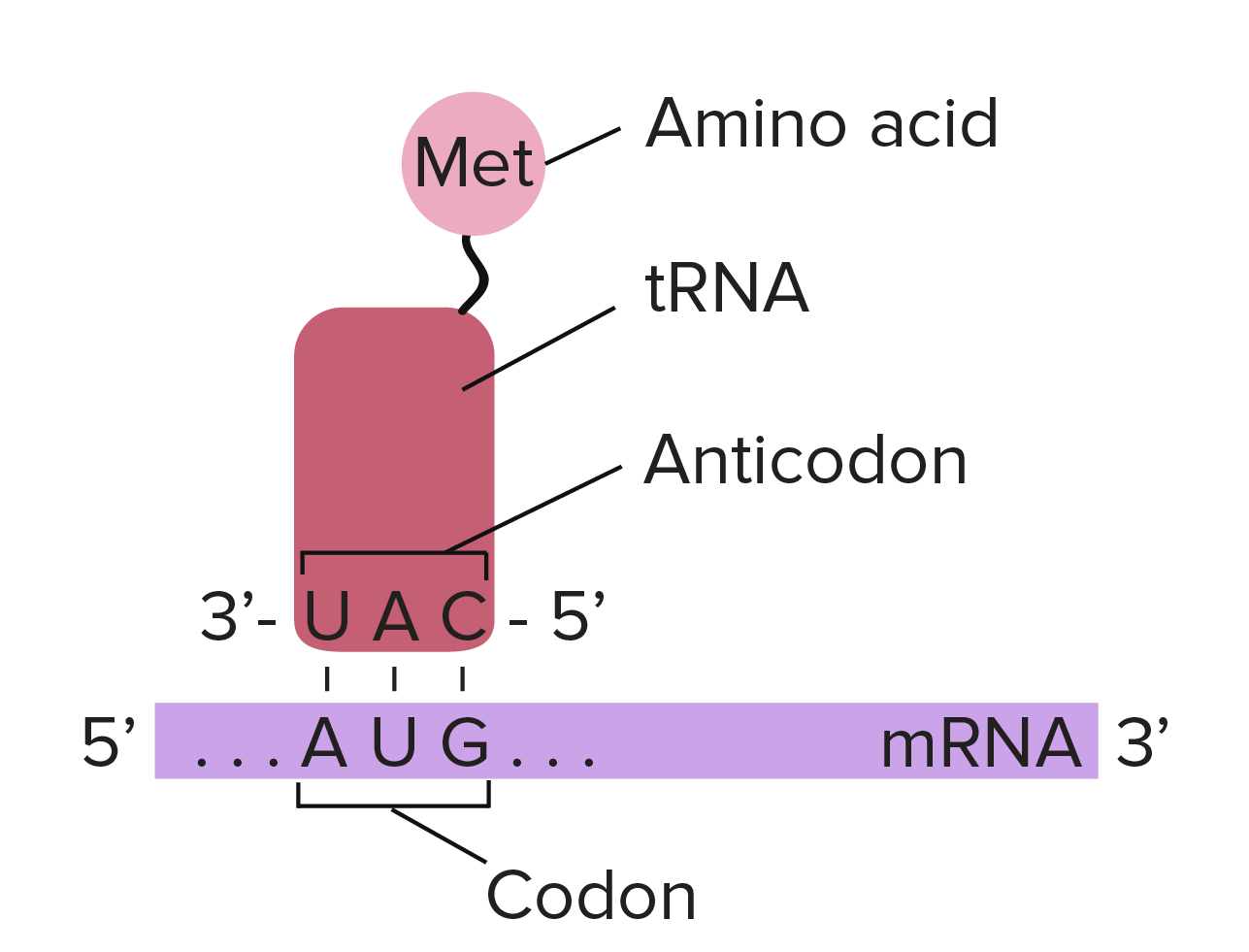 Imagem mostrando um RNAt atuando como um adaptador que conecta um códon de RNAm a um aminoácido . Em uma extremidade, o RNAt tem um anticódon de 3'-UAC-5', ele se liga a um códon no RNAm que contém uma sequência 5'-AUG-3', através do pareamento de bases complementares. A outra extremidade do RNAt carrega um aminoácido metionina (Met), que é o aminoácido especificado pelo códon AUG do RNAm.Imagem modificada de "Tradução: Figura 3," by OpenStax College, Biology (CC BY 4.0).Existem diferentes tipos de RNAts dispersos pelo citoplasma de uma célula, cada um com seu próprio anticódon e aminoácido correspondente. Na verdade, há usualmente de 4040 a 6060 tipos diferentes, o que depende da espécie3cubed. Os RNAts se ligam aos códons dentro do ribossomo, onde eles entregam os aminoácidos que são adicionados à cadeia proteica.
Imagem mostrando um RNAt atuando como um adaptador que conecta um códon de RNAm a um aminoácido . Em uma extremidade, o RNAt tem um anticódon de 3'-UAC-5', ele se liga a um códon no RNAm que contém uma sequência 5'-AUG-3', através do pareamento de bases complementares. A outra extremidade do RNAt carrega um aminoácido metionina (Met), que é o aminoácido especificado pelo códon AUG do RNAm.Imagem modificada de "Tradução: Figura 3," by OpenStax College, Biology (CC BY 4.0).Existem diferentes tipos de RNAts dispersos pelo citoplasma de uma célula, cada um com seu próprio anticódon e aminoácido correspondente. Na verdade, há usualmente de 4040 a 6060 tipos diferentes, o que depende da espécie3cubed. Os RNAts se ligam aos códons dentro do ribossomo, onde eles entregam os aminoácidos que são adicionados à cadeia proteica.
Alguns RNAt se ligam a diferentes códons ("wobble ou oscilação")
- Alguns tRNAs conseguem formar pares de bases com mais de um códon. De primeira, isso parece bastante estranho: o A não é o par de base de U e G de C?Bem... nem sempre. (A biologia é uma caixinha de surpresas, não é?) Pares de bases atípicos —entre outros nucleotídeos que não A-U e G-C— podem se formar na terceira posição do códon, um fenômeno conhecido como oscilação (do inglês - wobble).O pareamento oscilante não segue as regras normais, mas ele possui as suas próprias. Por exemplo, um G no anticódon pode parear com um C ou U (mas não um A ou G) na terceira posição do códon, como mostrado abaixo4start superscript, 4, end superscript. Regras como esta asseguram que os códons sejam lidos corretamente, independentemente da oscilação.
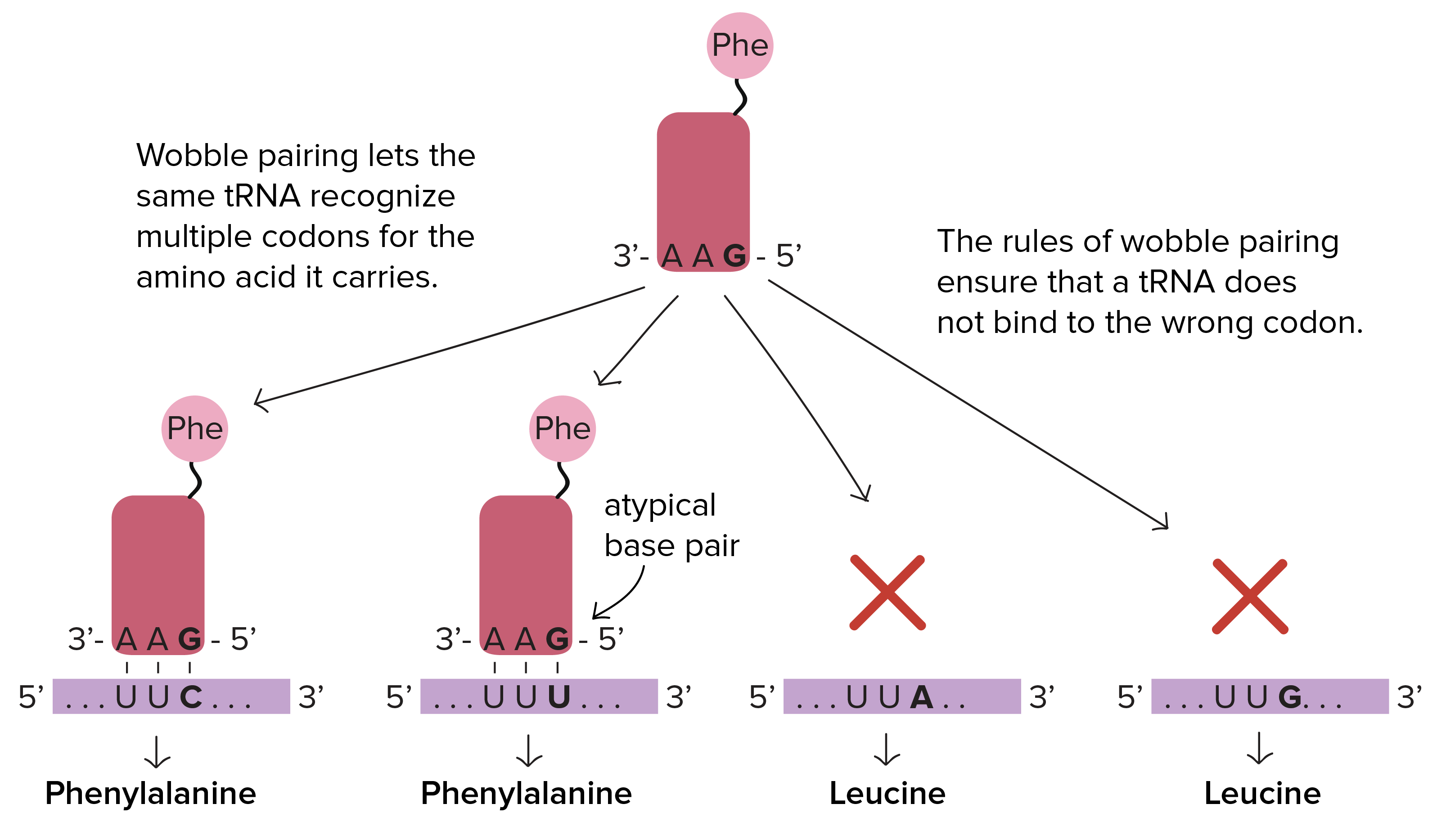 O pareamento oscilante permite que o mesmo RNAt reconheça múltiplos códons para o aminoácido que carrega. Por exemplo, o RNAt para a fenilalanina tem um anticódon de 3'-AAG-5'. Ele pode parear com os códons de RNAm 5'-UUC-3' ou 5'-UUU-3' (ambos os quais são códons que especificam a fenilalanina). O RNAt pode se ligar a ambos os códons porque consegue formar tanto um par de base normal com a terceira posição do códon (códon 5'-UUC-3' com anticódon 3'-AAG-5') quanto um par de base atípico com a terceira posição do códon (códon 5'-UUU-3' com anticódon 3'-AAG-5').As regras do pareamento oscilante asseguram que um RNAt não se ligue a um códon errado. O RNAt para a fenilalanina tem o anticódon 3'-AAG-5', o qual consegue parear com dois códons de fenilalanina (descritos acima), ma não com os códons 5'-UUA-3' ou 5'-UUG-3'. Esses códons especificam a leucina e não a fenilalanina, portanto esse é um exemplo de como as regras de pareamento oscilante permitem que um único RNAt cubra múltiplos códons para o mesmo aminoácido, mas não introduz nenhuma incerteza sobre qual aminoácido será entregue a um códon em particular.Imagem modificada de "Tradução: Figura 3," by OpenStax College, Biology (CC BY 4.0).Você deve estar se perguntando: por que diabos uma célula iria "querer" um fator complicante como a oscilação? A resposta pode ser que o pareamento oscilante permite que menos RNAts cubram todos os códons do código genético, enquanto ainda garantem que o código seja lido com acurácia.
O pareamento oscilante permite que o mesmo RNAt reconheça múltiplos códons para o aminoácido que carrega. Por exemplo, o RNAt para a fenilalanina tem um anticódon de 3'-AAG-5'. Ele pode parear com os códons de RNAm 5'-UUC-3' ou 5'-UUU-3' (ambos os quais são códons que especificam a fenilalanina). O RNAt pode se ligar a ambos os códons porque consegue formar tanto um par de base normal com a terceira posição do códon (códon 5'-UUC-3' com anticódon 3'-AAG-5') quanto um par de base atípico com a terceira posição do códon (códon 5'-UUU-3' com anticódon 3'-AAG-5').As regras do pareamento oscilante asseguram que um RNAt não se ligue a um códon errado. O RNAt para a fenilalanina tem o anticódon 3'-AAG-5', o qual consegue parear com dois códons de fenilalanina (descritos acima), ma não com os códons 5'-UUA-3' ou 5'-UUG-3'. Esses códons especificam a leucina e não a fenilalanina, portanto esse é um exemplo de como as regras de pareamento oscilante permitem que um único RNAt cubra múltiplos códons para o mesmo aminoácido, mas não introduz nenhuma incerteza sobre qual aminoácido será entregue a um códon em particular.Imagem modificada de "Tradução: Figura 3," by OpenStax College, Biology (CC BY 4.0).Você deve estar se perguntando: por que diabos uma célula iria "querer" um fator complicante como a oscilação? A resposta pode ser que o pareamento oscilante permite que menos RNAts cubram todos os códons do código genético, enquanto ainda garantem que o código seja lido com acurácia.
A estrutura 3D de um RNAt
- Eu gosto de desenhar RNAts como pequenos retângulos, para mostrar claramente o que está acontecendo (e ter espaço de sobra para colocar as letras do anticódon ali). Mas um RNAt de verdade possui um formato muito mais interessante; um que facilita o seu papel.Um RNAt, como o modelado abaixo, é composto por uma cadeia única de RNA (assim como o RNAm é). Entretanto, a cadeia toma uma estrutura tridimensional complexa pois pares de bases se formam entre nucleotídeos em diferentes partes da molécula. Isto gera regiões de fita dupla e alças, dobrando o RNAt num formato de "L".
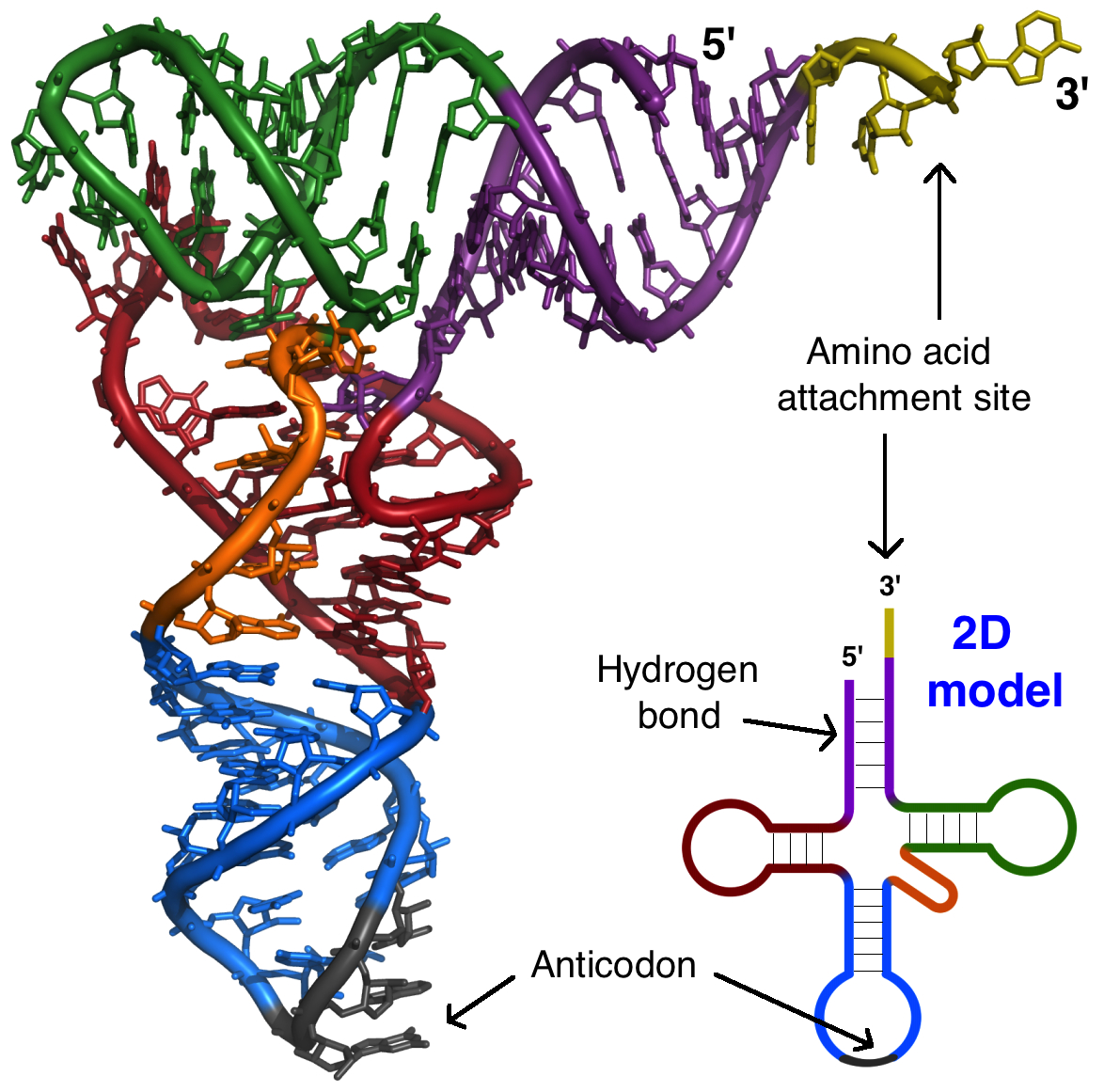 Uma molécula de RNAt tem uma estrutura em "L" mantida unida por pontes de hidrogênio entre bases de diferentes partes da sequência do RNAt. Uma extremidade do RNAt se liga um aminoácido específico (sítio de fixação do aminoácido) e a outra extremidade possui um anticódon que se ligará a um códon do RNAm._imagem modificada de "TRNA-Phe yeast," by Yikrazuul (CC BY-SA 3.0). A imagem modificada está autorizada sob um licença CC BY-SA 3.0._Uma das pontas do formato de L abriga o anticódon, enquanto a outra possui o sítio de ligação para o aminoácido. Diferentes RNAts possuem estruturas ligeiramente diferentes, e isto é importante para assegurar que eles serão acoplados ao aminoácido correto.
Uma molécula de RNAt tem uma estrutura em "L" mantida unida por pontes de hidrogênio entre bases de diferentes partes da sequência do RNAt. Uma extremidade do RNAt se liga um aminoácido específico (sítio de fixação do aminoácido) e a outra extremidade possui um anticódon que se ligará a um códon do RNAm._imagem modificada de "TRNA-Phe yeast," by Yikrazuul (CC BY-SA 3.0). A imagem modificada está autorizada sob um licença CC BY-SA 3.0._Uma das pontas do formato de L abriga o anticódon, enquanto a outra possui o sítio de ligação para o aminoácido. Diferentes RNAts possuem estruturas ligeiramente diferentes, e isto é importante para assegurar que eles serão acoplados ao aminoácido correto.
Carregando um RNAt com um aminoácido
- Como o aminoácido correto é acoplado ao RNAt certo (assegurando que os códons sejam lidos corretamente)? Enzimas chamadas aminoacil-RNAt-sintetases têm este papel importante.Há uma diferente enzima sintetase para cada aminoácido, uma que reconhece apenas aquele aminoácido e seu RNAt (e nenhum outro). Assim que ambos o aminoácido e seu RNAt se anexam à enzima, a enzima liga um ao outro, numa reação alimentada pela "moeda energética": uma molécula de adenosina trifosfato (ATP).
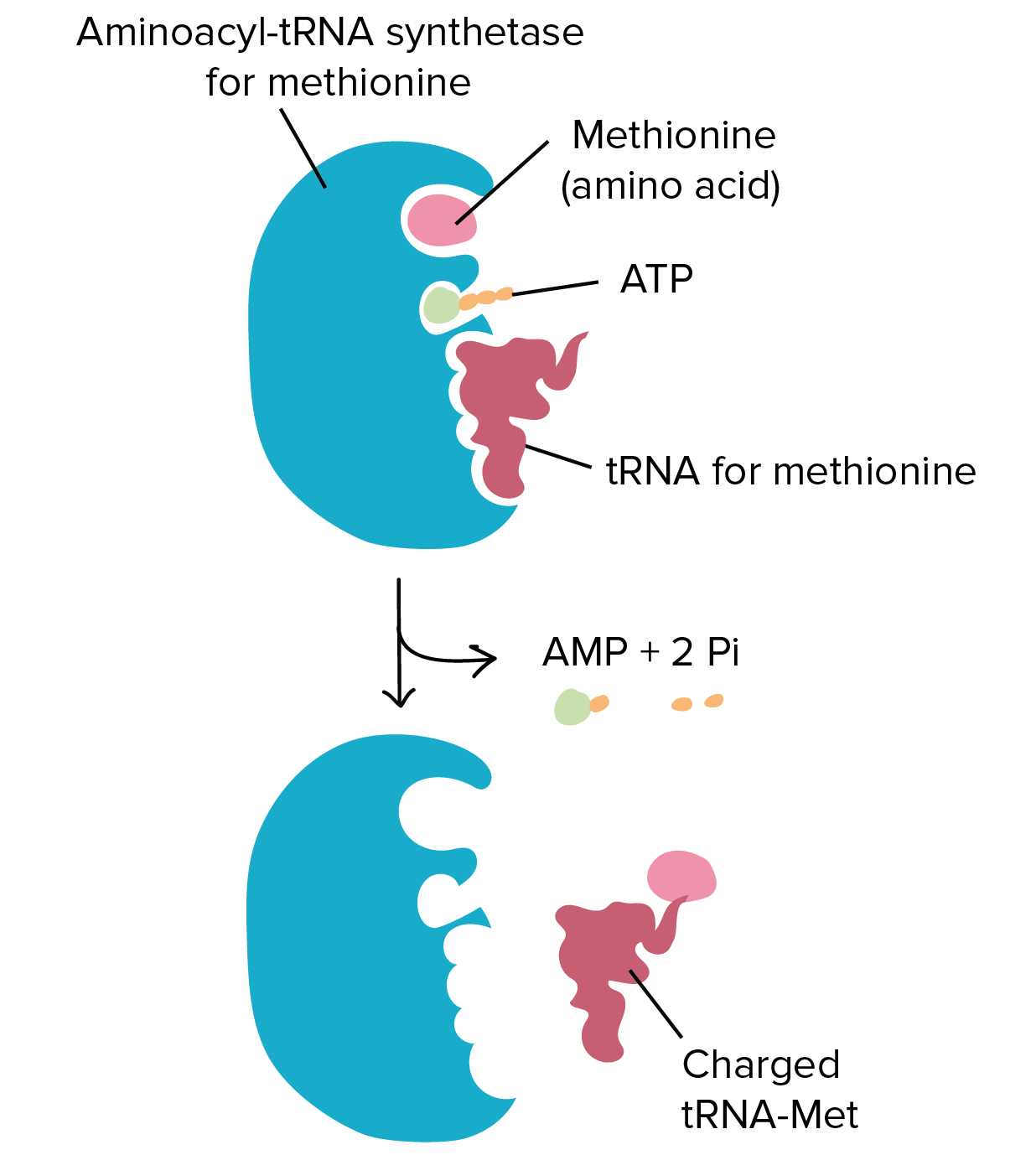 O sítio ativo de cada aminoacil-RNAt sintetase se combina a um RNAt associado e um aminoácido em particular como uma "chave e fechadura". Em seguida, o ATP é utilizado para ligar o aminoácido ao RNAt._Imagem modificada de "Charge tRNA," by Boumphreyfr (CC BY-SA 3.0). A imagem modificada está autorizada sob licença CC BY-SA 3.0 license._Ocasionalmente, uma aminoacil-RNAt-sintetase comete um erro: ela se liga ao aminoácido incorreto (um que "pareça semelhante" ao seu alvo). Por exemplo, a treonina sintetase às vezes captura uma serina acidentalmente e a anexa ao RNAt de treonina. Felizmente, a treonina sintetase possui um sítio de verificação, que remove o aminoácido do tNAt se ele for incorreto5start superscript, 5, end superscript.
O sítio ativo de cada aminoacil-RNAt sintetase se combina a um RNAt associado e um aminoácido em particular como uma "chave e fechadura". Em seguida, o ATP é utilizado para ligar o aminoácido ao RNAt._Imagem modificada de "Charge tRNA," by Boumphreyfr (CC BY-SA 3.0). A imagem modificada está autorizada sob licença CC BY-SA 3.0 license._Ocasionalmente, uma aminoacil-RNAt-sintetase comete um erro: ela se liga ao aminoácido incorreto (um que "pareça semelhante" ao seu alvo). Por exemplo, a treonina sintetase às vezes captura uma serina acidentalmente e a anexa ao RNAt de treonina. Felizmente, a treonina sintetase possui um sítio de verificação, que remove o aminoácido do tNAt se ele for incorreto5start superscript, 5, end superscript.
Resumindo
Assim que carregados com o aminoácido correto, como os RNAts interagem com RNAms e o ribossomo para construir uma proteína nova em folha? Aprenda mais sobre como este processo funciona no próximo artigo: estágios da tradução. Créditos:
Este artigo é uma derivação modificada de "Ribosomes and protein synthesis," by OpenStax College, Biology (CC BY 4.0). Baixe o artigo original gratuitamente em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@10.61.O artigo adaptado está sob a licença CC BY-NC-SA 4.0Referências:
- Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Ribosomes. Em Campbell biology (10th ed., pp. 347-48). San Francisco, CA: Pearson.
- Goodsell, D. S. (2000). Ribosomal subunits. Em RCSB molecule of the month. Disponível em http://pdb101.rcsb.org/motm/10.
- OpenStax College, Biology. (2015, September 30). Ribosomes and protein synthesis. Em OpenStax CNX. Disponível em https://cnx.org/contents/s8Hh0oOc@8.57:FUH9XUkW@6/Translation.
- Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). Some transfer RNA molecules recognize more than one codon because of wobble in base-pairing. Em Biochemistry. (5th ed., section 29.3.9). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK22335/#_A4185_.
- Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). Proofreading by aminoacyl-tRNA synthetases increases the fidelity of protein synthesis. Em Biochemistry. (5th ed., section 29.2.3). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK22356/#_A4151_.
Referências
Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). Aminoacyl-transfer RNA synthetases read the genetic code. In Biochemistry. (5th ed., section 29.2). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK22356/.Goodenbour, J. M. and Pan, T. (2006). Diversity of tRNA genes in eukaryotes. Nucl. Acids Res., 34(21), 6137-6146. http://dx.doi.org/10.1093/nar/gkl725.Goodsell, D. (April 2001). Aminoacyl-tRNA synthetases. In RCSB molecule of the month. Disponível em http://pdb101.rcsb.org/motm/16.Nelson, D. L., Lehninger, A. L., and Cox, M. M. (2008). Protein synthesis. InPrinciples of biochemistry(5th ed., pp. 1075 -1099). New York, NY: W.H. Freeman & Company.OpenStax College, Biology. (30 de setembro de 2015). Ribosomes and protein synthesis. In OpenStax CNX. Disponível em: https://cnx.org/contents/s8Hh0oOc@8.57:FUH9XUkW@6/Translation.OpenStax College, Biology. (n.d). Translation. In OpenStax CNX. Disponível em http://philschatz.com/biology-concepts-book/contents/m45479.html.Purves, W. K., Sadava, D., Orians, G. H., and Heller, H. C. (2004). From DNA to protein: Genotype to phenotype. In Life: the science of biology (7th ed., pp. 233-256). Sunderland, MA: Sinauer Associates, Inc.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Gene expression: from gene to protein. In Campbell biology.10 ed. p. 333-359. San Francisco: Pearson, 2011.Transfer RNA / tRNA. (2014). In Scitable. Disponível em http://www.nature.com/scitable/definition/trna-transfer-rna-256.Wobble base pair. (2 de dezembro de 2015). Acesso em 21 de dezembro de 2015 em Wikipedia: https://en.wikipedia.org/wiki/Wobble_base_pair. Etapas da traduçãoIntrodução
Já se perguntou de que maneira os antibióticos matam as bactérias—por exemplo, quando você tem uma sinusite? Diferentes antibióticos atuam de maneiras diferentes, mas alguns atacam um processo bem básico nas células bacterianas: eles eliminam a capacidade de produzir novas proteínas.Para usarmos um pouco do vocabulário da biologia molecular, esses antibióticos bloqueiam a tradução. No processo de tradução, a célula lê a informação de uma molécula chamada RNA mensageiro (RNAm) e usa essa informação para construir uma proteína. A tradução acontece constantemente em uma célula bacteriana normal, assim como na maioria das células de seu corpo e é fundamental para mantê-lo vivo (e suas bactérias "visitantes" também).Quando você toma certos antibióticos (por exemplo, eritromicina), a molécula de antibiótico se associa a moléculas fundamentais para a tradução de dentro da célula bacteriana e basicamente as paralisa. Sem uma maneira de produzir proteína, a bactéria para de funcionar e vai morrer eventualmente. Por esse motivo que infecções são curadas quando tratadas com antibióticos.1,2start superscript, 1, comma, 2, end superscriptAs células precisam da tradução para se manterem vivas , e entender o funcionamento (para que possamos impedi-la com antibióticos) pode nos salvar das infecções bacterianas. Vamos analisar como acontece a tradução, da primeira etapa até o produto final.Tradução: visão geral
A tradução envolve "decodificar" um RNA mensageiro (RNAm) e usar sua informação para produzir um polipeptídeo ou cadeia de aminoácidos. No geral, polipeptídeo é basicamente uma proteína (com a diferença técnica que algumas proteínas grandes são formadas por muitas cadeias de polipeptídeos.O código genético
Em um RNAm, as instruções para a produção de um polipeptídeo vêm em grupos de três nucleotídeos chamados códons. Aqui estão algumas características principais dos códons para termos em mente ao seguirmos a diante:- Existem 6161 códons diferentes para os aminoácidos
- Três códons de "parada" marcam o polipeptídeo como terminado
- Um códon AUG é um sinal de "início" para começar a tradução (também especifica o aminoácido metionina)
Essas relações entre os códons do RNAm e os aminoácidos são conhecidas como código genético (sobre o qual você pode explorar mais no artigo código genético.De códons a aminoácidos
Na tradução, os códons de um RNAm são lidos por ordem (da extremidade 5' para a extremidade 3') por moléculas chamadas de RNAs de transferência ou RNAt.Cada RNAt possui um anticódon, um conjunto de três nucleotídeos que se liga ao códon correspondente no RNAm através do pareamento de bases. A outra extremidade do RNAt traz o aminoácido especificado pelo códon.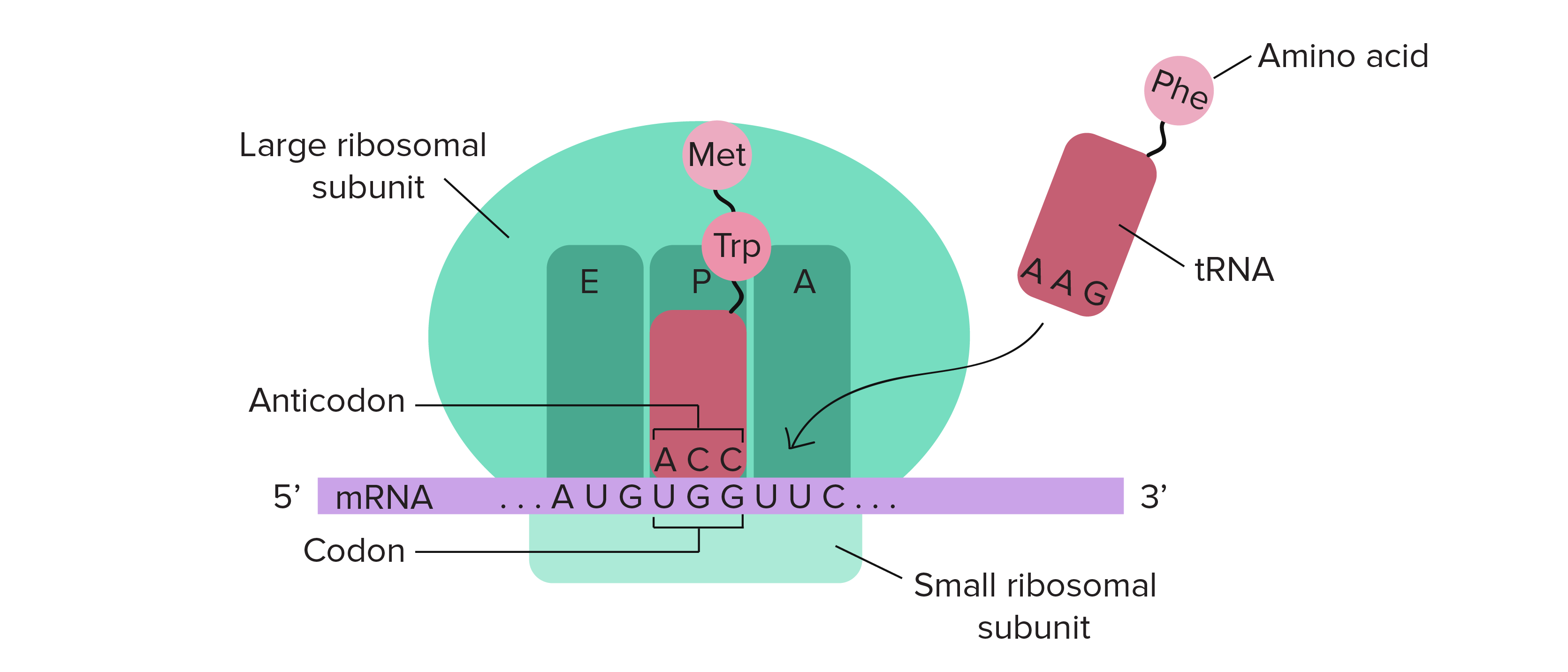 O ribossomo provê um lugar no qual um RNAm pode interagir com RNAt que transportam aminoácidos. Há três compartimentos no ribossomo onde os RNAt se ligam: os sítios A, P e E. O sítio A recebe um RNAt de entrada ligado a um aminoácido. O sítio P detém um RNAt que carrega um polipeptídeo crescente (o primeiro aminoácido adicionado é a metionina (Met)). O sítio E é o local para onde um RNAt vai após estar vazio, o que significa que transferiu seu polipeptídeo para outro RNAt (que agora ocupa o sítio P). No diagrama, o RNAt vazio já deixou o sítio E, portanto não é representado.Imagem modificada de "Tradução: Figura 3," by OpenStax College, Biology (CC BY 4.0).Os RNAt se ligam aos RNAm dentro de uma estrutura de RNA e proteína chamada ribossomo. À medida que os RNAt preenchem os compartimentos do ribossomo e se ligam aos códons, seus aminoácidos são adicionados à cadeia crescente de polipeptídeos em uma reação química. O produto final é um polipeptídeo cuja sequência de aminoácidos reflete a sequência de códons no RNAm.
O ribossomo provê um lugar no qual um RNAm pode interagir com RNAt que transportam aminoácidos. Há três compartimentos no ribossomo onde os RNAt se ligam: os sítios A, P e E. O sítio A recebe um RNAt de entrada ligado a um aminoácido. O sítio P detém um RNAt que carrega um polipeptídeo crescente (o primeiro aminoácido adicionado é a metionina (Met)). O sítio E é o local para onde um RNAt vai após estar vazio, o que significa que transferiu seu polipeptídeo para outro RNAt (que agora ocupa o sítio P). No diagrama, o RNAt vazio já deixou o sítio E, portanto não é representado.Imagem modificada de "Tradução: Figura 3," by OpenStax College, Biology (CC BY 4.0).Os RNAt se ligam aos RNAm dentro de uma estrutura de RNA e proteína chamada ribossomo. À medida que os RNAt preenchem os compartimentos do ribossomo e se ligam aos códons, seus aminoácidos são adicionados à cadeia crescente de polipeptídeos em uma reação química. O produto final é um polipeptídeo cuja sequência de aminoácidos reflete a sequência de códons no RNAm.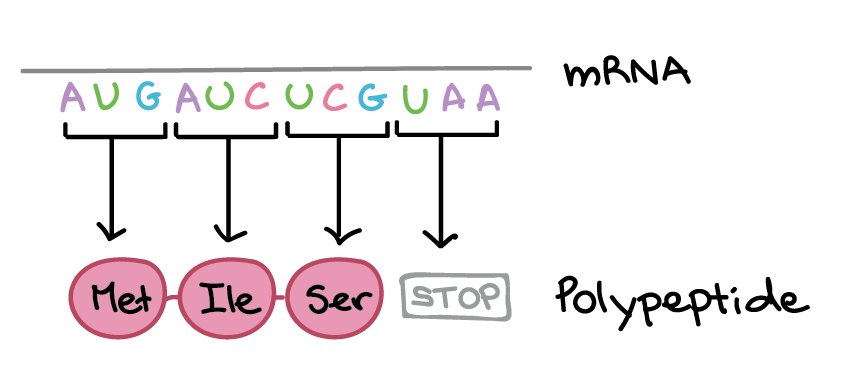 A sequência de RNAm é:5'-AUGAUCUCGUAA-5'A tradução envolve a leitura dos nucleotídeos do RNAm em grupos de três, cada qual especifica um aminoácido (ou fornece um sinal de parada indicando que a tradução terminou).3'-AUG AUC UCG UAA-5'AUG →right arrow Metionina AUC →right arrow Isoleucina UCG →right arrow Serina UAA →right arrow "Pare"Sequência de polipeptídeo: (N-terminal) Metionina-Isoleucina-Serina (C-terminal)Essa é a visão geral da tradução. E quanto aos pormenores de como a tradução se inicia, procede e termina? Vamos examinar.
A sequência de RNAm é:5'-AUGAUCUCGUAA-5'A tradução envolve a leitura dos nucleotídeos do RNAm em grupos de três, cada qual especifica um aminoácido (ou fornece um sinal de parada indicando que a tradução terminou).3'-AUG AUC UCG UAA-5'AUG →right arrow Metionina AUC →right arrow Isoleucina UCG →right arrow Serina UAA →right arrow "Pare"Sequência de polipeptídeo: (N-terminal) Metionina-Isoleucina-Serina (C-terminal)Essa é a visão geral da tradução. E quanto aos pormenores de como a tradução se inicia, procede e termina? Vamos examinar.Tradução: começo, meio e fim
Um livro ou um filme possuem três partes básicas: um começo, um meio e um fim. A tradução tem praticamente estas mesmas três partes, mas com nomes mais elegantes: iniciação, alongamento e terminação.- Iniciação ("começo"): nesta etapa, o ribossomo se junta ao RNAm e ao primeiro RNAt para que a tradução possa ter início.
- Alongamento ("meio"): nesta etapa, os aminoácidos são trazidos ao ribossomo pelos RNAt e são ligados entre si para formar uma cadeia.
- Terminação ("fim"): na última etapa, o polipeptídeo final é liberado para que possa cumprir sua função na célula.
Vamos analisar como funciona cada etapa.Iniciação
Para que se possa dar início à tradução, alguns ingredientes são necessários. Esses incluem:- Um ribossomo (o qual tem duas parte, uma pequena e uma grande)
- Um RNAm com as instruções para a proteína que será construída
- Um RNAt "iniciador" transportando o primeiro aminoácido da proteína, que quase sempre é a metionina (Met)
Durante a iniciação, essas peças devem se unir de maneira correta. Juntas, elas formam o complexo de iniciação, a configuração molecular necessária para começar a fazer uma nova proteína.Dentro de suas células (e das células de outros eucariontes), a iniciação da tradução acontece assim: primeiro o RNAt transportando a metionina se liga a subunidade ribossômica pequena. Juntos, se ligam à extremidade 5' do RNAm através do reconhecimento do cap 5' GTP (adicionado durante o processamento no núcleo). Em seguida, eles "caminham" ao longo do RNAm na direção 3' e param quando alcançam o códon de iniciação (com frequência, mas nem sempre, o primeiro AUG).6start superscript, 6, end superscript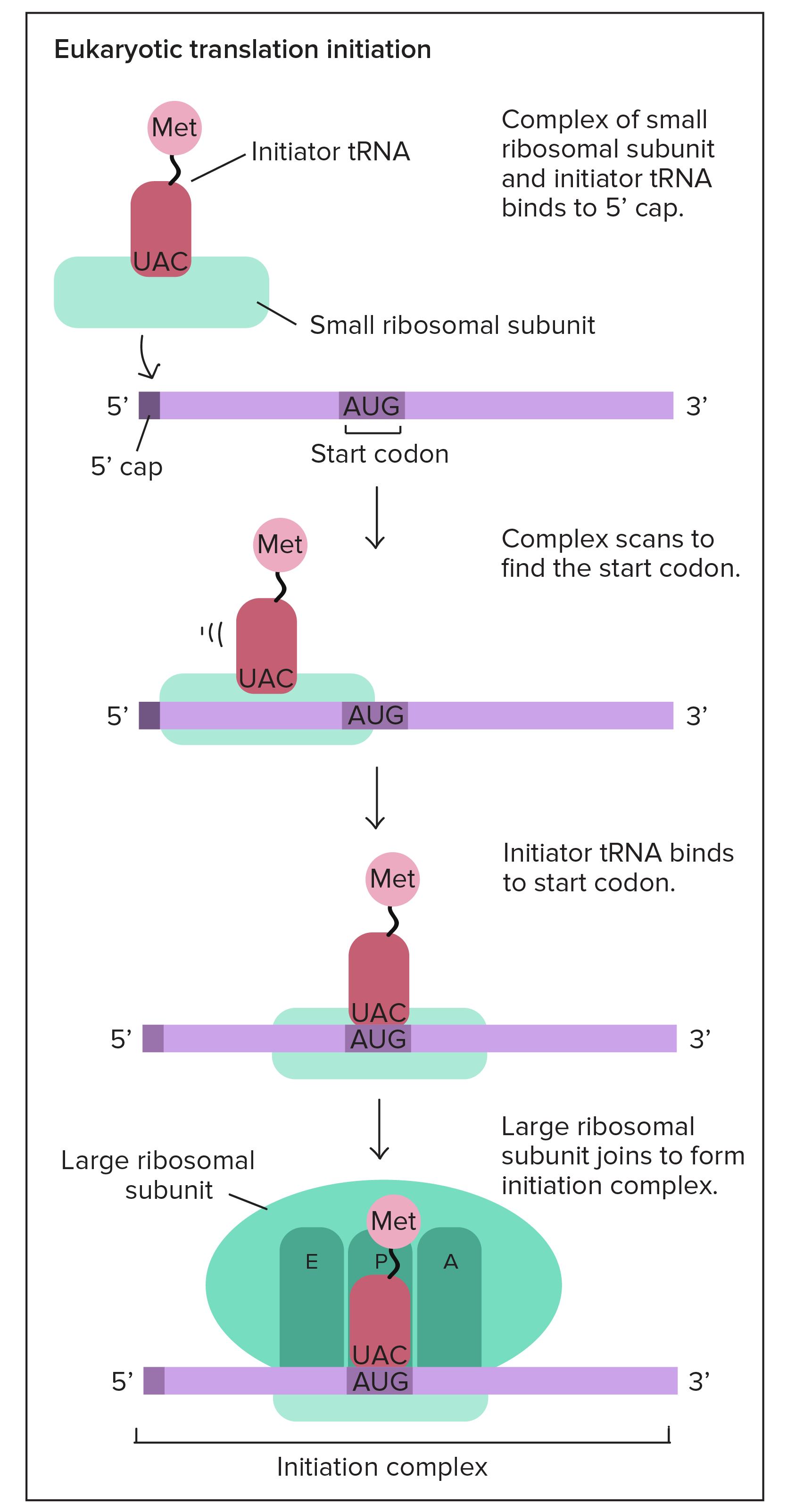 Iniciação da tradução eucariótica:
Iniciação da tradução eucariótica:- Complexo de subunidade ribossômica pequena e RNAt iniciador (transportando metionina) se liga ao 5' do RNAm.
- O complexo escaneia do 5' para o 3' para encontrar o códon de iniciação (AUG).
- O RNAt iniciador se liga ao códon de iniciação.
- A subunidade ribossômica grande se reúne a RNAm, RNAt iniciador e subunidade ribossômica pequena para formar o complexo de iniciação. O RNAt iniciador é posicionado no sítio P do ribossomo montado.
Essas etapas são auxiliadas por fatores de iniciação (não representados no diagrama).Baseado em diagrama similar de Berg et al.1start superscript, 1, end superscriptNas bactérias, a situação é um pouco diferente. Aqui, a subunidade ribossômica pequena não começa pela extremidade 5' e caminha em direção a 3'. Em vez disso, ela se associa diretamente a determinadas sequências no RNAm. Essas sequências Shine-Dalgarno antecedem os códons e "os apontam" para o ribossomo.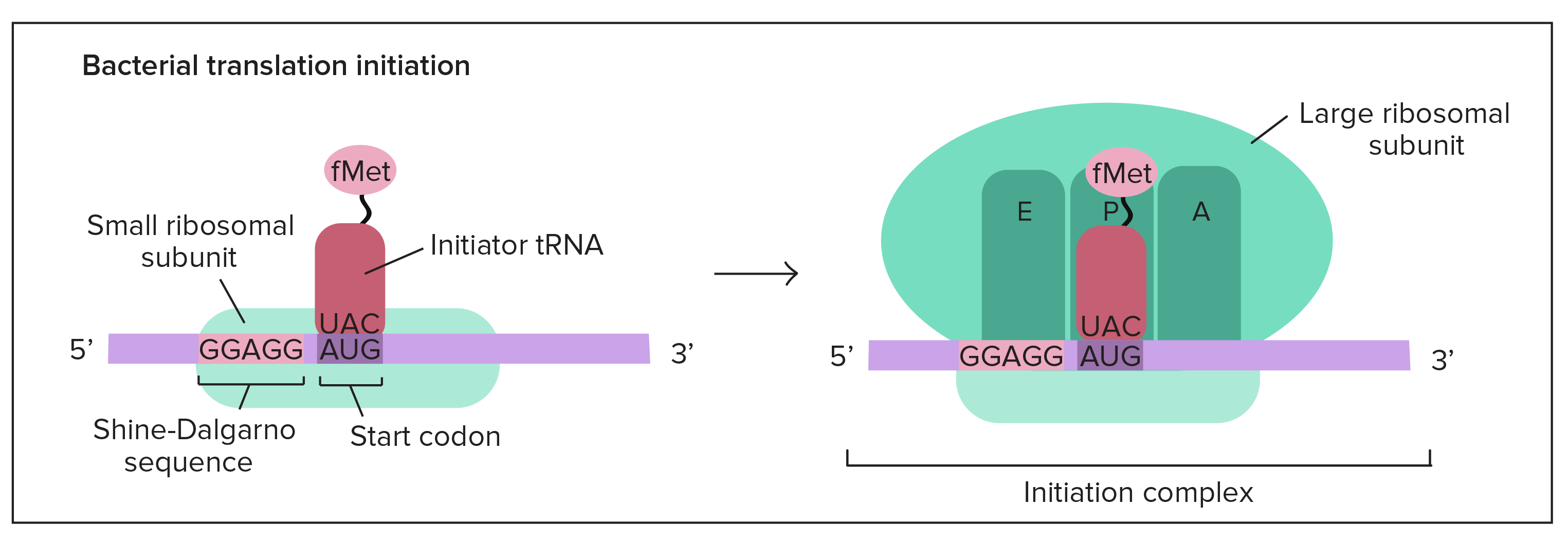 Iniciação da tradução bacteriana:Em um RNAm bacteriano, uma sequência rica em G/A chamada de sequência Shine-Dalgarno é encontrada um pouco acima (5') do códon de início (AUG). A subunidade ribossômica pequena também se liga ao iniciador RNAt (transportando fMET), que forma pares de bases complementares com o códon de início. Como percebido no texto, a subunidade ribossômica pequena pode, às vezes, se ligar primeiro ao RNAm (e então ao RNAt iniciador), e, às vezes, ao inverso (primeiro o iniciador RNAt e depois o RNAm). Acredita-se que a ordem desses eventos seja aleatória.Uma vez que esses componentes se juntaram, a subunidade ribossômica grande se junta a eles. A reunião do ribossomo com o RNAm ligada ao RNAt iniciador formam o complexo de iniciação. O RNAt iniciador está no sítio P do ribossomo montado.Bactérias usam a fMet (uma metionina quimicamente modificada) como primeiro aminoácido.Por que usar sequências Shine-Dalgarno? Os genes bacterianos são frequentemente traduzidos em grupos chamados operons), portanto um RNAm bacteriano pode conter as sequências codificadoras de muitos genes. Uma sequência de Shine-Dalgarno marca o início de cada sequência codificadora, o que permite que o ribossomo encontre o códon de iniciação certo para cada gene.
Iniciação da tradução bacteriana:Em um RNAm bacteriano, uma sequência rica em G/A chamada de sequência Shine-Dalgarno é encontrada um pouco acima (5') do códon de início (AUG). A subunidade ribossômica pequena também se liga ao iniciador RNAt (transportando fMET), que forma pares de bases complementares com o códon de início. Como percebido no texto, a subunidade ribossômica pequena pode, às vezes, se ligar primeiro ao RNAm (e então ao RNAt iniciador), e, às vezes, ao inverso (primeiro o iniciador RNAt e depois o RNAm). Acredita-se que a ordem desses eventos seja aleatória.Uma vez que esses componentes se juntaram, a subunidade ribossômica grande se junta a eles. A reunião do ribossomo com o RNAm ligada ao RNAt iniciador formam o complexo de iniciação. O RNAt iniciador está no sítio P do ribossomo montado.Bactérias usam a fMet (uma metionina quimicamente modificada) como primeiro aminoácido.Por que usar sequências Shine-Dalgarno? Os genes bacterianos são frequentemente traduzidos em grupos chamados operons), portanto um RNAm bacteriano pode conter as sequências codificadoras de muitos genes. Uma sequência de Shine-Dalgarno marca o início de cada sequência codificadora, o que permite que o ribossomo encontre o códon de iniciação certo para cada gene.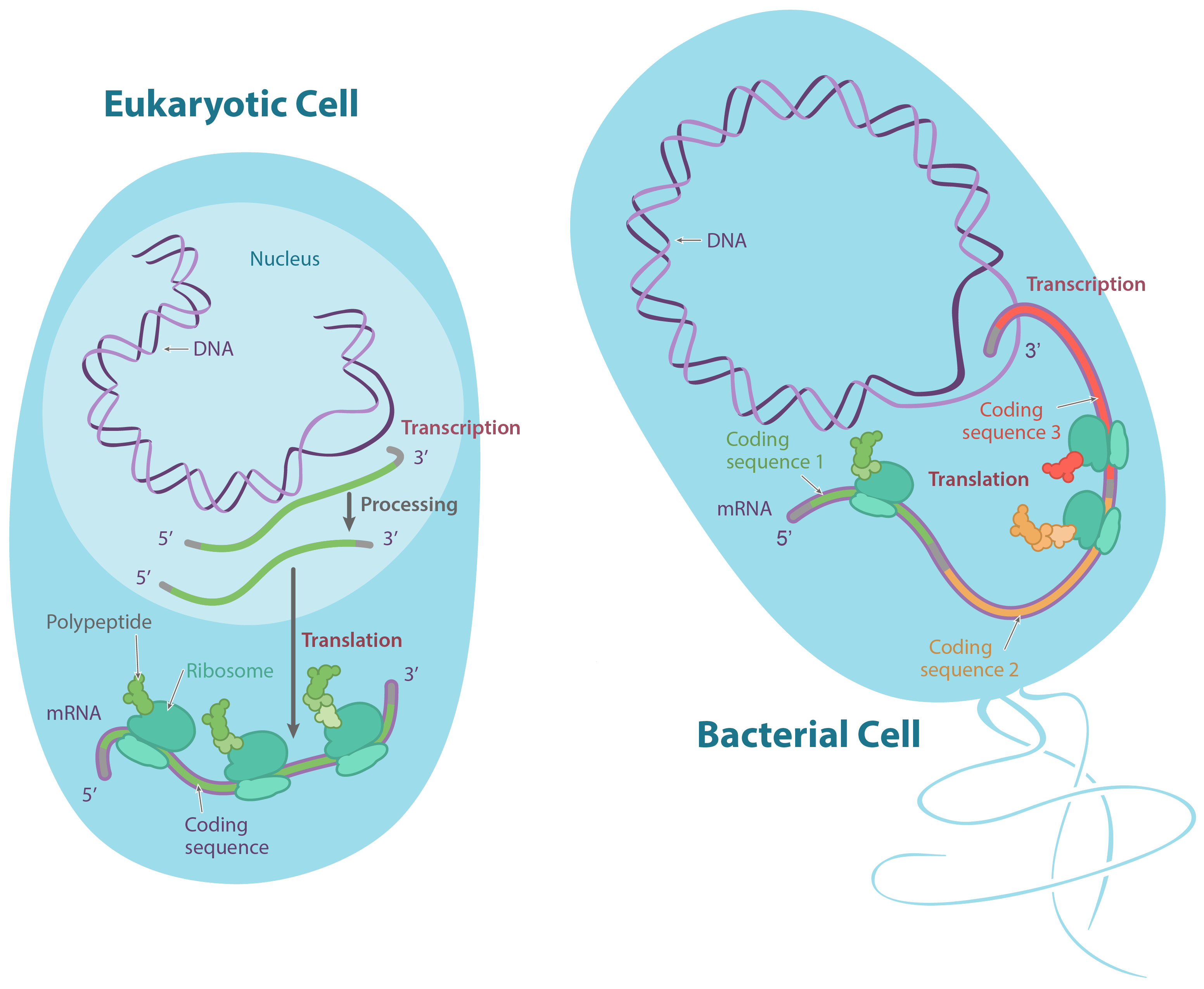 Célula eucarionte:
Célula eucarionte:- O DNA é transcrito para fazer um RNA dentro do núcleo. O RNA inicial transcrito é processado em um RNAm maduro antes da exportação para o hialoplasma.
- O RNAm contém apenas uma sequência codificadora (que especifica um polipeptídeo).
Célula bacteriana:- O DNA é transcrito para fazer um RNAm no hialoplasma. Os ribossomos podem começar a tradução no RNAm antes que ele esteja totalmente transcrito. Nenhum processo pós transcrição é necessário.
- O RNAm contém três sequências codificadoras de três genes diferentes, cada um especificando seu próprio polipeptídeo.
Alongamento
Eu gosto de lembrar o que acontece nessa etapa do "meio" da tradução por seu nome prático: alongamento é quando o polipeptídeo se torna mais longo.Mas como exatamente a cadeia aumenta? Para descobrirmos, vamos examinar a primeira rodada do alongamento—depois de formado o complexo de iniciação, porém antes que qualquer aminoácido tenha sido adicionado para formar a cadeia.Nosso primeiro RNAt transportador de metionina começa no compartimento do meio no ribossomo, chamado de sítio P. Ao lado, um novo códon é exposto em outro compartimento, chamado de sítio A. O sítio A será o "desembarque" para o próximo RNAt, aquele cujo anticódon é o correspondente perfeito (complementar) do códon em exposição.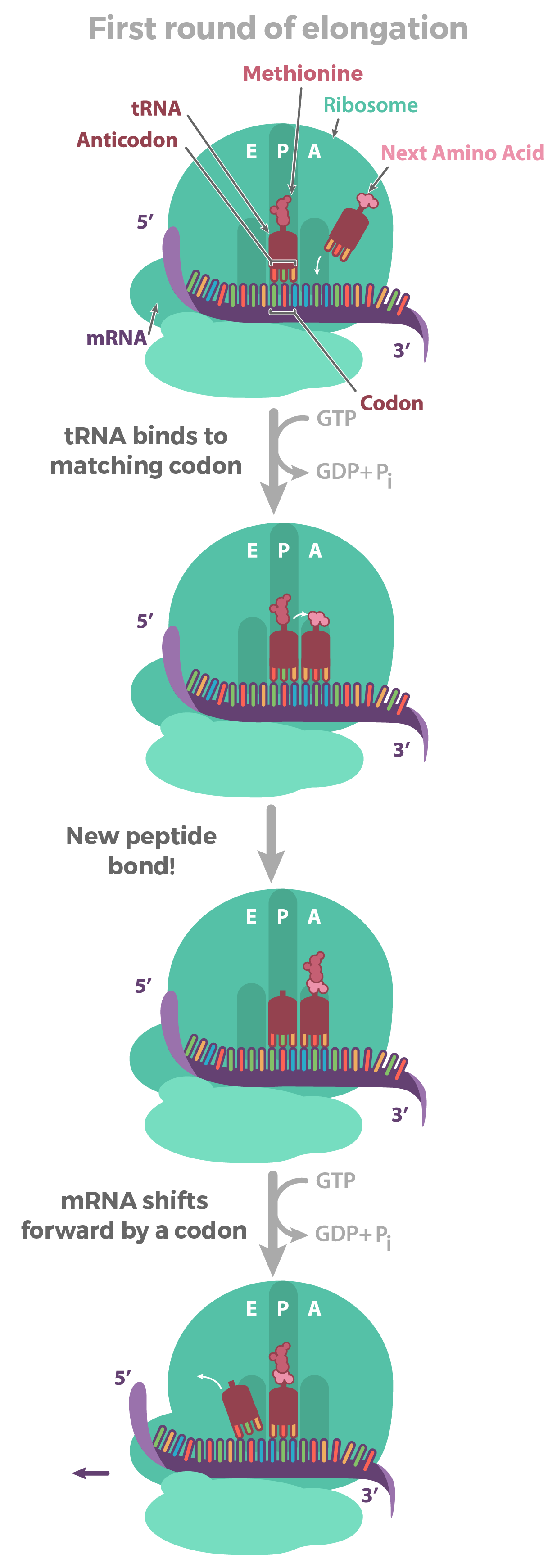 Na primeira rodada de alongamento, um aminoácido de entrada se anexa à metionina já presente no sítio P do ribossomo. Essa ação inicia o crescimento do polipeptídeo. As três etapas dessa primeira rodada de alongamento são descritas abaixo.1) Reconhecimento de códon: um RNAt de entrada com um anticódon que é complementar ao códon exposto no sítio A se liga ao RNAm. A energia do GTP é gasta para aumentar a acurácia do reconhecimento de códons.2) Formação da ligação peptídica: uma ligação peptídica é formada entre o aminoácido de entrada (transportado por um RNAt no sítio A) e uma metionina (um RNAt carregado com metionina anexado ao sítio P durante a iniciação). Essa ação passa o polipeptídeo (os dois aminoácidos ligados) do RNAt no sítio P ao RNAt no sítio A. O RNAt no sítio P agora está "vazio" porque não detém o polipeptídeo.3) Translocação: o ribossomo move um códon sobre o RNAm na direção da extremidade 3'. Isso desloca o RNAt no sítio A para o sítio P, e o RNAt no sítio P para o sítio E. O RNAt vazio no sítio E então sai do ribossomo.Uma vez que o RNAt correspondente desembarca no sítio A é hora da ação: isto é, a formação de uma ligação peptídica que conecta um aminoácido a outro. Esta etapa transfere a metionina do primeiro RNAt para o aminoácido do segundo RNAt no sítio A.Nada mal—agora temos dois aminoácidos, um (minúsculo) polipeptídeo! A metionina forma o N-terminal do polipeptídeo, e o outro amioácido é o C-terminal.Contudo... as maiores chances são que queiramos um polipeptídeo mais longo do que dois aminoácidos. Como a cadeia continua a aumentar? Depois que a ligação peptídica é formada, o RNAm é puxado para frente no ribossomo por exatamente um códon. Esse deslocamento permite que o primeiro RNAt, agora vazio, saia através do sítio E (do inglês "exit"). Isso também faz com que um novo códon fique exposto no sítio A, para que todo ciclo se repita.E se repete... de alguma vezes até espantosas 33,33, comma000000 vezes! A proteína titina, que é encontrada em nossos músculos e é o mais longo polipeptídeo conhecido, pode ter mais de 33,33, comma000000 aminoácidos8,9start superscript, 8, comma, 9, end superscript.
Na primeira rodada de alongamento, um aminoácido de entrada se anexa à metionina já presente no sítio P do ribossomo. Essa ação inicia o crescimento do polipeptídeo. As três etapas dessa primeira rodada de alongamento são descritas abaixo.1) Reconhecimento de códon: um RNAt de entrada com um anticódon que é complementar ao códon exposto no sítio A se liga ao RNAm. A energia do GTP é gasta para aumentar a acurácia do reconhecimento de códons.2) Formação da ligação peptídica: uma ligação peptídica é formada entre o aminoácido de entrada (transportado por um RNAt no sítio A) e uma metionina (um RNAt carregado com metionina anexado ao sítio P durante a iniciação). Essa ação passa o polipeptídeo (os dois aminoácidos ligados) do RNAt no sítio P ao RNAt no sítio A. O RNAt no sítio P agora está "vazio" porque não detém o polipeptídeo.3) Translocação: o ribossomo move um códon sobre o RNAm na direção da extremidade 3'. Isso desloca o RNAt no sítio A para o sítio P, e o RNAt no sítio P para o sítio E. O RNAt vazio no sítio E então sai do ribossomo.Uma vez que o RNAt correspondente desembarca no sítio A é hora da ação: isto é, a formação de uma ligação peptídica que conecta um aminoácido a outro. Esta etapa transfere a metionina do primeiro RNAt para o aminoácido do segundo RNAt no sítio A.Nada mal—agora temos dois aminoácidos, um (minúsculo) polipeptídeo! A metionina forma o N-terminal do polipeptídeo, e o outro amioácido é o C-terminal.Contudo... as maiores chances são que queiramos um polipeptídeo mais longo do que dois aminoácidos. Como a cadeia continua a aumentar? Depois que a ligação peptídica é formada, o RNAm é puxado para frente no ribossomo por exatamente um códon. Esse deslocamento permite que o primeiro RNAt, agora vazio, saia através do sítio E (do inglês "exit"). Isso também faz com que um novo códon fique exposto no sítio A, para que todo ciclo se repita.E se repete... de alguma vezes até espantosas 33,33, comma000000 vezes! A proteína titina, que é encontrada em nossos músculos e é o mais longo polipeptídeo conhecido, pode ter mais de 33,33, comma000000 aminoácidos8,9start superscript, 8, comma, 9, end superscript.Terminação
Polipeptídeos, assim como todas as boas coisas, devem eventualmente chegar a um fim. A tradução acaba em um processo chamado terminação. A terminação acontece quando um códon de parada no RNAm (UAA, UAG ou UGA) entra no sítio A.Códons de parada são reconhecidos por proteínas chamadas de fatores de liberação, os quais se adaptam perfeitamente no sítio P (embora não sejam RNAt). Fatores de liberação confundem a enzima que normalmente forma as ligações peptídicas: fazem-na adicionar uma molécula de água ao último aminoácido da cadeia. Essa reação separa a cadeia do RNAt, assim a proteína recém-produzida é liberada.E depois? Por sorte, o "equipamento" de tradução é bastante reutilizável. Depois que as unidades ribossômicas se separam do RNAm e entre si, cada elemento pode (e normalmente fazem isso rapidamente) tomar parte em um novo ciclo de tradução.Epílogo: Processamento
Agora nosso polipeptídeo tem todos os seus aminoácidos—isso significa que esteja pronto para atuar na célula?Não necessariamente. Polipeptídeos muitas vezes precisam de algumas "edições". Durante e após a tradução, aminoácidos podem ser quimicamente alterados ou removidos. O novo polipeptídeo também vai se dobrar em uma estrutura em 3D distinta e podem se associar a outros polipeptídeos para formar uma proteína com múltiplas partes.Muitas proteínas são boas em se dobrarem sozinhas, mas algumas precisam de auxiliares ("chaperonas") para evitar que se unam incorretamente durante o processo complexo que é o dobramento.Algumas proteínas também contêm sequências especiais de aminoácidos que as direcionam para determinadas partes das células. Estas sequências, muitas vezes encontradas próximas aos terminais N e C, podem ser entendidas como as "passagens de trem" das proteínas para suas destinações finais. Para entender melhor como isso funciona, veja o artigo sobre direcionamento de proteínas.Créditos
Esse artigo é uma adaptação de "Ribosomes and protein synthesis," por OpenStax College, Biology, CC BY 4.0. Baixe o artigo original sem custos em http://cnx.org/contents/185cbf87-c72e-48f5-b51e-f14f21b5eabd@10.59.O artigo adaptado está sob a licença CC BY-NC-SA 4.0Trabalhos citados
- Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). Eukaryotic protein synthesis differs from prokaryotic protein synthesis primarily in translation initiation. In Biochemistry. (5th ed., section 29.5.1). New York, NY: W. H. Freeman. Disponível em https://www.ncbi.nlm.nih.gov/books/NBK22531/#_A4206_.
- Purves, W.K., Sadava, D., Orians, G.H., and Heller, H.C. (2004). From DNA to protein: Genotype to phenotype. In Life: The science of biology (7th ed., pp. 246-247). Sunderland, MA: Sinauer Associates, Inc.
- Ophardt, Charles E. (2003). Other antibiotics. In Virtual Chembook. Acesso em 22 de outubro de 2016 em http://chemistry.elmhurst.edu/vchembook/654antibiotic.html.
- Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). Protein factors play key roles in protein synthesis. In Biochemistry. (5th ed., section 29.4.1). New York, NY: W. H. Freeman. Disponível em https://www.ncbi.nlm.nih.gov/books/NBK22408/#_A4190_.
- Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). Eukaryotic protein synthesis differs from prokaryotic protein synthesis primarily in translation initiation. In Biochemistry. (5th ed., section 29.5). New York, NY: W. H. Freeman. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK22531/.
- Marintchev, A. and Wagner, G. (2004). Translation initiation: Structures, mechanisms, and evolution. In Quarterly Reviews of Biophysics, 37(3/4), 214-215. http://dx.doi.org/10.1017/S0033583505004026. Disponível em https://gwagner.med.harvard.edu/sites/gwagner.med.harvard.edu/files/marintchev_qrb.pdf.
- Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). Protein factors play key roles in protein synthesis. In Biochemistry. (5th ed., section 29.4.2). New York, NY: W. H. Freeman. Disponível em https://www.ncbi.nlm.nih.gov/books/NBK22408/#_A4192_
- Titin. (12 de outubro de 2016). Acesso em 22 de outubro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Titin.
- Opitz, C. A., Kulke, M., Leake, M. C., Neagoe, C., Hinssen, H., Hajjar, R. J., and Linke, W. A. (2003). Damped elastic recoil of the titin spring in myofibrils of human myocardium. PNAS, 100(22), 12688. http://dx.doi.org/10.1073/pnas.2133733100.
Referências
Berg, J. M., Tymoczko, J. L., and Stryer, L. (2002). A ribosome is a ribonucleoprotein particle (70S) made of a small (30S) and a large (50S) subunit. In Biochemistry. (5th ed., section 29.2). New York, NY: W. H. Freeman. Disponível em https://www.ncbi.nlm.nih.gov/books/NBK22335/.Berg, J. M.; Tymoczko, J. L.; Stryer, L. Eukaryotic protein synthesis differs from prokaryotic protein synthesis primarily in translation initiation. In Biochemistry. 5 ed. New York: W. H. Freeman, 2002. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK22531/.Berg, J. M.; Tymoczko, J. L.; Stryer, L. Protein factors play key roles in protein synthesis. In Biochemistry. 5 ed. New York: W. H. Freeman, 2002. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK22408/.Cooper, G. M. Translation of mRNA. In The cell: A molecular approach. 2 ed, 2000. Disponível em http://www.ncbi.nlm.nih.gov/books/NBK9849/.Amino acids. (9 de setembro de 2015). In MedLine Plus. Disponível em: https://www.nlm.nih.gov/medlineplus/ency/article/002222.htm.Erythromycin. (27 de dezembro de 2015). Acesso em 31 de dezembro de 2015 em Wikipedia: https://en.wikipedia.org/wiki/Erythromycin.Eukaryotes use a complex of many initiation factors. (2007). In Protein synthesis. Disponível em http://bioscience.jbpub.com/cells/MBIO269.aspx.Initiation involves base pairing between mRNA and rRNA. (2007). In Protein synthesis. Disponível em http://bioscience.jbpub.com/cells/MBIO267.aspx.Initiator tRNA. (n.d). A special initiator tRNA starts the polypeptide chain. Disponível em http://molecularstudy.blogspot.com/2012/10/a-special-initiator-trna-starts.html. Acesso em 31 de dezembro de 2015.Laursen, B. S.; Sørensen, H. P.; Mortensen, K. K.; Sperling-Petersen, H. U. Initiation of protein synthesis in bacteria. Microbiol. Mol. Biol. Rev , v. 69, n.1, p. 101-123, 2005.Marintchev, A. and Wagner, G. Translation initiation: Structures, mechanisms, and evolution. In Quarterly Reviews of Biophysics, v. 37 n.3/4, p. 197-284, 2004. Disponível em https://gwagner.med.harvard.edu/sites/gwagner.med.harvard.edu/files/marintchev_qrb.pdf.Nakagawa, S.; Niimura, Y.; Miura, K.,; Gojobori, T. Dynamic evolution of translation initiation mechanisms in prokaryotes. PNAS, v. 107, n. 14, p. 6382-6387, 2010.Neyfakh, A.; Mankin, A. Fundamentals of drug action I: lecture 3, 2000. Disponível em http://www.uic.edu/classes/phar/phar331/.Diphtheria toxin. In: Wikipédia: a enciclopédia livre. Disponível em: http://pt.wikipedia.org/w/index.php?title=Conteúdo_aberto&oldid=15696001. Acesso em 21 de dezembro de 2015.OpenStax College, Biology. (30 de setembro de 2015). Ribosomes and protein synthesis. In OpenStax CNX. Disponível em: https://cnx.org/contents/s8Hh0oOc@8.57:FUH9XUkW@6/Translation.OpenStax College, Biology. (n.d.). Translation. In OpenStax CNX. Disponível em: http://philschatz.com/biology-concepts-book/contents/m45479.html.Ophardt, Charles E. (2003). Other antibiotics. In Virtual Chembook. Acesso em 22 de outubro de 2016 em http://chemistry.elmhurst.edu/vchembook/654antibiotic.html.Opitz, C. A., Kulke, M., Leake, M. C., Neagoe, C., Hinssen, H., Hajjar, R. J., and Linke, W. A. (2003). Damped elastic recoil of the titin spring in myofibrils of human myocardium. PNAS, 100(22), 12688-12693. http://dx.doi.org/10.1073/pnas.2133733100.Purves, W.K.; Sadava, D.; Orians, G.H.; Heller, H.C. From DNA to protein: Genotype to phenotype. In Life: the science of biology. 7 ed., p. 233-256, Sunderland: Sinauer Associates, 2004.Rasmussen, L. C. V.; Laursen, B. S.; Mortensen, K. K.; Sperling-Petersen, H. U. Initiator tRNAs in bacteria and eukaryotes. eLS. 2009.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Translation is the RNA-directed synthesis of a polypeptide: A closer look. In Campbell biology (10th ed., pp. 345-353). San Francisco, CA: Pearson.Small subunits scan for initiation sites on eukaryotic mRNA. In Protein synthesis. 2007. Disponível em http://bioscience.jbpub.com/cells/MBIO268.aspx.Titin. (12 de outubro de 2016). Acesso em 22 de outubro de 2016 em Wikipedia: https://en.wikipedia.org/wiki/Titin. Direcionamento de proteínasIntrodução
Diversas proteínas precisam ser transportadas para diversas partes da célula eucariótica, ou em alguns casos, exportadas da célula para o espaço extracelular. Como as proteínas certas chegam aos locais certos?Para garantir que as proteínas cheguem aos destinos certos, as células possuem vários sistemas de entrega, assim como os nossos serviços postais. Nesses sistemas, rótulos ou identificadores moleculares (muitas vezes, sequências de aminoácidos) são utilizados para endereçar as proteínas para entrega nos locais específicos. Vamos dar uma olhada como esses sistemas de entrega funcionam.Aspectos gerais das rotas de transporte celular
O transporte de todas as proteínas de uma célula eucariótica começa no citosol (exceto umas poucas proteínas feitas nas mitocôndrias e cloroplastos). Assim que uma proteína é produzida, ela segue passo a passo pela "árvore decisória" da entrega. A cada passo, a proteína é checada em busca de marcadores moleculares, para ver se ela precisa ser redirecionada para um caminho ou destino diferente.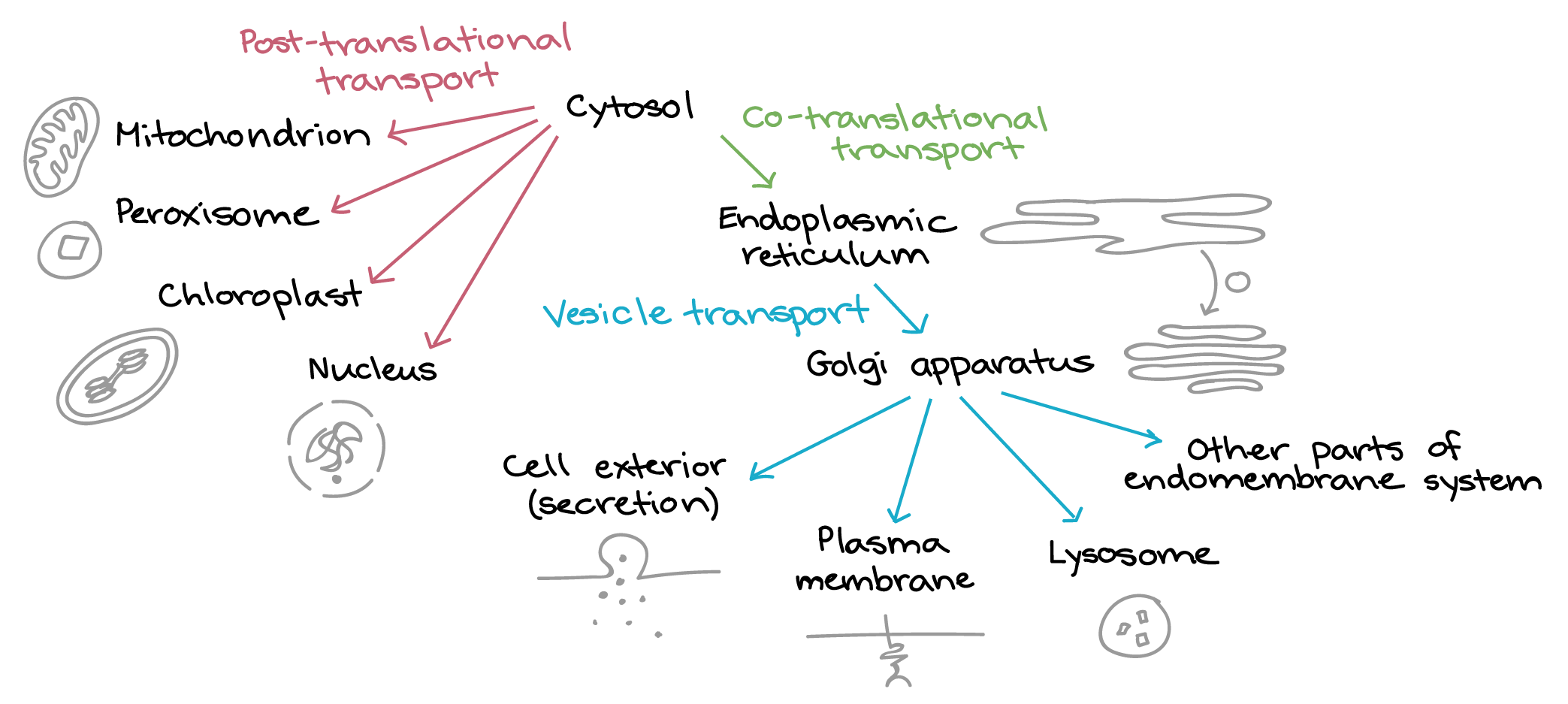 Todas as proteínas começam sua síntese no citosol. Muitas ficam ali para sempre, mas algumas são transportadas para outros destinos celulares.Algumas são completamente sintetizadas no citosol. Elas podem ser importadas para o interior da mitocôndria, peroxissomo, cloroplasto e núcleo, após o transporte.Outras proteínas são importadas para o interior do retículo endoplasmático. Daí, a maioria vai até o Complexo de Golgi por transporte vesicular. Do Complexo de Golgi (ainda por transporte vesicular), as proteínas podem alcançar o exterior celular (por secreção), membrana plasmática, lisossomo, ou outras partes do sistema de endomenbranas.Diagrama baseado em Alberts et al. 1start superscript, 1, end superscript.O primeiro ponto principal da ramificação é logo no início do transporte. Neste ponto, a proteína pode permanecer no citosol pelo resto do processo, ou ser enviada para o interior do retículo endoplasmático (RE), pelo transporte2squared.
Todas as proteínas começam sua síntese no citosol. Muitas ficam ali para sempre, mas algumas são transportadas para outros destinos celulares.Algumas são completamente sintetizadas no citosol. Elas podem ser importadas para o interior da mitocôndria, peroxissomo, cloroplasto e núcleo, após o transporte.Outras proteínas são importadas para o interior do retículo endoplasmático. Daí, a maioria vai até o Complexo de Golgi por transporte vesicular. Do Complexo de Golgi (ainda por transporte vesicular), as proteínas podem alcançar o exterior celular (por secreção), membrana plasmática, lisossomo, ou outras partes do sistema de endomenbranas.Diagrama baseado em Alberts et al. 1start superscript, 1, end superscript.O primeiro ponto principal da ramificação é logo no início do transporte. Neste ponto, a proteína pode permanecer no citosol pelo resto do processo, ou ser enviada para o interior do retículo endoplasmático (RE), pelo transporte2squared.- As proteínas são entregues no RE pelo transporte se elas tiverem uma sequência de aminoácidos chamada de peptídeo sinal. Em geral as proteínas destinadas às organelas do sistema de endomembranas (tais como o RE, aparelho de Golgi e lisossomo) ou ao exterior da célula, entram no RE nesta etapa.
- As proteínas não sinalizadas com peptídeo permanecem no citosol pelo restante do transporte. Se elas não tiverem outros "rótulos de endereçamento," ficarão de forma permanente no citosol. Mas se elas tiverem o rótulo correto, elas serão encaminhadas para a mitocôndria, cloroplastos, peroxissomos ou núcleo pelo transporte.
Sistema de endomembranas e via secretória
As proteínas destinadas a qualquer parte do sistema de endomembranas (ou para o meio extracelular) são trazidas ao RE pelo sistema de transporte e inseridas conforme são produzidas.Peptídeos-sinal
O peptídeo-sinal que envia a proteína para o interior do retículo endoplasmático durante a tradução é constituído por uma série de aminoácidos hidrofóbicos ("que temem a água”), geralmente encontrados próximo ao início (N-terminal) da proteína. Quando essa sequência sai do ribossomo, é reconhecida por um complexo proteico chamado de partícula reconhecedora de sinal (PRS), que leva o ribossomo para o RE. Ali o ribossomo entrega a sua cadeia de aminoácidos no lúmen (interior) do RE, conforme produzida.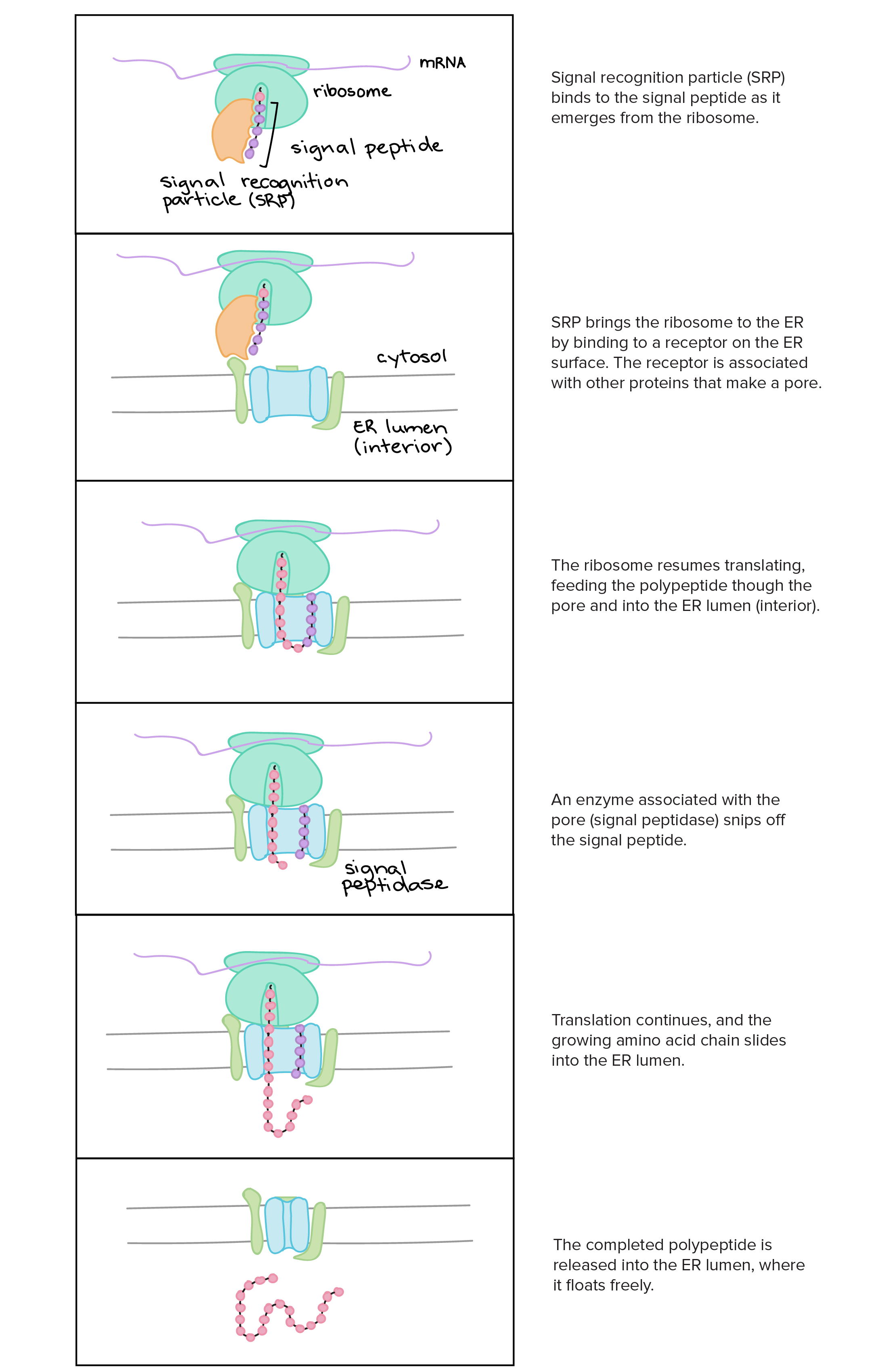
- A Partícula Reconhecedora de Sinal (PRS) liga-se ao peptídeo sinal conforme ele emerge do ribossoma.
- A PRS trás o ribossomo ao RE, ligando-se a um receptor na superfície do RE. O receptor está associado com outras proteínas que fazem um poro.
- O ribossomo retoma a tradução, alimentando o polipeptídeo através do poro e para dentro do lúmen (interior) do RE.
- Uma enzima associada com o poro corta o peptídeo sinal.
- A tradução continua e a cadeia crescente de aminoácidos desliza para o interior do RE.
- O polipeptídeo completo é liberado no lúmen do RE, onde fica flutuando livremente.
Em alguns casos, o peptídeo-sinal é cortado durante a tradução e a proteína final é liberada no interior do RE (como mostrado acima). Em outros casos o peptídeo-sinal ou outro segmento de aminoácidos hidrofóbicos fica incorporado na membrana do RE. Isto cria um segmento transmembranar (membrana cruzada) que ancora a proteína à membrana.O transporte através do sistema de endomembranas
No RE, as proteínas dobram-se nas suas formas corretas e podem também ter grupos de açúcares ligados à elas. A maioria das proteínas é então transportada para o complexo de Golgi, em vesículas membranosas. Algumas proteínas, no entanto, precisam ficar no RE e fazer seu trabalho ali mesmo. Essas proteínas têm aminoácidos marcadores que asseguram que elas sejam trazidas de volta para o RE se "escaparem" para dentro do Golgi.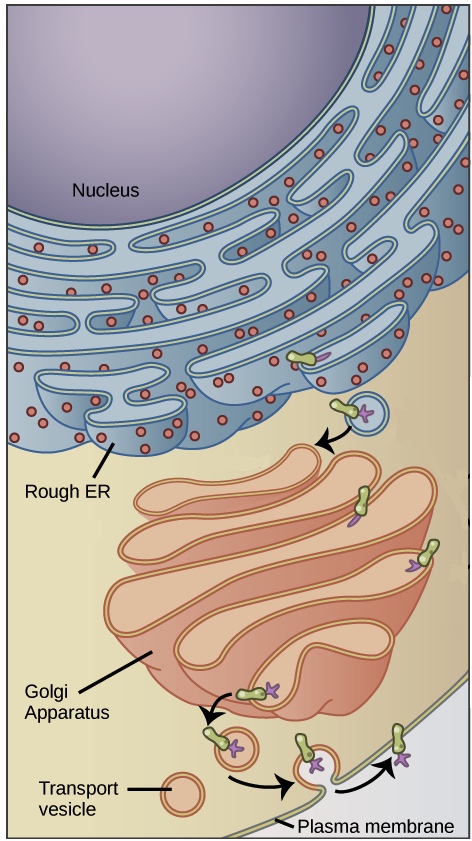 Imagem mostrando o transporte de uma proteína membranar a partir do RE rugoso, através do Golgi, até a membrana plasmática. A proteína é inicialmente modificada pela adição de cadeias de carboidratos ramificadas no RE rugoso; depois, estas são reduzidas e substituídas por outras cadeias de carboidratos no complexo de Golgi. A proteína, com seu conjunto final de cadeias de carboidratos é então transportada para a membrana plasmática em uma vesícula de transporte. A vesícula se funde com a membrana plasmática, seus lipídios e carga de proteína se tornam parte da membrana plasmática._Imagem modificada de "The endomembrane system and proteins: Figure 1," por OpenStax College, Biology (CC BY 3.0)._No complexo de Golgi, as proteínas podem sofrer mais modificações (tais como adição de grupos de açúcar), antes de alcançarem seus destinos finais. Esses destinos incluem lisossomos, membrana plasmática e exterior da célula. Algumas proteínas precisam executar suas funções no Golgi (são as "Golgi residentes"), e uma variedade de sinais moleculares, incluindo marcações de aminoácidos e características estruturais, são usados para mantê-las ali e para trazê-las de volta3cubed.Se elas não têm nenhum marcador específico, as proteínas são enviadas do Golgi para a superfície celular, onde são secretadas para o exterior celular (se forem livres) ou entregues para a membrana plasmática (se forem incorporadas às membranas). Essa via padrão é mostrada no diagrama acima para a proteína de membrana, colorida em verde, que carrega grupos de açúcar, coloridos de roxo.As proteínas são enviadas para outros destinos se contiverem os marcadores moleculares certos. Por exemplo, as proteínas destinadas aos lisossomos têm marcador molecular consistindo de um açúcar com um grupo fosfato aderido. No complexo de Golgi, as proteínas com marcadores são organizadas nas vesículas destinadas aos lisossomos.
Imagem mostrando o transporte de uma proteína membranar a partir do RE rugoso, através do Golgi, até a membrana plasmática. A proteína é inicialmente modificada pela adição de cadeias de carboidratos ramificadas no RE rugoso; depois, estas são reduzidas e substituídas por outras cadeias de carboidratos no complexo de Golgi. A proteína, com seu conjunto final de cadeias de carboidratos é então transportada para a membrana plasmática em uma vesícula de transporte. A vesícula se funde com a membrana plasmática, seus lipídios e carga de proteína se tornam parte da membrana plasmática._Imagem modificada de "The endomembrane system and proteins: Figure 1," por OpenStax College, Biology (CC BY 3.0)._No complexo de Golgi, as proteínas podem sofrer mais modificações (tais como adição de grupos de açúcar), antes de alcançarem seus destinos finais. Esses destinos incluem lisossomos, membrana plasmática e exterior da célula. Algumas proteínas precisam executar suas funções no Golgi (são as "Golgi residentes"), e uma variedade de sinais moleculares, incluindo marcações de aminoácidos e características estruturais, são usados para mantê-las ali e para trazê-las de volta3cubed.Se elas não têm nenhum marcador específico, as proteínas são enviadas do Golgi para a superfície celular, onde são secretadas para o exterior celular (se forem livres) ou entregues para a membrana plasmática (se forem incorporadas às membranas). Essa via padrão é mostrada no diagrama acima para a proteína de membrana, colorida em verde, que carrega grupos de açúcar, coloridos de roxo.As proteínas são enviadas para outros destinos se contiverem os marcadores moleculares certos. Por exemplo, as proteínas destinadas aos lisossomos têm marcador molecular consistindo de um açúcar com um grupo fosfato aderido. No complexo de Golgi, as proteínas com marcadores são organizadas nas vesículas destinadas aos lisossomos.Direcionando para organelas não endomembranosas
As proteínas que são feitas no citosol (não entram no RE durante a tradução) podem ficar permanentemente no citosol. Porém elas também podem ser encaminhadas para outros destinos não membranosos na célula. Por exemplo, proteínas limitadas pela mitocôndria, cloroplastos, peroxissomos e núcleo, são geralmente produzidas no citosol e entregues após a conclusão da tradução.Para ser entregue a algumas dessas organelas após a tradução, a proteína precisa conter um aminoácido específico "rótulo de endereço." O rótulo é reconhecido por outras proteínas na célula, o que ajuda o transporte da proteína para o destino correto.Como exemplo, vamos considerar a entrega ao peroxissomo, uma organela envolvida na destoxificação. As proteínas necessárias ao peroxissomo têm uma sequência específica de aminoácidos chamada sinal alvo do peroxissomo. O sinal clássico consiste de apenas três aminoácidos, serina-lisina-leucina, encontrado no final (C-terminal) da proteína. Esse padrão de aminoácidos é reconhecido pela proteína auxiliar no citosol, que trás a proteína para o peroxissomo4start superscript, 4, end superscript.O direcionamento mitocondrial, para o cloroplasto e nuclear, geralmente são similares ao alvo peroxissomal. Isto é, uma certa sequência de aminoácidos envia a proteína para sua organela-alvo (ou um compartimento dentro da organela). No entanto, a natureza dos "rótulos de endereçamento" são diferentes em cada caso. Este artigo está licenciado pela CC BY-NC-SA 4.0Referências:
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., and Walter, P. (2002). A simplified “roadmap” of protein traffic. In Molecular biology of the cell (4th ed.). New York, NY: Garland Science. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK26907/figure/A2143/?report=objectonly.
- Purves, W.K., Sadava, D., Orians, G.H., and Heller, H.C. (2003). Chemical signals in proteins direct them to their cellular destinations. In Life: the science of biology (7th ed., p. 247). Sunderland, MA: Sinauer Associates.
- Banfield, D. K. (2011). Mechanisms of protein retention in the Golgi. Cold Spring Harbor Perspectives in Biology, 3, a005264. http://dx.doi.org/10.1101/cshperspect.a005264.
- Lodish, H., Berk, A., Zipursky, S.L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000) Synthesis and targeting of peroxisomal proteins. In Molecular cell biology (4th ed., section 17.2). New York, NY: W. H. Freeman. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK21520/.
Referências
Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., and Walter, P. (2002). The compartmentalization of cells. In Molecular biology of the cell (4th ed.). New York, NY: Garland Science. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK26907/.Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., and Walter, P. (2002). Transport from the ER through the Golgi apparatus. In Molecular biology of the cell (4th ed.). New York, NY: Garland Science. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK26941/.Cooper, G. M. (2000). Lysosomes. In The cell: a molecular approach. (2nd ed.). Sunderland, MA: Sinauer Associates. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK9953/.Cooper, G. M. (2000). The endoplasmic reticulum. In The cell: a molecular approach. (2nd ed.). Sunderland, MA: Sinauer Associates. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK9838/.Cooper, G. M. (2000). The Golgi apparatus. In The cell: a molecular approach. (2nd ed.). Sunderland, MA: Sinauer Associates. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK9838/.Karp, G. (2010). Posttranslational uptake of proteins by peroxisomes, mitochondria, and chloroplasts. In Cell and molecular biology: Concepts and experiments (6th ed., pp. 309-311). Hoboken, NJ: John Wiley & Sons.Lodish, H., Berk, A., Zipursky, S.L., Matsudaira, P., Baltimore, D., and Darnell, J. (2000) Synthesis and targeting of mitochondrial and chloroplast proteins. In Molecular cell biology (4th ed., section 17.1). New York, NY: W. H. Freeman. Disponível em: http://www.ncbi.nlm.nih.gov/books/NBK21652/.OpenStax College, Biology. (30 de setembro de 2015). Ribosomes and protein synthesis. In OpenStax CNX. Disponível em: https://cnx.org/contents/s8Hh0oOc@8.57:FUH9XUkW@6/Translation.OpenStax College, Biology. (n.d.). Translation. In OpenStax CNX. Disponível em: http://philschatz.com/biology-concepts-book/contents/m45479.html.Peroxisomal targeting signal. (2014, 17 December). Disponível em: Wikipedia: https://en.wikipedia.org/wiki/Peroxisomal_targeting_signal. Acesso em 2 fev. 2016Protein synthesis. (s.d.) In NobelPrize.org. Disponível em: http://www.nobelprize.org/nobel_prizes/medicine/laureates/1999/illpres/protein.html.Purves, W.K., Sadava, D., Orians, G.H., and Heller, H.C. (2003). Chemical signals in proteins direct them to their cellular destinations. In Life: the science of biology (7th ed., pp. 247-249). Sunderland, MA: Sinauer Associates.Reece, J. B., Urry, L. A., Cain, M. L., Wasserman, S. A., Minorsky, P. V., and Jackson, R. B. (2011). Gene expression: from gene to protein. In Campbell biology (10th ed., pp. 333-359). San Francisco, CA: Pearson.Stavely, B. E. (2015). Protein targeting and sorting. In Principles of cell biology (BIOL2060). Disponível em: http://www.mun.ca/biology/desmid/brian/BIOL2060/BIOL2060-22/CB22.html. Exercícios de Fixação:- 1-O RNA é um ácido nucleico, assim como o DNA. Entretanto, em sua estrutura, encontramos uma base nitrogenada exclusiva desse tipo de ácido nucleico. Marque a alternativa que indica o nome dessa base:
a) Timina.
b) Guanina.
c) Uracila.
d) Adenina.
e) Citosina.
2- Sabemos que existem diferentes tipos de RNA. Analise as alternativas abaixo e marque aquela que apresenta o nome correto da classe de RNA que é responsável por codificar as proteínas e ter seus códons lidos na tradução.
a) RNA mensageiro.
b) RNA codificador.
c) RNA transportador.
d) RNA de códons.
e) RNA ribossomal.
3- Sabemos que existem três tipos básicos de RNA. Observe cada uma das alternativas abaixo e indique aquela que apresenta a função do RNA transportador.
a) Codificar as proteínas.
b) Transportar a proteína pronta.
c) Transportar um aminoácido.
d) Formar os ribossomos.
e) Transportar as unidades ribossomais.
4- Em que local da célula o RNA ribossômico forma as unidades ribossomais?
a) Membrana plasmática.
b) Citoplasma.
c) Retículo endoplasmático.
d) Nucléolo.
e) Mitocôndria.
5- (Unisinos) Durante o processo de tradução do código genético, o ________ promoverá a síntese de proteínas através da leitura de trincas de bases nitrogenadas, chamadas de ________, e cada uma corresponderá a um aminoácido da proteína que está sendo formado. Assim, diferentes sequências de aminoácidos formam diferentes proteínas no interior da célula.
Sobre o processo de tradução do código genético descrito acima, qual das alternativas abaixo preenche, correta e respectivamente, as lacunas?
a) RNA transportador; éxons.
b) RNA transportador; códon.
c) RNA ribossômico; éxons.
d) RNA mensageiro; íntrons.
e) RNA mensageiro; códon.